| 1 | Найти точное значение | sin(30) | |
| 2 | Найти точное значение | sin(45) | |
| 3 | Найти точное значение | sin(30 град. ) | |
| 4 | Найти точное значение | sin(60 град. ) | |
| 5 | Найти точное значение | tan(30 град. ) | |
| 6 | Найти точное значение | arcsin(-1) | |
| 7 | Найти точное значение | sin(pi/6) | |
| 8 | cos(pi/4) | ||
| 9 | Найти точное значение | sin(45 град. ) ) | |
| 10 | Найти точное значение | sin(pi/3) | |
| 11 | Найти точное значение | arctan(-1) | |
| 12 | Найти точное значение | cos(45 град. ) | |
| 13 | Найти точное значение | cos(30 град. ) | |
| 14 | Найти точное значение | tan(60) | |
| 15 | Найти точное значение | csc(45 град. ) | |
| 16 | Найти точное значение | tan(60 град. ) | |
| 17 | Найти точное значение | sec(30 град. ) ) | |
| 18 | Найти точное значение | cos(60 град. ) | |
| 19 | Найти точное значение | cos(150) | |
| 20 | Найти точное значение | sin(60) | |
| 21 | Найти точное значение | cos(pi/2) | |
| 22 | Найти точное значение | tan(45 град. ) | |
| 23 | Найти точное значение | arctan(- квадратный корень из 3) | |
| 24 | Найти точное значение | csc(60 град. ) | |
| 25 | Найти точное значение | sec(45 град. ) ) | |
| 26 | Найти точное значение | csc(30 град. ) | |
| 27 | Найти точное значение | sin(0) | |
| 28 | Найти точное значение | sin(120) | |
| 29 | Найти точное значение | cos(90) | |
| 30 | Преобразовать из радианов в градусы | pi/3 | |
| 31 | Найти точное значение | tan(30) | |
| 32 | Преобразовать из градусов в радианы | 45 | |
| 33 | Найти точное значение | cos(45) | |
| 34 | Упростить | sin(theta)^2+cos(theta)^2 | |
| 35 | Преобразовать из радианов в градусы | pi/6 | |
| 36 | Найти точное значение | cot(30 град.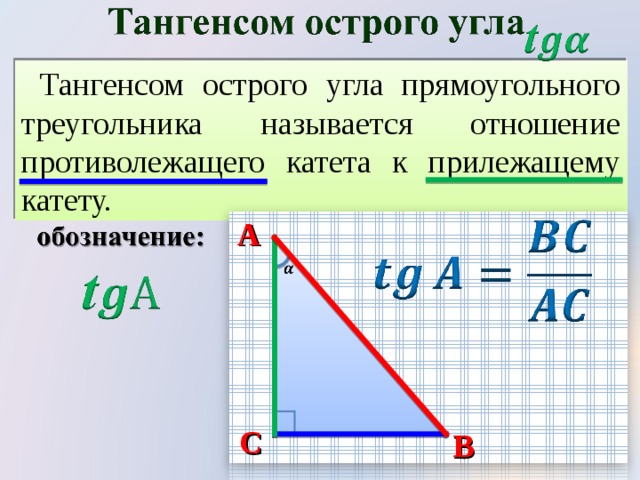 ) ) | |
| 37 | Найти точное значение | arccos(-1) | |
| 38 | Найти точное значение | arctan(0) | |
| 39 | Найти точное значение | cot(60 град. ) | |
| 40 | Преобразовать из градусов в радианы | 30 | |
| 41 | Преобразовать из радианов в градусы | (2pi)/3 | |
| 42 | Найти точное значение | sin((5pi)/3) | |
| 43 | Найти точное значение | sin((3pi)/4) | |
| 44 | Найти точное значение | tan(pi/2) | |
| 45 | Найти точное значение | sin(300) | |
| 46 | Найти точное значение | cos(30) | |
| 47 | Найти точное значение | cos(60) | |
| 48 | Найти точное значение | cos(0) | |
| 49 | Найти точное значение | cos(135) | |
| 50 | Найти точное значение | cos((5pi)/3) | |
| 51 | Найти точное значение | cos(210) | |
| 52 | Найти точное значение | sec(60 град.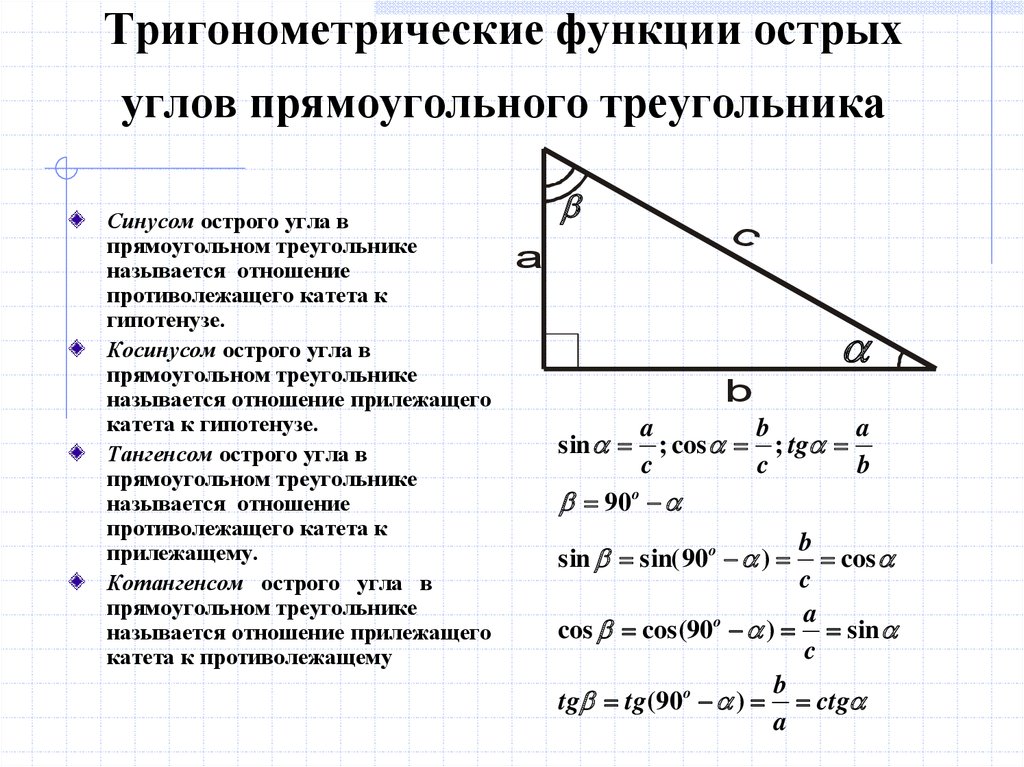 ) ) | |
| 53 | Найти точное значение | sin(300 град. ) | |
| 54 | Преобразовать из градусов в радианы | 135 | |
| 55 | Преобразовать из градусов в радианы | 150 | |
| 56 | Преобразовать из радианов в градусы | (5pi)/6 | |
| 57 | Преобразовать из радианов в градусы | (5pi)/3 | |
| 58 | Преобразовать из градусов в радианы | 89 град. | |
| 59 | Преобразовать из градусов в радианы | 60 | |
| 60 | Найти точное значение | sin(135 град.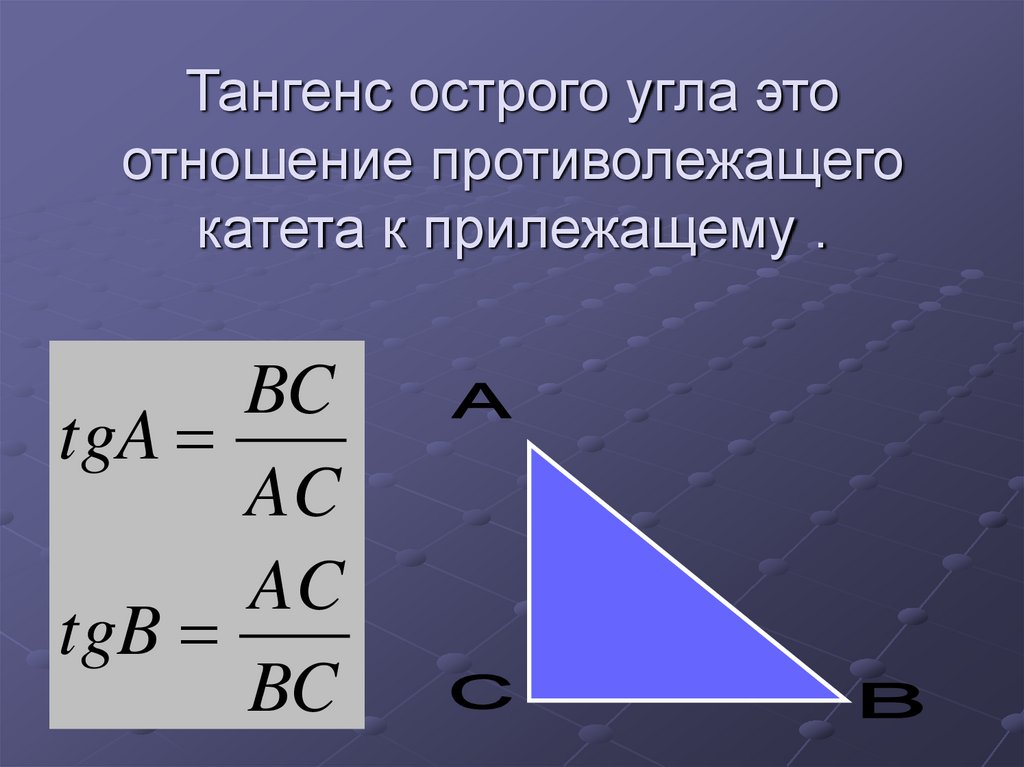 ) ) | |
| 61 | Найти точное значение | sin(150) | |
| 62 | Найти точное значение | sin(240 град. ) | |
| 63 | Найти точное значение | cot(45 град. ) | |
| 64 | Преобразовать из радианов в градусы | (5pi)/4 | |
| 65 | Найти точное значение | sin(225) | |
| 66 | Найти точное значение | sin(240) | |
| 67 | Найти точное значение | ||
| 68 | Найти точное значение | tan(45) | |
| 69 | Вычислить | sin(30 град.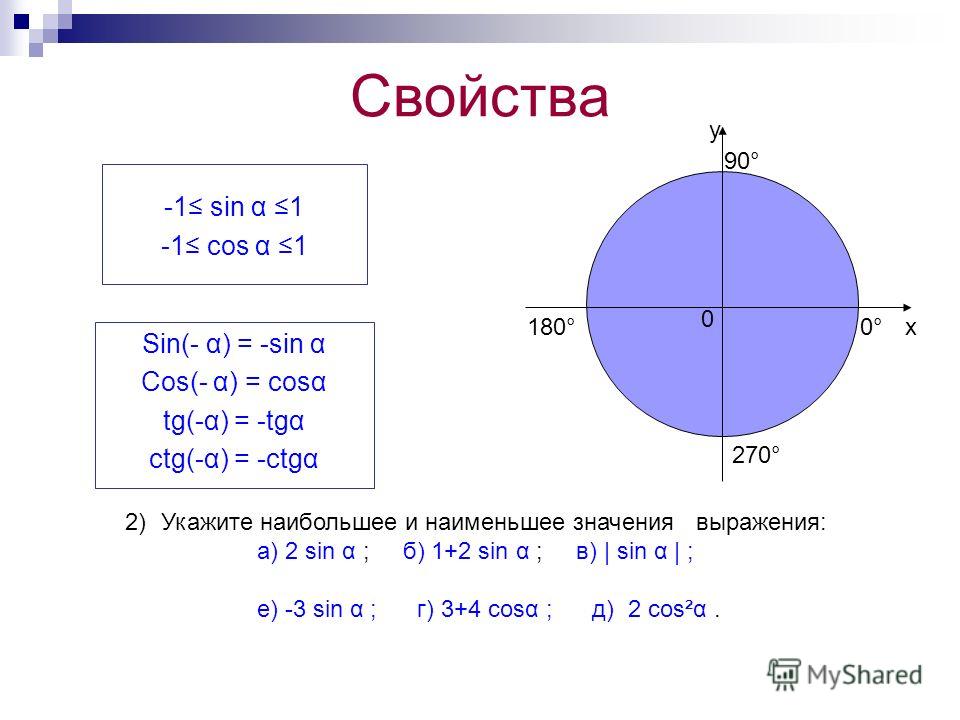 ) ) | |
| 70 | Найти точное значение | sec(0) | |
| 71 | Найти точное значение | cos((5pi)/6) | |
| 72 | Найти точное значение | csc(30) | |
| 73 | Найти точное значение | arcsin(( квадратный корень из 2)/2) | |
| 74 | Найти точное значение | tan((5pi)/3) | |
| 75 | Найти точное значение | tan(0) | |
| 76 | Вычислить | sin(60 град. ) | |
| 77 | Найти точное значение | arctan(-( квадратный корень из 3)/3) | |
| 78 | Преобразовать из радианов в градусы | (3pi)/4 | |
| 79 | Найти точное значение | sin((7pi)/4) | |
| 80 | Найти точное значение | arcsin(-1/2) | |
| 81 | Найти точное значение | sin((4pi)/3) | |
| 82 | Найти точное значение | csc(45) | |
| 83 | Упростить | arctan( квадратный корень из 3) | |
| 84 | Найти точное значение | sin(135) | |
| 85 | Найти точное значение | sin(105) | |
| 86 | Найти точное значение | sin(150 град.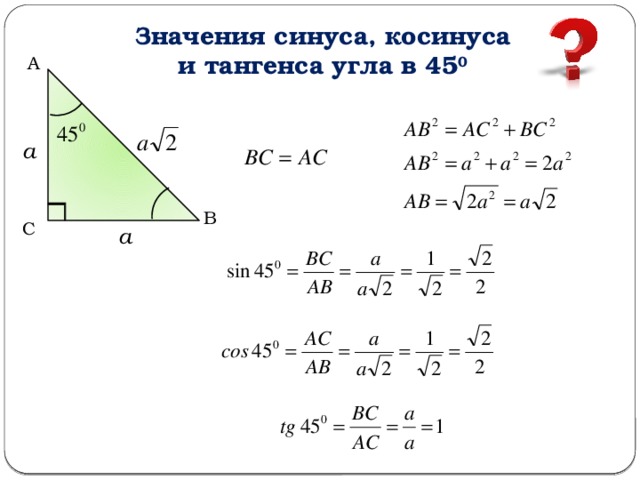 ) ) | |
| 87 | Найти точное значение | sin((2pi)/3) | |
| 88 | Найти точное значение | tan((2pi)/3) | |
| 89 | Преобразовать из радианов в градусы | pi/4 | |
| 90 | Найти точное значение | sin(pi/2) | |
| 91 | Найти точное значение | sec(45) | |
| 92 | Найти точное значение | cos((5pi)/4) | |
| 93 | Найти точное значение | cos((7pi)/6) | |
| 94 | Найти точное значение | arcsin(0) | |
| 95 | Найти точное значение | sin(120 град. ) ) | |
| 96 | Найти точное значение | tan((7pi)/6) | |
| 97 | Найти точное значение | cos(270) | |
| 98 | Найти точное значение | sin((7pi)/6) | |
| 99 | Найти точное значение | arcsin(-( квадратный корень из 2)/2) | |
| 100 | Преобразовать из градусов в радианы | 88 град. |
Синус 30 градусов, sin 30
Синус 30 градусов равняется одной второй или ноль целых пять десятых.
sin 30° = 1/2 или sin 30° = 0,5
В радианной мере измерения углов синусу 30 градусов соответствует синус π/6:
sin 30° = sin π/6
Как это ни странно, но справедливо и обратное равенство, которое утверждает, что синус π/6 (синус пи на 6) равняется синусу 30 градусов:
sin π/6 = sin 30°
Равняется синус пи / 6 так же одной второй или ноль целых пять десятых.
sin π/6 = 1/2 или sin π/6 = 0,5
Это было для блондинок. Для брюнеток и лысых академиков математических наук можно записать всё это в общем виде, пусть распутывают:
sin 30
Для полного счастья здесь явно не хватает картинки синуса 30 градусов. А вот и она:
Надеюсь, первую часть задачи я решил и мне удалось объяснить блондинкам, чему равен синус тридцати градусов. Теперь нужно решить вторую половину задачи, с которой не в состоянии справиться даже вся академия математических наук, вместе взятая. Нужно в Интернете найти блондинок, которые ищут синус 30 градусов. Попробую вооружиться логикой блондинок и перечислю ниже поисковые запросы, которые блондинки могут вводить в поисковые системы при поиске ответа на вопрос: чему равен синус 30 градусов? И так, поисковые запросы, разбавленные моими комментариями, дабы поисковые системы не отлучили мое творение от блондинок.
Синус — это математики сверяют свои знания с Интернетом. Синус, косинус — появилось начальство математиков, чтобы проверить, как математики сверили свои знания. Синус угла — это очкастые ботаники, будущие Билы Гейтсы, чешут свои умные репы и пытаются вспомнить школьный курс математики. Синус градусов — шустрые школьники мимоходом забрасывают свой вопрос, что бы побыстрее разделаться с этой бякой и продолжить игру. Таблица синусов, тангенсов — добросовестные школьники и добропорядочные брюнетки соскребли все свои познания в тригонометрии и пытаются сверить их с Интернетом. Значение синуса — математики, после долгих блужданий по сайтам с блондинками, наконец-то поняли, как лучше сформулировать поисковый запрос. Значения косинусов и синусов — математики вдруг вспомнили, что не синусом единым живет тригонометрия. Синус чему равен? — а вот и первые признаки присутствия блондинок в Интернете с их подкупающей непосредственностью общения, даже с компьютером.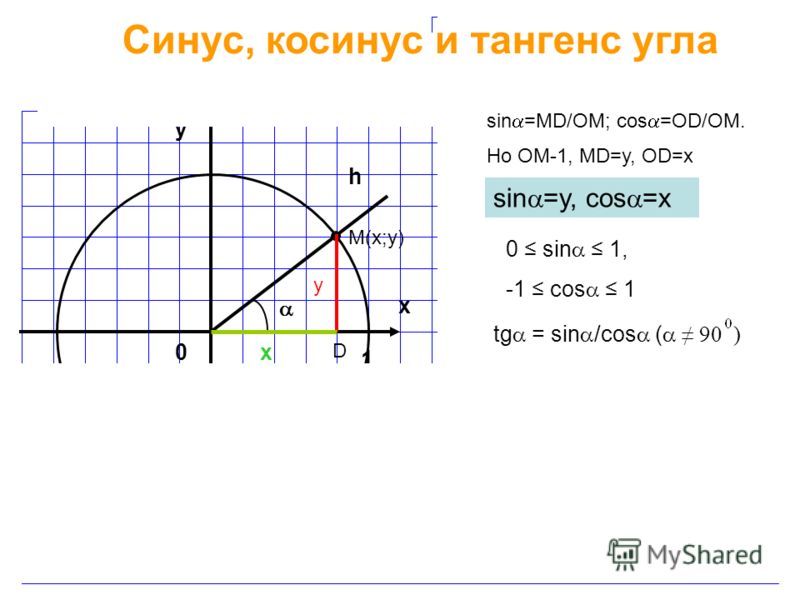 Синусы и косинусы углов. Таблица значений синусов. Синус угла равен — это мамы пытаются проверить, как их чада сделали уроки. Как найти синус? — это уже типичный вопрос растерянной блондинки. Синус острого угла. Синус и косинус 30 — мамы с трудом, но уже начинают понимать, что требуется найти в задаче. Синус числа — бедные мамы, они даже не подозревают, что углы можно измерять радианами. Синус альфа — мамы вспомнили, какой буквой когда-то в школе они сами обозначали углы. Калькулятор синусов — на помощь беспомощным мамам приходят умные папы. Как найти синус угла? — умные папы нашли калькулятор синуса, теперь нужно узнать, как же этой штукой пользоваться. Геометрия синус, косинус. Скачать таблицу синусов. Значения синусов углов — это чада оторвались от своих виртуальных игр и пытаются доказать умным папам, что папы не правильно нажимают на кнопочки калькулятора, потому что синус угла не может равняться трем банкам пива.
Синусы и косинусы углов. Таблица значений синусов. Синус угла равен — это мамы пытаются проверить, как их чада сделали уроки. Как найти синус? — это уже типичный вопрос растерянной блондинки. Синус острого угла. Синус и косинус 30 — мамы с трудом, но уже начинают понимать, что требуется найти в задаче. Синус числа — бедные мамы, они даже не подозревают, что углы можно измерять радианами. Синус альфа — мамы вспомнили, какой буквой когда-то в школе они сами обозначали углы. Калькулятор синусов — на помощь беспомощным мамам приходят умные папы. Как найти синус угла? — умные папы нашли калькулятор синуса, теперь нужно узнать, как же этой штукой пользоваться. Геометрия синус, косинус. Скачать таблицу синусов. Значения синусов углов — это чада оторвались от своих виртуальных игр и пытаются доказать умным папам, что папы не правильно нажимают на кнопочки калькулятора, потому что синус угла не может равняться трем банкам пива. Вычисления синуса — математики-теоретики пытаются слямзить результаты работы математиков-прикладников. Вычислить синус — это математики-прикладники тырят результаты работы своих коллег. Синус бесконечности — это уже физики пытаются проверить расчеты математиков. Нахождение синуса — блондинки пытаются узнать, где живут синусы. Тригонометрический синус — все с удивлением обнаружили, что синусы бывают не только в математике.
Вычисления синуса — математики-теоретики пытаются слямзить результаты работы математиков-прикладников. Вычислить синус — это математики-прикладники тырят результаты работы своих коллег. Синус бесконечности — это уже физики пытаются проверить расчеты математиков. Нахождение синуса — блондинки пытаются узнать, где живут синусы. Тригонометрический синус — все с удивлением обнаружили, что синусы бывают не только в математике.
Сколько синус 30 — ноль целых пять десятых. Скольки равен sin 30 — стольки же, ноль пять. Сколько будет синус 30? — вопрос, конечно, интересный… ышт 30 — можно и так, только лучше переключаться на английский язык. Таблица sin альфа 30градусов — здесь не совсем таблица, но и сину, с и 30 градусов точно есть. Числа из которых синусы натуральные — математики, блондинка задала вопрос. Есть варианты ответов? Честь мундира поставлена на кон.
Кто может объяснить, почему синус 30 градусов равен 1/2? — ну, вундеркинды, блондинка задала вопрос. Кто осмелится ответить?
Кто осмелится ответить?
синус какого угла равен одной второй? — Синус тридцати градусов равен нулю, а также минус единица в степени эн умноженная на 30 градусов плюс эн умноженное на число пи. Вот, целая куча углов, синус которых равен одной второй.
Синусов теорема с примерами
Добавлено: 5 ноября 2021 в 18:07
Содержание
При подготовке к ЕГЭ по математике одиннадцатиклассник должен помнить базовый набор формул, которые помогут решать задачи. Одной из них является синусов теорема, которая отражает взаимосвязь между сторонами и углами треугольника. Напомним, доказательство теоремы учить не нужно, поскольку экзамен ориентирован на проверку практических навыков. Лучше посвятить время разбору примеров, в которых можно применить указанную математическую закономерность.
Теорема синусов с примерами
Человечество знакомо с теоремой синусов довольно давно — еще в начале XXI века ее доказательство приводил в своей работе «Книга о неизвестных дугах сферы» западноарабский астроном и математик Ибн Муаз аль-Джайяни.
Существует два варианта теоремы синусов:
- обычный — устанавливает соотношения между сторонами треугольника и синусами его углов;
- расширенный — связывает соотношение сторон треугольника с радиусами описанной окружности.
Формулировка обычной синусов теоремы: отношение сторон треугольника к синусам противолежащих углов равны или стороны пропорциональны синусам противолежащих углов.
Синусов теорема с примерами
Пример 1. В треугольнике АВС сторона АВ равна 5 см, а синус противолежащего угла АСВ = 3/5. Найти сторону ВС, если синус угла САВ, прилежащего к стороне АВ, равен 1/2.
Решение
Составим соотношение фигурирующих в условии сторон и синусов их углов:
АВ : sin ∠АСВ = ВС : sin ∠САВ.
Подставим известные значения:
5 : 3/5 = ВС : 1/2.
Выразим из этого выражения ВС:
ВС = (5 : 3/5) : 1/2 = 5 : 1/2 = 10 см.
Ответ: ВС = 10 см.
Пример 2.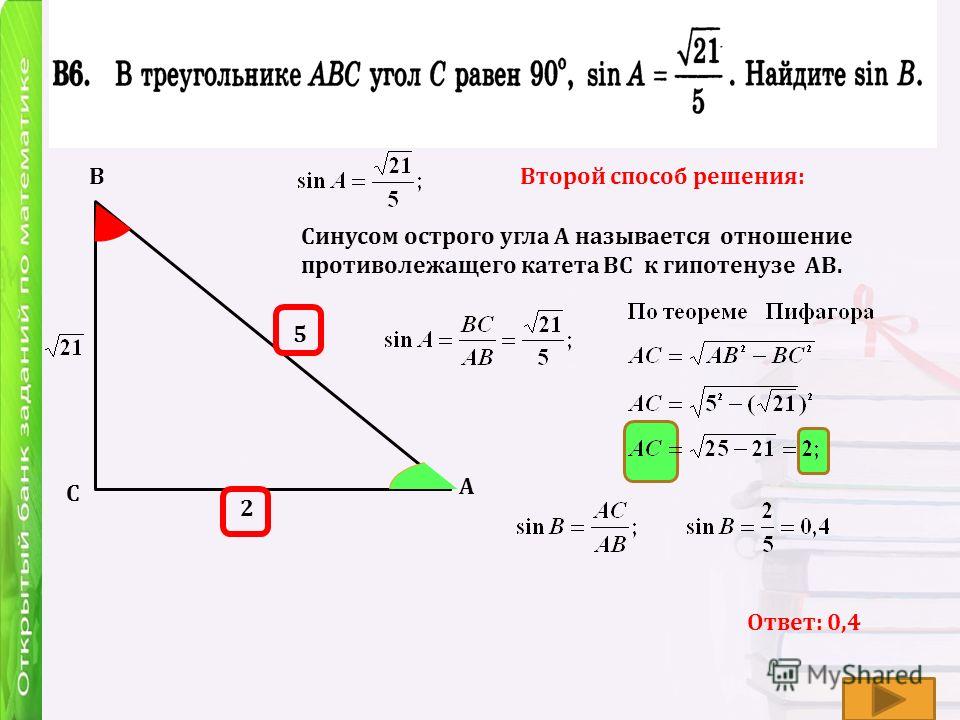 В треугольнике АВС сторона АВ равна 10 см, а противолежащий угол АСВ = 30°. Найти остальные стороны, если угол САВ равен 60°.
В треугольнике АВС сторона АВ равна 10 см, а противолежащий угол АСВ = 30°. Найти остальные стороны, если угол САВ равен 60°.
Решение
Для решения этой задачи воспользуемся прилагаемой таблицей, в которой указаны значения синусов основных углов. В остальном ход решения будет аналогичен предыдущему примеру за исключением одного маленького хода. Для начала составим соотношение сторон и синусов противолежащих углов:
АВ : sin ∠АСВ = ВС : sin ∠САВ = АС : sin ∠ВАС.
На первом этапе нам известны только три из шести членов этого равенства, причем два из них в косвенном виде:
10 : sin 30° = ВС : sin 60° = АС : sin ∠ВАС.
Если вспомнить, что сумма углов треугольника равна 180°, то легко найти оставшийся угол:
∠ВАС = 180° – (∠АСВ + ∠САВ) = 180° – (30° + 60°) = 90°.
Мы уже знаем и третий угол, поэтому уравнение приобретет следующий вид:
10 : sin 30° = ВС : sin 60° = АС : sin 90°.
Дальше поступаем, как в предыдущей задаче, выразив стороны через известные члены выражений:
ВС = sin 60° ∙ 10 : sin 30°,
АС = sin 90° ∙ 10 : sin 30°.
Обратимся к таблице, приведенной выше и выберем из нее соответствующие синусы известных углов:
ВС = √3/2∙ 10 : 1/2 = 10√3 см,
АС = 1 ∙ 10 : 1/2 = 20 см.
Ответ: ВС = 10√3 см; АС = 20 см.
Синусов теорема с примерами
Расширенная синусов теорема с примерамиФормулировка расширенной теоремы синусов: отношение сторон треугольника к синусам противолежащих углов равны друг другу и удвоенному радиусу окружности, описанной вокруг него.
Пример 3. Найти площадь треугольника, если диаметр описанной окружности D равен 20 см. Угол АСВ = 30°, а угол САВ = 60°.
Решение
Для решения воспользуемся расширенной формулировкой теоремы синусов:
АВ : sin ∠АСВ = ВС : sin ∠САВ = АС : sin ∠ВАС = 2R.
В этой формулировке нам известны два из семи компонентов и еще лва мы можем определить из базовых знаний по геометрии:
- R = ½ D, следовательно 2 R = D = 20 см;
- ∠ВАС = 180° – (∠АСВ + ∠САВ) = 180° – (30° + 60°) = 90°.

Подставим в исходное выражение известные величины и получим соотношение:
АВ : sin 30° = ВС : sin 60° = АС : sin 90° = 20.
Основным отличием от предыдущей задачи является то, что нам неизвестна сторона АВ, зато известен удвоенный радиус описанной окружности. Это позволяет составить выражения для нахождения всех сторон треугольника:
ВС = 20 ∙ sin 60°
АС = 20 ∙ sin 90°,
АВ = 20 ∙ sin 30°.
Выберем из таблицы значения синусов углов и вычитаем стороны треугольника:
ВС = 20 ∙ sin 60° = 20 ∙ √3/2 = 10√3 см,
АС = 20 ∙ sin 90° = 20 ∙ 1 = 20 см,
АВ = 20 ∙ sin 30° = 20 ∙ 1/2 = 10 см.
Синусов теорема с примерами
Внимательный читатель заметил, что мы «зашифровали» в этой задаче треугольник из предыдущего примера. Теперь осталось найти его площадь. Для этого берем стандартную формулу площади произвольного треугольника, которая равна половине произведения сторон на синус угла между ними
S = ½ ∙ a ∙ b ∙ sin α
Поскольку нам известны все стороны и все углы, то мы можем выбрать любые из них.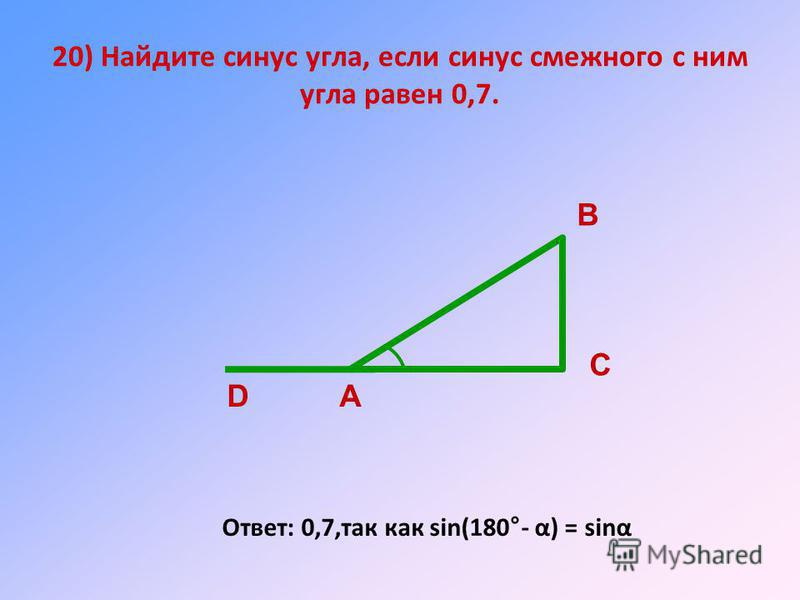 Возьмем стороны АС и АВ, а также угол САВ между ними:
Возьмем стороны АС и АВ, а также угол САВ между ними:
S = ½ ∙ АС ∙АВ ∙ sin 60° = ½ ∙ 20 ∙10 ∙ √3/2 = 50√3 см2.
Примечание: внимательный читатель заметил, что наш треугольник — прямоугольный, так как один из его углов равен 90°. В таком случае можно обойтись без знания синуса угла, вычислив площадь треугольника как половину площади прямоугольника, длина и ширина которого равна катетам треугольника.
S = ½ ∙ ВС ∙АВ = ½ ∙ 10√3 ∙ 10 = 50√3 см2.
Ответ: S = 50√3 см2.
Занимайтесь на курсах ЕГЭ и ОГЭ в паре TwoStu и получите максимум баллов на экзамене:
Владислав Барышников
Эксперт по подготовке к ЕГЭ, ОГЭ и ВПР
Задать вопрос
Закончил Московский физико-технический институт (Физтех) по специальности прикладная физика и математика. Магистр физико-математических наук. Преподавательский стаж более 13 лет. Соучредитель курсов ЕГЭ и ОГЭ в паре TwoStu.
Соучредитель курсов ЕГЭ и ОГЭ в паре TwoStu.
Читайте также:
На синус угла между ними. Теоремы косинусов и синусов
Площадь треугольника равна половине произведения его сторон на синус угла между ними.
Доказательство:
Рассмотрим произвольный треугольник ABC. Пусть в нем сторона BC = a, сторона CA = b и S — площадь этого треугольника. Необходимо доказать, что S = (1/2)*a*b*sin(C) .
Для начала введем прямоугольную систему координат и поместим начало координат в точку С. Расположим нашу систему координат так, чтобы точка B лежала на положительном направлении оси Сх, а точка А имела бы положительную ординату.
Если все выполнить правильно, то должен получится следующий рисунок.
Площадь данного треугольника можно вычислить по следующей формуле: S = (1/2)*a*h , где h — это высота треугольника. В нашем случае высота треугольника h равна ординате точки А, то есть h = b*sin(C).
Учитывая полученные результат, формулу площади треугольника можно переписать следующим образом: S = (1/2)*a*b*sin(C). 2. Найдите сторону AB, если AC = 15 см, угол А = 30˚.
2. Найдите сторону AB, если AC = 15 см, угол А = 30˚.
Положим S — площадь треугольника ABC. По теореме о площади треугольника имеем:
S = (1/2)*AB*AC*sin(A).
Подставим в неё имеющиеся у нас значения:
60 = (1/2)*AB*15*sin30˚ = (1/2)*15*(1/2)*AB=(15/4)*AB.
Отсюда выражаем длину стороны AB: AB = (60*4)/15 = 16.
Сторону треугольника дозволено обнаружить не только по периметру и площади, но и по заданной стороне и углам. Для этого применяются тригонометрические функции – синус и косинус . Задачи с их применением встречаются в школьном курсе геометрии, а также в вузовском курсе аналитической геометрии и линейной алгебры.
Инструкция
1. Если знаменита одна из сторон треугольника и угол между ней и иной его стороной, воспользуйтесь тригонометрическими функциями – синус ом и косинус ом. Представьте себе прямоугольный треугольник НBC , у которого угол? равен 60 градусам. Треугольник НBC показан на рисунке. От того что синус , как знаменито, представляет собой отношение противолежащего катета к гипотенузе, а косинус – отношение прилежащего катета к гипотенузе, для решения поставленной задачи воспользуйтесь дальнейшим соотношением между этими параметрами:sin ?=НB/BCСоответственно, если вы хотите узнать катет прямоугольного треугольника, выразите его через гипотенузу дальнейшим образом:НB=BC*sin ?
От того что синус , как знаменито, представляет собой отношение противолежащего катета к гипотенузе, а косинус – отношение прилежащего катета к гипотенузе, для решения поставленной задачи воспользуйтесь дальнейшим соотношением между этими параметрами:sin ?=НB/BCСоответственно, если вы хотите узнать катет прямоугольного треугольника, выразите его через гипотенузу дальнейшим образом:НB=BC*sin ?
2. Если в условии задачи, напротив, дан катет треугольника, обнаружьте его гипотенузу, руководствуясь дальнейшим соотношением между заданными величинами:BC=НB/sin ?По аналогии обнаружьте стороны треугольника и с применением косинус а, изменив предыдущее выражение дальнейшим образом:cos ?=НC/BC
3. В элементарной математике существует представление теоремы синус ов. Руководствуясь фактами, которые описывает данная теорема, также дозволено обнаружить стороны треугольника. Помимо этого, она разрешает обнаружить стороны треугольника, вписанного в окружность, если знаменит вестим радиус последней.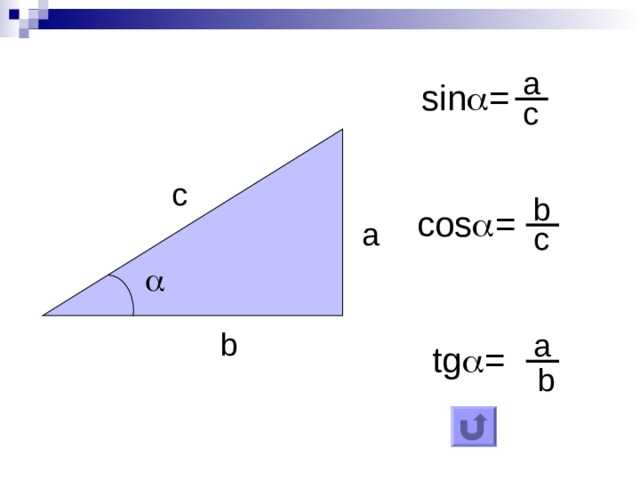 2-2ab*cos ?
2-2ab*cos ?
Геометрическая фигура, состоящая из трёх точек, не принадлежащих одной прямой называемых вершинами, и трёх попарно соединяющих их отрезков, называемых сторонами, именуется треугольником. Существует уйма задач на нахождение сторон и углов треугольника по ограниченному числу начальных данных, одна из таких задач – нахождение стороны треугольника по одной из его сторон и двум углам .
Инструкция
1. Пускай построен треугольник?ABC и знамениты – сторона BC и углы?? и??.Знаменито, что сумма углов всякого треугольника равна 180?, следственно в треугольнике?ABC угол?? будет равен?? = 180? – (?? + ??).Обнаружить стороны AC и AB дозволено применяя теорему синусов, которая гласитAB/sin?? = BC/sin?? = AC/sin?? = 2 * R, где R – радиус описанной около треугольника?ABC окружности,тогда получаемR = BC/sin??,AB = 2 * R * sin??,AC = 2 * R * sin??.Теорему синусов дозволено использовать при всяких данных 2-х углах и стороне.
2. Стороны заданно треугольника дозволено обнаружить, вычислив его площадь по формулеS = 2 * R? * sin?? * sin?? * sin??,где R вычисляется по формулеR = BC/sin??, R – радиус описанной около треугольника?ABC отсюдаТогда сторону AB дозволено обнаружить, вычислив высоту, опущенную на неёh = BC * sin??,отсель по формуле S = 1/2 * h * AB имеемAB = 2 * S/hАналогичным образом дозволено вычислить сторону AC.
3. Если в качестве углов даны внешние углы треугольника?? и??, то обнаружить внутренние углы дозволено с поддержкой соответствующих соотношений?? = 180? – ??,?? = 180? – ??,?? = 180? – (?? + ??).Дальше действуем подобно первым двум пунктам.
Постижение треугольников ведется математиками на протяжении нескольких тысячелетий. Наука о треугольниках – тригонометрия – использует особые величины: синус и косинус.
Прямоугольный треугольник
Изначально синус и косинус появились из-за необходимости рассчитывать величины в прямоугольных треугольниках. Было подмечено, что если значение градусной меры углов в прямоугольном треугольнике не менять, то соотношение сторон, насколько бы эти стороны ни изменялись в длине, остается неизменно идентичным.Именно так и были введены представления синуса и косинуса. Синус острого угла в прямоугольном треугольнике – это отношение противолежащего катета к гипотенузе, а косинус – прилежащего к гипотенузе.
Теоремы косинусов и синусов
Но косинусы и синусы могут использоваться не только в прямоугольных треугольниках. Дабы обнаружить значение тупого либо острого угла, стороны всякого треугольника, довольно применить теорему косинусов и синусов.Теорема косинусов достаточно примитивна: «Квадрат стороны треугольника равен сумме квадратов 2-х других сторон за вычетом удвоенного произведения этих сторон на косинус угла между ними». Существует две трактовки теоремы синусов: малая и расширенная. Согласно малой: «В треугольнике углы пропорциональны противолежащим сторонам». Данную теорему зачастую расширяют за счет свойства описанной около треугольника окружности: «В треугольнике углы пропорциональны противолежащим сторонам, а их отношение равно диаметру описанной окружности».
Дабы обнаружить значение тупого либо острого угла, стороны всякого треугольника, довольно применить теорему косинусов и синусов.Теорема косинусов достаточно примитивна: «Квадрат стороны треугольника равен сумме квадратов 2-х других сторон за вычетом удвоенного произведения этих сторон на косинус угла между ними». Существует две трактовки теоремы синусов: малая и расширенная. Согласно малой: «В треугольнике углы пропорциональны противолежащим сторонам». Данную теорему зачастую расширяют за счет свойства описанной около треугольника окружности: «В треугольнике углы пропорциональны противолежащим сторонам, а их отношение равно диаметру описанной окружности».
Производные
Производная – математический инструмент, показывающий, как стремительно меняется функция касательно метаморфозы ее довода. Производные применяются в алгебре, геометрии, экономике и физике, ряде технических дисциплин. При решении задач требуется знать табличные значения производных тригонометрических функций: синуса и косинуса. Производной синуса является косинус, а косинуса – синус, но со знаком «минус».
Производной синуса является косинус, а косинуса – синус, но со знаком «минус».
Применение в математике
Особенно зачастую синусы и косинусы применяются при решении прямоугольных треугольников и задач, связанных с ними. Удобство синусов и косинусов обнаружило свое отражение и в технике. Углы и стороны было примитивно оценивать по теоремам косинусов и синусов, разбивая трудные фигуры и объекты на «примитивные» треугольники. Инженеры и архитекторы, зачастую имеющие дело с расчетами соотношения сторон и градусных мер, тратили много времени и усилий для вычисления косинусов и синусов не табличных углов. Тогда «на подмогу» пришли таблицы Брадиса, содержащие тысячи значений синусов, косинусов, тангенсов и котангенсов различных углов. В советское время некоторые преподаватели принуждали своих подопечных учить страницы таблиц Брадиса назубок.
Синус является одной из основных тригонометрических функций, применение которой не ограничено одной лишь геометрией. Таблицы вычисления тригонометрических функций, как и инженерные калькуляторы, не всегда под рукой, а вычисление синуса порой нужно для решения различных задач.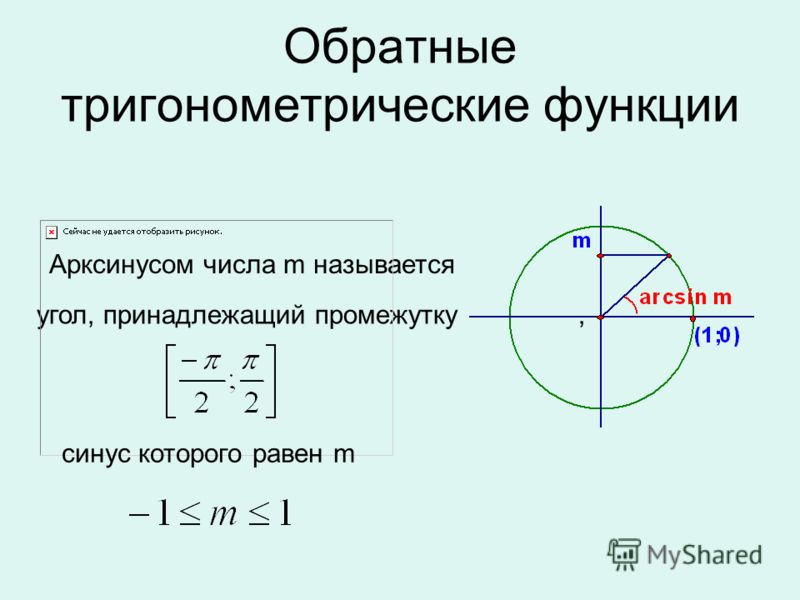 Вообще, вычисление синуса поможет закрепить чертёжные навыки и знание тригонометрических тождеств.
Вообще, вычисление синуса поможет закрепить чертёжные навыки и знание тригонометрических тождеств.
Игры с линейкой и карандашом
Простая задача: как найти синус угла, нарисованного на бумаге? Для решения понадобится обычная линейка, треугольник (или циркуль) и карандаш. Простейшим способом вычислить синус угла можно, разделив дальний катет треугольника с прямым углом на длинную сторону — гипотенузу. Таким образом, сначала нужно дополнить острый угол до фигуры прямоугольного треугольника, прочертив перпендикулярную одному из лучей линию на произвольном расстоянии от вершины угла. Потребуется соблюсти угол именно 90°, для чего нам и понадобится канцелярский треугольник.
Использование циркуля немного точнее, но займёт больше времени. На одном из лучей нужно отметить 2 точки на некотором расстоянии, настроить на циркуле радиус, примерно равный расстоянию между точками, и прочертить полуокружности с центрами в этих точках до получения пересечений этих линий. Соединив точки пересечения наших окружностей между собой, мы получим строгий перпендикуляр к лучу нашего угла, остаётся лишь продлить линию до пересечения с другим лучом.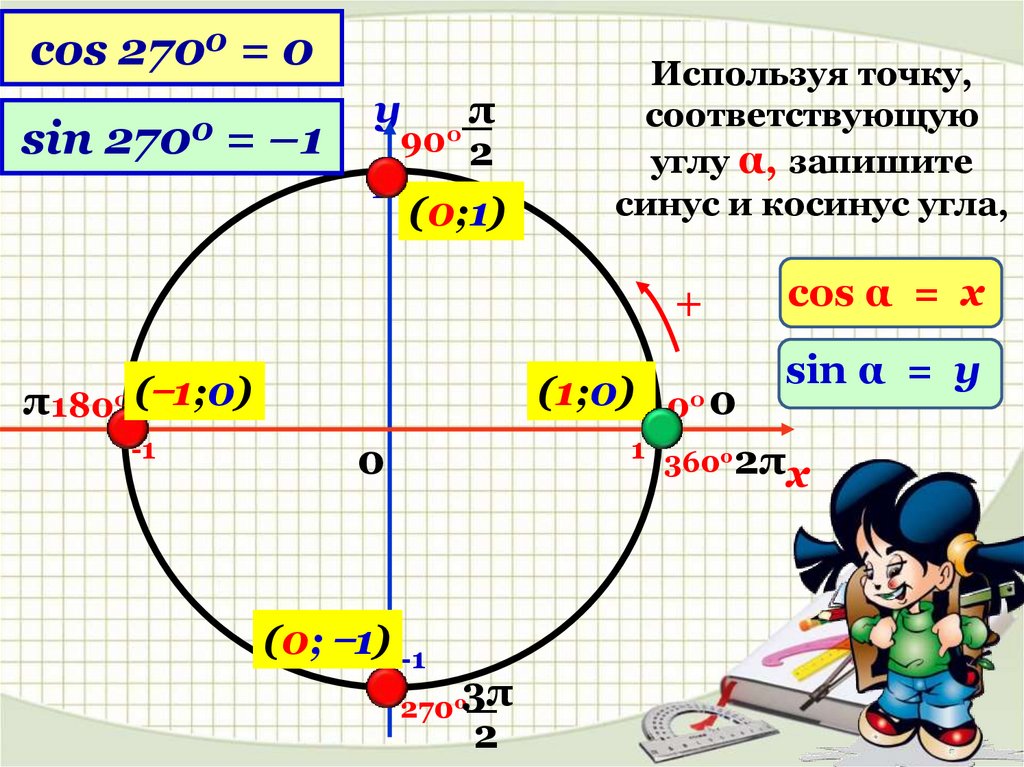
В полученном треугольнике нужно линейкой измерить сторону напротив угла и длинную сторону на одном из лучей. Отношение первого измерения ко второму и будет искомой величиной синуса острого угла.
Найти синус для угла больше 90°
Для тупого угла задача не намного сложнее. Нужно прочертить луч из вершины в противоположную сторону с помощью линейки для образования прямой с одним из лучей интересующего нас угла. С полученным острым углом следует поступать как описано выше, синусы смежных углов, образующих вместе развёрнутый угол 180°, равны.
Вычисление синуса по другим тригонометрическим функциям
Также вычисление синуса возможно, если известны значения других тригонометрических функций угла или хотя бы длины сторон треугольника. В этом нам помогут тригонометрические тождества. Разберём распространённые примеры.
Как находить синус при известном косинусе угла? Первое тригонометрическое тождество, исходящее из теоремы Пифагора, гласит, что сумма квадратов синуса и косинуса одного и того же угла равна единице.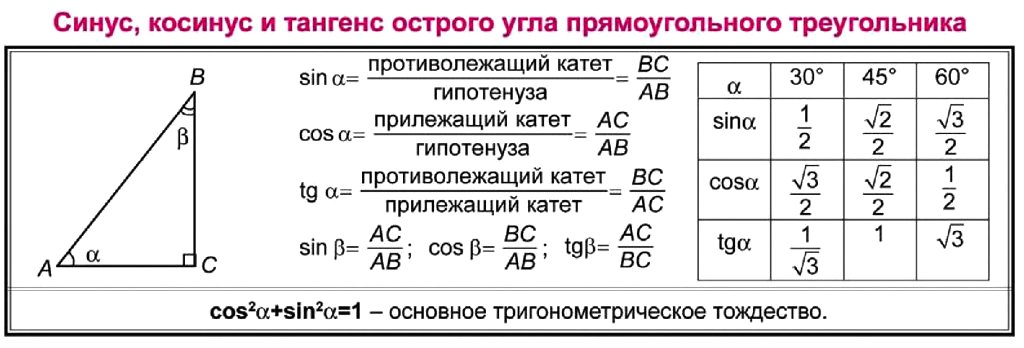
Как находить синус при известном тангенсе угла? Тангенс получают делением дальнего катета на ближний или делением синуса на косинус. Таким образом, синусом будет произведение косинуса на тангенс, а квадратом синуса будет квадрат этого произведения. Заменяем косинус в квадрате на разность между единицей и квадратным синусом согласно первому тригонометрическому тождеству и путём нехитрых манипуляций приводим уравнение к вычислению квадратного синуса через тангенс, соответственно, для вычисления синуса придётся извлечь корень из полученного результата.
Как находить синус при известном котангенсе угла? Значение котангенса можно вычислить, разделив длину ближнего от угла катета на длину дальнего, а также поделив косинус на синус, то есть котангенс — функция, обратная тангенсу относительно числа 1. Для расчёта синуса можно вычислить тангенс по формуле tg α = 1 / ctg α и воспользоваться формулой во втором варианте. Также можно вывести прямую формулу по аналогии с тангенсом, которая будет выглядеть следующим образом.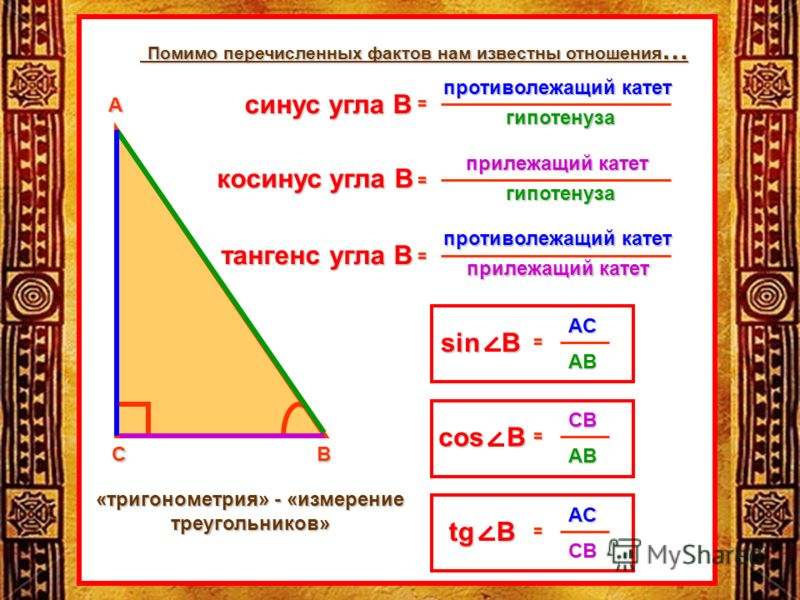
Как находить синус по трём сторонам треугольника
Существует формула для нахождения длины неизвестной стороны любого треугольника, не только прямоугольного, по двум известным сторонам с использованием тригонометрической функции косинуса противолежащего угла. Выглядит она так.
Ну, а синус можно далее рассчитать по косинусу согласно формулам выше.
В жизни нам часто придется сталкиваться с математическими задачами: в школе, в университете, а затем помогая своему ребенку с выполнением домашнего задания. Люди определенных профессий будут сталкиваться с математикой ежедневно. Поэтому полезно запоминать или вспоминать математические правила. В этой статье мы разберем одно из них: нахождение катета прямоугольного треугольника.
Что такое прямоугольный треугольник
Для начала вспомним, что такое прямоугольный треугольник. Прямоугольный треугольник – это геометрическая фигура из трех отрезков, которые соединяют точки, не лежащие на одной прямой, и один из углов этой фигуры равен 90 градусам.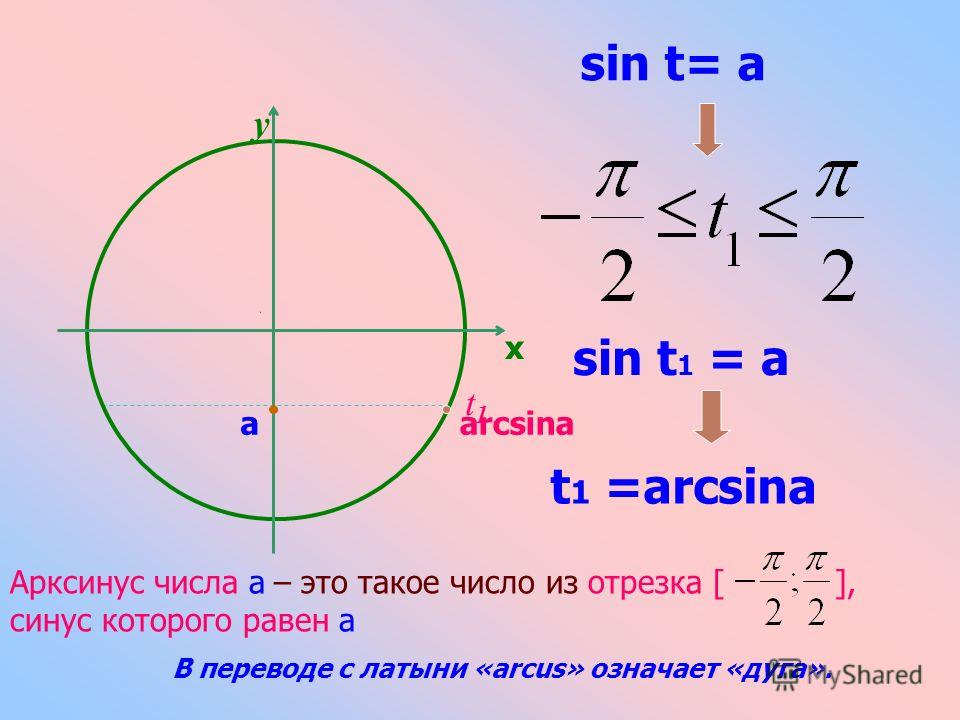 Стороны, образующие прямой угол, называются катетами, а сторона, которая лежит напротив прямого угла – гипотенузой.
Стороны, образующие прямой угол, называются катетами, а сторона, которая лежит напротив прямого угла – гипотенузой.
Находим катет прямоугольного треугольника
Существует несколько способов, позволяющих узнать длину катета. Хотелось бы рассмотреть бы их подробнее.
Теорема Пифагора, чтобы найти катет прямоугольного треугольника
Если нам известны гипотенуза и катет, то мы можем найти длину неизвестного катета по теореме Пифагора. Звучит она так: “Квадрат гипотенузы равен сумме квадратов катетов”. Формула: c²=a²+b², где c – гипотенуза, a и b – катеты. Преобразовываем формулу и получаем: a²=c²-b².
Пример. Гипотенуза равна 5 см, а катет – 3 см. Преобразовываем формулу: c²=a²+b² → a²=c²-b². Далее решаем: a²=5²-3²; a²=25-9; a²=16; a=√16; a=4 (см).
Тригонометрические соотношения, чтобы найти катет прямоугольного треугольника
Также можно найти неизвестный катет, если известны любая другая сторона и любой острый угол прямоугольного треугольника. Есть четыре варианта нахождения катета при помощи тригонометрических функций: по синусу, косинусу, тангенсу, котангенсу. Для решения задач нам поможет таблица, которая находится чуть ниже. Рассмотрим эти варианты.
Есть четыре варианта нахождения катета при помощи тригонометрических функций: по синусу, косинусу, тангенсу, котангенсу. Для решения задач нам поможет таблица, которая находится чуть ниже. Рассмотрим эти варианты.
Найти катет прямоугольного треугольника при помощи синуса
Синус угла (sin) – это отношение противолежащего катета к гипотенузе. Формула: sin=a/c, где а – катет, лежащий против данного угла, а с – гипотенуза. Далее преобразуем формулу и получаем: a=sin*c.
Пример. Гипотенуза равна 10 см, угол А равен 30 градусов. По таблице вычисляем синус угла А, он равен 1/2. Затем по преобразованной формуле решаем: a=sin∠А*c; a=1/2*10; a=5 (см).
Найти катет прямоугольного треугольника при помощи косинуса
Косинус угла (cos) – это отношение прилежащего катета к гипотенузе. Формула: cos=b/c, где b – катет, прилежащий к данному углу, а с – гипотенуза. Преобразуем формулу и получим: b=cos*c.
Пример. Угол А равен 60 градусов, гипотенуза равна 10 см. По таблице вычисляем косинус угла А, он равен 1/2. Далее решаем: b=cos∠A*c; b=1/2*10, b=5 (см).
По таблице вычисляем косинус угла А, он равен 1/2. Далее решаем: b=cos∠A*c; b=1/2*10, b=5 (см).
Найти катет прямоугольного треугольника при помощи тангенса
Тангенс угла (tg) – это отношение противолежащего катета к прилежащему. Формула: tg=a/b, где а – противолежащий к углу катет, а b – прилежащий. Преобразуем формулу и получаем: a=tg*b.
Пример. Угол А равен 45 градусов, гипотенуза равна 10 см. По таблице вычисляем тангенс угла А, он равен Решаем: a=tg∠A*b; a=1*10; a=10 (см).
Найти катет прямоугольного треугольника при помощи котангенса
Котангенс угла (ctg) – это отношение прилежащего катета к противолежащему. Формула: ctg=b/a, где b – прилежащий к углу катет, а – противолежащий. Иначе говоря, котангенс – это “перевернутый тангенс”. Получаем: b=ctg*a.
Пример. Угол А равен 30 градусов, противолежащий катет равен 5 см. По таблице тангенс угла А равен √3. Вычисляем: b=ctg∠A*a; b=√3*5; b=5√3 (см).
Итак, теперь вы знаете, как находить катет в прямоугольном треугольнике. Как видите, это не так уж и сложно, главное – запомнить формулы.
Как видите, это не так уж и сложно, главное – запомнить формулы.
Если в задаче даны длины двух сторон треугольника и угол между ними, то можно применить формулу площади треугольника через синус.
Пример расчета площади треугольника через синус. Даны стороны a = 3, b = 4, и угол γ= 30°. По синус угла в 30° равен 0.5
Площадь треугольника будет равна 3 кв. см.
Также могут быть и другие условия. Если дана длина одной стороны и углы, то для начала нужно вычислить недостающий угол. Т.к. сумма всех углов треугольника равняется 180°, то:
Площадь будет равна половине квадрата стороны, умноженной на дробь. В ее числителе находится произведение синусов прилегающих углов, а в знаменателе синус противолежащего угла. Теперь рассчитываем площадь по следующим формулам:
Например, дан треугольник со стороной a=3 и углами γ=60°, β=60°. Вычисляем третий угол:
Подставляем данные в формулу
Получаем, что площадь треугольника равняется 3,87 кв. см.
см.
II. Площадь треугольника через косинус
Чтобы найти площадь треугольника, нужно знать длины всех сторон. По теореме косинусов можно найти не известные стороны, а уже потом использовать .
По теореме косинусов квадрат неизвестной стороны треугольника равняется сумме квадратов остальных сторон минус удвоенное произведение этих сторон на косинус угла, находящегося между ними.
Из теоремы выводим формулы для поиска длины неизвестной стороны:
Зная как найти недостающую сторону, имея две стороны и угол между ними можно легко посчитать площадь. Формула площади треугольника через косинус помогает легко и быстро найти решение различных задач.
Пример расчета формулы площади треугольника через косинус
Дан треугольник с известными сторонами a = 3, b = 4, и углом γ= 45°. Для начала найдем недостающую сторону с . По косинус 45°=0,7. Для этого подставим данные в уравнение, выведенное из теоремы косинусов.
Теперь используя формулу, найдем
Как компьютер считает синусы — Журнал «Код» программирование без снобизма
Это текст про математику и компьютеры. Если тема интересна, посмотрите также на математический тренажёр Практикума — он бесплатный и интерактивный.
Если тема интересна, посмотрите также на математический тренажёр Практикума — он бесплатный и интерактивный.
А сейчас — про синусы.
Что такое синус и зачем он нужен?
Вульгарное объяснение: синус — это математическая коробка, в которую засовывают любое число, а она в ответ выдаёт числа от −1 до 1. Если эти числа выстроить подряд на некой оси, то получится кривая вот такого вида:
Как читать эту кривую: если затолкать в коробку «sin» число, примерно равное 1,57, то коробка выдаст число, близкое к единице. Если затолкать число 2, на выходе будет примерно 0,909. Если затолкать примерно 3,14 — вернёт примерно 0. Синус от 4,712 даст примерно −0,999. И так дальше: число может быть сколько угодно большим, а синус всегда будет возвращать какие-то дробные значения от −1 до 1.
Это число взято из тригонометрии — то есть из науки, которая занимается углами и сторонами треугольника. В частности, синус описывает отношение сторон прямоугольного треугольника: насколько один из катетов (короткая сторона треугольника) короче, чем гипотенуза (длинная сторона треугольника). Но чаще всего мы знаем не длины сторон, а угол между ними, поэтому в синусы всегда запихивают значения углов.
Но чаще всего мы знаем не длины сторон, а угол между ними, поэтому в синусы всегда запихивают значения углов.
Грубо говоря, вы говорите коробке: «Коробка, у меня тут прямоугольный треугольник. Я смотрю на его острый угол, он равен 30º. Что ты мне на это скажешь»? А коробка отвечает: «Если у тебя угол 30º, то короткая сторона твоего треугольника вдвое короче, чем длинная гипотенуза. Так что sin(30º) = ½».
Это число нужно много где в математике и компьютерах. Например, без синуса невозможно соединить две точки прямой линией на плоскости. Люди это делают без труда с помощью линейки, а компьютеру нужно очень чётко считать, куда поставить пиксель, и для этого нужен синус.
Помимо синуса есть ещё три аналогичные функции — косинус, тангенс и котангенс. Они такие же по принципу работы, но описывают отношения других сторон.
Во многих языках программирования есть встроенная команда нахождения синуса угла — sin(). Внутри этой функции зашита какая-то логика для нахождения этого числа.
Чаще всего, когда не нужна высокая точность, компьютер берёт значения синуса из готовых таблиц — он находит там нужный угол и возвращает значение, ничего не вычисляя. Это быстро и достаточно точно для бытовых вычислений. Вы наверняка использовали его в школе, когда считали синусы по таблице Брадиса.
Но когда нужна высокая точность вычислений (например, 20 знаков после запятой), то синусы и другие тригонометрические функции высчитывают каждый раз с нуля. Для этого используют много разных алгоритмов, и самый простой из них — использование рядов Тейлора.
Таблица Брадиса, по которой компьютер находит значения синусов с точностью 4 знака после запятойЧто такое ряд Тейлора
Брук Тейлор — это английский математик из 17-го века, в честь которого назвали формулу, связывающую значение функции и значение всех её производных в выбранной точке. Если сильно упростить и перевести на понятный язык, то формула будет звучать так:
Представим, что значение функции, например, синуса — это круг, заполненный на 100%. Производная функции в этой точке — это часть круга:
Производная функции в этой точке — это часть круга:
Есть производные первого порядка в этой точке, второго, третьего и так до бесконечности. Каждая производная следующего порядка добавляет в круг сектор поменьше:
Если сложить все производные до бесконечности, то получим полный круг — это и будет значение синуса:
В общем виде ряд Тейлора функции выглядит так:
Для каждой функции ряд Тейлора выглядит по-своему. Для синуса он выглядит так:
Выглядит сложно. Но если разложить эту формулу сумму в понятный вид, она будет выглядеть так:
Восклицательный знак — это факториал. Это просто произведение всех целых чисел до этого числа. Например: 5! = 1 × 2 × 3 × 4 × 5 = 120.
Получается, что компьютеру для нахождения синуса достаточно использовать умножение — и для факториала, и для возведения в степень. Зная это, можно написать простой алгоритм.
Точность расчётов
Особенность ряда Тейлора в том, что это бесконечный ряд — а значит, и вычисления тоже придётся делать бесконечно. Чтобы обойти это ограничение, используют погрешность — с какой точностью нам нужно посчитать значение формулы. Для этого делают так:
Чтобы обойти это ограничение, используют погрешность — с какой точностью нам нужно посчитать значение формулы. Для этого делают так:
- Определяем точность, например 7 знаков после запятой.
- Минимальное число из 7 знаков после запятой — это 0,0000001. Это и будет наша погрешность.
- Считаем очередное слагаемое ряда Тейлора.
- Если это слагаемое меньше нашей погрешности — прибавляем и останавливаемся, потому что мы достигли нужного результата. Если не меньше — продолжаем.
С таким подходом можно найти синус любого угла с любой точностью, главное, чтобы у компьютера хватило памяти на все эти вычисления.
Радианы
Чтобы использовать ряд Тейлора для вычисления синуса, нам нужно перевести градусы в радианы. Радиан — это мера измерения углов в тригонометрии, которая привязана к числу π.
Радианы с углами связаны так:
1 радиан = 180/π градусов
Получается, что для того, чтобы перевести углы в градусах в радианы, нам нужно градусы разделить на 180 и умножить на π.
Теперь мы знаем всё, чтобы написать код вычисления синуса на Python
Пишем код
Логика алгоритма будет такая:
- спрашиваем градусы;
- спрашиваем погрешность, с которой нужно вычислить синус;
- в цикле считаем очередное слагаемое ряда Тейлора и прибавляем его к общей сумме;
- если очередное слагаемое меньше погрешности — останавливаемся и выводим результат.
Читайте комментарии, чтобы разобраться в коде, а потом запустите его у себя, чтобы проверить результаты:
# импортируем математическую библиотеку, чтобы взять оттуда модуль и число пи
import math
# объявляем свою функцию, которая посчитает синус
def computerSinus (x,n):
# переводим градусы в радианы
x = x/180*math.pi
# для проверки выведем результат, который посчитает компьютер
print(str(math.sin(x)) + " — результат вычислений встроенного синуса")
# сразу берём x как первое слагаемое ряда Тейлора
q = x
# сумма ряда на старте равна нулю
s = 0
# порядковый номер слагаемого в ряду Тейлора
i = 1
# пока очередное слагаемое больше погрешности — цикл работает
while abs(q) > n:
# добавляем слагаемое к общей сумме
s = s + q
# вычисляем следующее слагаемое
q = q* (-1) * (x*x) / ((2*i+1) * (2*i))
# увеличиваем порядковый номер слагаемого в ряду Тейлора
i = i+1
# возвращаем сумму как результат работы функции
return s
# запрашиваем стартовые значения
x = float(input("Введите градусы: "))
n = float(input("Введите погрешность: "))
# выводим результат, который мы посчитали сами
print(str(computerSinus(x,n)) + " — синус, который мы посчитали")Что дальше
В следующий раз попробуем сделать то же самое с квадратным корнем — посмотрим, как компьютер сможет посчитать его без таблиц.
Текст:
Михаил Полянин
Редактор:
Максим Ильяхов
Художник:
Алексей Сухов
Корректор:
Ирина Михеева
Вёрстка:
Кирилл Климентьев
Соцсети:
Виталий Вебер
Значение, Синонимы, Определение, Предложения . Что такое синус
Вскрыл мозговую оболочку и обнажил общий у них венозный синус. | |
Экг, нормальный синус, нормальные интервалы. | |
Доктор Рэмзи добралась до менингиомы через сагиттальный синус. | |
А этот синус кавернозный это опасно? | |
Слив вниз наоборот поперечный синус и вниз сигмовидной к внутренней яремной? | |
Плюс одна пятая a… на синус два ax. (Заряд батареи…) | |
И здесь всё становится ещё интереснее, потому что sin это ещё и синус. | |
Которая будет использована как новый сагиттальный синус. | |
Сейчас я разделяю сагиттальный синус. | |
Что такое затылочный венозный синус? | |
Проблема в том, что прут давит на затылочный венозный синус. | |
Просто небольшой ушиб, но реберно-диафрагмальный синус в норме. | |
Один общий сагиттальный синус снабжает мозг кровью. | |
Синус угла A равен отношению BC к AB и равен отношению перпендикуляра к гипотенузе. | |
Затемнённый синус и и повреждение решётчатой пластины. | |
В худшем случае прут может разорвать синус, и он умрет за одну секунду. | |
Окончательный урогенитальный синус состоит из каудальной цефальной части и промежуточного узкого канала-тазовой части. | |
Установлено, что атрезия влагалища возникает тогда, когда урогенитальный синус не способствует формированию нижней части влагалища. | |
x86 также включает в себя ряд трансцендентных функций, включая синус, Косинус, тангенс, арктангенс, возведение в степень с основанием 2 и логарифмы к основаниям 2, 10 или e. | |
В дифференциальных уравнениях функция eix часто используется для упрощения решений, даже если конечным ответом является реальная функция, включающая синус и косинус. | |
Синус между основаниями боковых чашелистиков загибается внутрь и имеет небольшую выемку в центре. | |
От головы до хвоста это инфундибулум, бульбус Кордис, примитивный желудочек, примитивное предсердие и синус венозный. | |
Первоначально вся венозная кровь поступает в венозный синус и движется от хвоста к голове к артериальному стволу. | |
Кавернозный синус находится в полости черепа, между слоями мозговых оболочек и является главным каналом венозного дренажа из головного мозга. | |
Если кавернозный синус заражен, это может привести к свертыванию крови внутри синуса, что приведет к тромбозу кавернозного синуса. | |
Амиопатический ДМ, также называемый ДМ синус миозит, был назван в 2002 году. | |
Синус этого угла можно записать как длину его противоположного отрезка, разделенного гипотенузой прямоугольного треугольника. | |
Использование компьютерной томографии в сложных случаях также помогает хирургу идентифицировать и избежать жизненно важных структур, таких как Нижний альвеолярный нерв и синус. | |
Для угла целого числа градусов синус и косинус могут быть выражены через квадратные корни и кубический корень нереального комплексного числа. | |
Для угла, который измеряется в градусах, является рациональным числом, синус и косинус являются алгебраическими числами, которые могут быть выражены в терминах n-го корня. | |
Версин или версированный синус-это тригонометрическая функция, найденная в некоторых из самых ранних тригонометрических таблиц. | |
Версин или версированный синус-это тригонометрическая функция, уже фигурирующая в некоторых из самых ранних тригонометрических таблиц. | |
С другой стороны, искомый синус θ-это расстояние CD от центра хорды до центра дуги. | |
Исторически сложилось так, что искомый синус считался одной из важнейших тригонометрических функций. | |
Плевиг и Клигман добавили к своей триаде прыщей еще один элемент-пилонидальный синус. | |
Для угла, который измеряется в градусах, не является рациональным числом, тогда либо угол, либо синус и косинус являются трансцендентными числами. | |
Сонный синус чувствителен к изменениям давления в артериальной крови на этом уровне. | |
Каротидный синус-это рефлекторная область сонной артерии, состоящая из барорецепторов, контролирующих артериальное давление. | |
Нейроны, иннервирующие каротидный синус, Центрально проецируются на одиночное ядро в мозговом веществе ствола головного мозга. | |
Субкапсулярный синус сливается в трабекулярные синусы и, наконец, в медуллярные синусы. | |
Из этого следует, что тангенс тригонометрической функции 60° равен √3, а синус 60° и косинус 30° равны √3/2. | |
Они входят в череп через сонный канал, проходят через кавернозный синус и входят в субарахноидальное пространство. | |
Кровь стекает из внешнего мозга в большой верхний сагиттальный синус, который находится в средней линии на вершине мозга. | |
Клиновидный синус снабжается задними этмоидальными нервами. | |
Почечный синус в совокупности содержит почечную лоханку и чашечки и отделяет эти структуры от почечно-мозговой ткани. | |
Задний аортальный синус не дает начало коронарной артерии. | |
Урогенитальный синус можно разделить на три составные части. | |
Венозный синус соединяется с кровообращением плода. | |
От головы до хвоста это усеченный артериус, бульбус Кордис, примитивный желудочек, примитивное предсердие и синус венозный. | |
В середине четвертой недели венозный синус получает венозную кровь от полюсов правого и левого синуса. | |
При исчезновении левой общей кардинальной вены на десятой неделе остаются только косая Вена левого предсердия и коронарный синус. | |
Кавернозный синус — это один из дуральных венозных синусов головы. | |
Эти нервы, за исключением CN V2, проходят через кавернозный синус, чтобы войти в вершину орбиты через верхнюю орбитальную щель. | |
Поражения, поражающие кавернозный синус, могут поражать изолированные нервы или все проходящие через него нервы. | |
Косой разрез через кавернозный синус. | |
Инфекция может распространиться на контралатеральный кавернозный синус в течение 24-48 часов после первоначального появления. | |
Эта часть артерии известна как каротидный синус или сонная луковица. | |
В результате инфекция лица может распространиться на кавернозный синус и крыловидное венозное сплетение. | |
Это может привести к повреждению нервов, проходящих через кавернозный синус. | |
Верхний петросальный синус — это один из дуральных венозных синусов, расположенных под головным мозгом. | |
Он получает кровь из кавернозного синуса и проходит назад и в стороны, чтобы стекать в поперечный синус. | |
Синус получает некоторые мозжечковые вены, нижние мозговые вены и вены из барабанной полости. | |
Каждый нижний петросальный синус дренирует кавернозный синус во внутреннюю яремную вену. | |
Это представление указывает либо на сжатие структур в верхней орбитальной щели, либо на кавернозный синус. | |
Важность этого пассажа заключается в том, что инфицированный тромб из экстракраниального источника может попасть в кавернозный синус. | |
Дуральные артериовенозные свищи I типа снабжаются менингеальными артериями и сливаются в менингеальную вену или дуральный венозный синус. | |
Как правило, это происходит потому,что синус имеет препятствие оттока. | |
Клиновидный синус — это один из четырех парных околоносовых пазух, которые содержатся в теле клиновидной кости. | |
Если опухоль распространится латерально, то кавернозный синус и все составляющие его нервы могут оказаться в опасности. | |
Эта борозда соединяет кавернозный синус и внутреннюю сонную артерию, причем последняя окружена сплетением симпатических нервов. | |
| Другие результаты | |
Прослеживая историю вымирания LINE и SINE у сигмодонтных грызунов | Мобильная ДНК
- Исследования
- Открытый доступ
- Опубликовано:
- Лэй Ян 1,2 ,
- ЛуЭнн Скотт 1,2 и
- Холли А.
 Вичман 1,2
Вичман 1,2
Мобильная ДНК том 10 , Номер статьи: 22 (2019) Процитировать эту статью
2020 Количество доступов
5 Цитаты
13 Альтметрический
Сведения о показателях
Abstract
Исходная информация
Ретротранспозоны L1 эволюционировали совместно со своими хозяевами на протяжении всей истории млекопитающих и в настоящее время составляют ~ 20% генома млекопитающих. Ретротранспозоны B1 зависят от L1 для ретротранспозиции и охватывают эволюционную историю грызунов с момента их излучения. Было обнаружено, что L1 потеряли свою активность у группы южноамериканских грызунов, Sigmodontinae, и инактивация B1 предшествовала исчезновению L1 в той же группе. Следовательно, базальная группа сигмодонтинов имеет активные L1, но неактивные B1 и производная клада имеют как неактивные L1, так и B1. Было высказано предположение, что B1 вымерли в течение длительного периода покоя L1 и что L1 впоследствии вновь появились в базальной группе.
Ретротранспозоны B1 зависят от L1 для ретротранспозиции и охватывают эволюционную историю грызунов с момента их излучения. Было обнаружено, что L1 потеряли свою активность у группы южноамериканских грызунов, Sigmodontinae, и инактивация B1 предшествовала исчезновению L1 в той же группе. Следовательно, базальная группа сигмодонтинов имеет активные L1, но неактивные B1 и производная клада имеют как неактивные L1, так и B1. Было высказано предположение, что B1 вымерли в течение длительного периода покоя L1 и что L1 впоследствии вновь появились в базальной группе.
Результаты
Здесь мы исследуем эволюционную историю L1 и B1 у сигмодонтных грызунов и показываем, что активность L1 продолжалась до тех пор, пока L1-вымершая клада и базальная группа не дивергировались. После разделения у L1 был небольшой всплеск активности в первой группе, за которым последовало угасание. В основной группе активность изначально была низкой, но затем последовало резкое увеличение активности L1. Мы обнаружили, что последняя волна ретротранспозиции B1 была большой и, вероятно, предшествовала расколу между двумя ветвями грызунов.
Мы обнаружили, что последняя волна ретротранспозиции B1 была большой и, вероятно, предшествовала расколу между двумя ветвями грызунов.
Выводы
Учитывая, что L1 стабильно ретротранспозировали в течение времени, соответствующего угасанию B1, и что всплеск активности B1, предшествующий угасанию B1, был большим, мы заключаем, что угасание B1 не было результатом покоя L1. Скорее, всплеск активности B1 мог способствовать вымиранию L1 как за счет конкуренции с L1, так и за счет сильного избирательного давления на хозяина с целью контроля ретротранспозиции.
Исходная информация
LINE (длинные перемежающиеся элементы) являются автономными ретротранспозонами, не относящимися к LTR. Они проходят через интермедиат РНК, но имеют негомологичные концы и используют обратную транскрипцию, ориентированную на мишень [1]. L1 (LINE-1) является наиболее успешным семейством LINE у плацентарных млекопитающих [2] и составляет ~ 20% генома млекопитающих [3,4,5,6,7].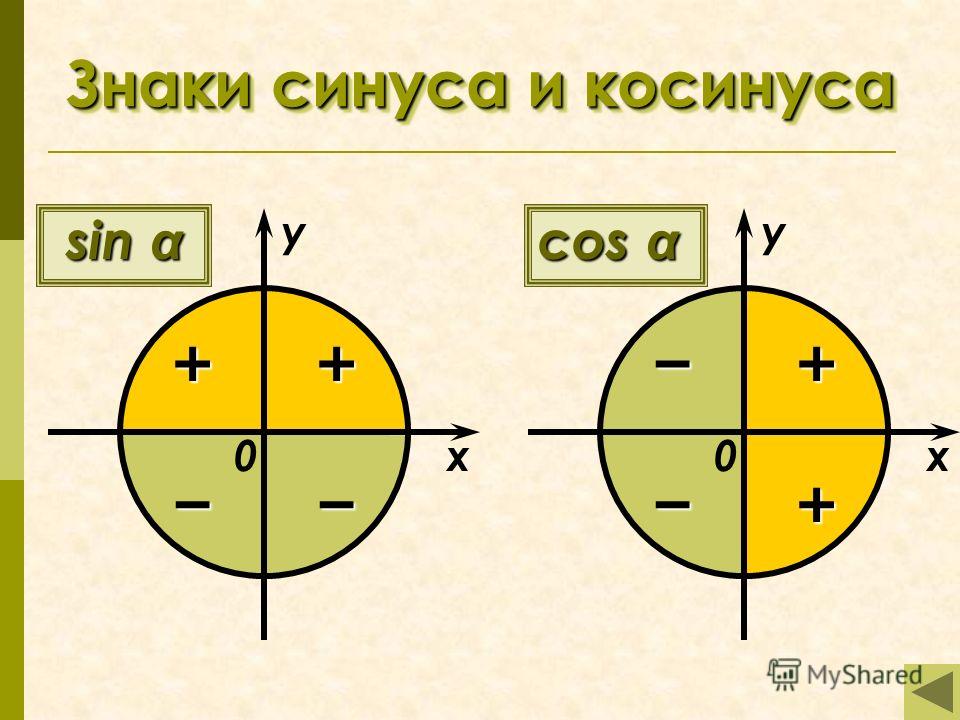 Функциональный полноразмерный L1 обычно имеет длину 6000–7000 п.н. и состоит из 5′-нетранслируемой области (5’UTR), содержащей промотор РНК-полимеразы II, двух неперекрывающихся открытых рамок считывания (ORF), известных как ORF1 и ORF2, и 3’UTR, за которой следует полиаденозиновая последовательность [8]. Структура L1 может различаться у разных млекопитающих, особенно в 5’UTR и ORF1 [5]. Белки, кодируемые ORF, строго необходимы для ретротранспозиции L1 и очень цис -преимущественно [9, 10]. L1 богаты аденозином (~ 40%) на своей кодирующей цепи, что приводит к предвзятому использованию кодонов по сравнению с генами-хозяевами [11, 12], дефектам элонгации [13] и преждевременному сплайсингу РНК [14]. Это A-богатство способствует неэффективности ретротранспозиции L1 и предполагается, что оно регулирует соседние гены [13].
Функциональный полноразмерный L1 обычно имеет длину 6000–7000 п.н. и состоит из 5′-нетранслируемой области (5’UTR), содержащей промотор РНК-полимеразы II, двух неперекрывающихся открытых рамок считывания (ORF), известных как ORF1 и ORF2, и 3’UTR, за которой следует полиаденозиновая последовательность [8]. Структура L1 может различаться у разных млекопитающих, особенно в 5’UTR и ORF1 [5]. Белки, кодируемые ORF, строго необходимы для ретротранспозиции L1 и очень цис -преимущественно [9, 10]. L1 богаты аденозином (~ 40%) на своей кодирующей цепи, что приводит к предвзятому использованию кодонов по сравнению с генами-хозяевами [11, 12], дефектам элонгации [13] и преждевременному сплайсингу РНК [14]. Это A-богатство способствует неэффективности ретротранспозиции L1 и предполагается, что оно регулирует соседние гены [13].
SINE (короткие вкрапленные элементы) представляют собой относительно короткие неавтономные ретротранспозоны, не относящиеся к LTR. SINEs не кодируют белки для их собственной ретротранспозиции и зависят от обратной транскриптазы, кодируемой другими мобильными элементами, такими как LINEs [15, 16]. Хотя L1 сильно cis -preferential [9, 10], SINE могут использовать кодируемые L1 белки для своей собственной ретротранспозиции [15,16,17], а белка ORF2 L1 достаточно для управления ретротранспозицией B1. Несмотря на свою короткую длину, SINE составляют ~ 10% типичного генома млекопитающих из-за их большого числа копий [3, 4]. Среди ~ 70 семейств SINE, обнаруженных у млекопитающих [18], B1 является наиболее распространенным у мышей [4] и, возможно, у большинства видов грызунов [19]. B1 происходят от РНК-компонента сигнальной частицы 7SL РНК [20, 21] и имеют общие черты со своими предками. Функциональный B1 имеет длину ~ 150 bp и транскрибируется РНК-полимеразой III с помощью двух боксов, связывающих факторы транскрипции [22, 23]. Последовательности B1 богаты сайтами CpG, которые метилированы и, таким образом, склонны к мутациям в геномах млекопитающих [24], а повышенная частота мутаций выражена по сравнению с A-богатыми L1 [25, 26].
Хотя L1 сильно cis -preferential [9, 10], SINE могут использовать кодируемые L1 белки для своей собственной ретротранспозиции [15,16,17], а белка ORF2 L1 достаточно для управления ретротранспозицией B1. Несмотря на свою короткую длину, SINE составляют ~ 10% типичного генома млекопитающих из-за их большого числа копий [3, 4]. Среди ~ 70 семейств SINE, обнаруженных у млекопитающих [18], B1 является наиболее распространенным у мышей [4] и, возможно, у большинства видов грызунов [19]. B1 происходят от РНК-компонента сигнальной частицы 7SL РНК [20, 21] и имеют общие черты со своими предками. Функциональный B1 имеет длину ~ 150 bp и транскрибируется РНК-полимеразой III с помощью двух боксов, связывающих факторы транскрипции [22, 23]. Последовательности B1 богаты сайтами CpG, которые метилированы и, таким образом, склонны к мутациям в геномах млекопитающих [24], а повышенная частота мутаций выражена по сравнению с A-богатыми L1 [25, 26].
И L1, и B1 имеют долгую историю совместной эволюции с геномами своих хозяев.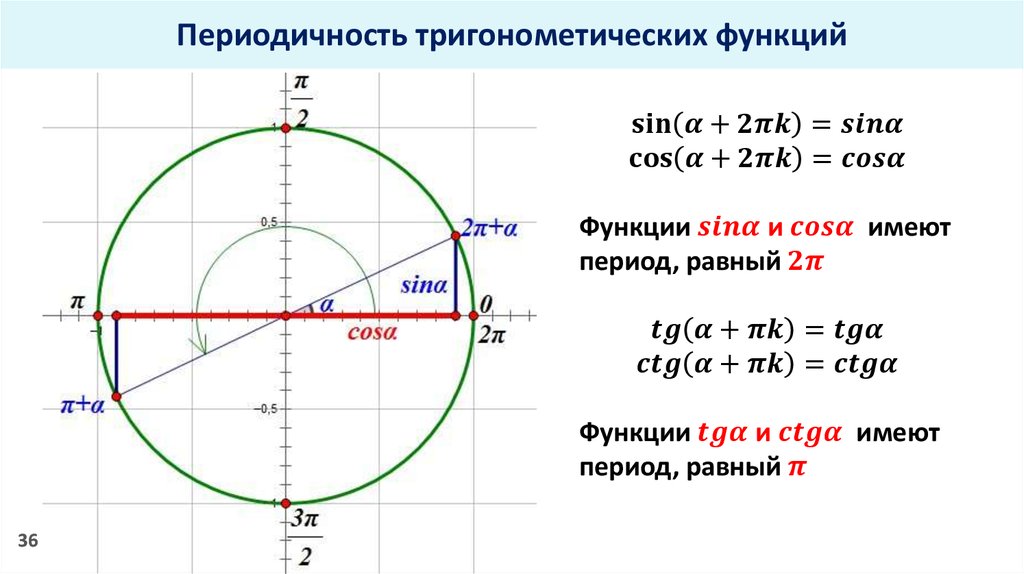 В отличие от некоторых ретротранспозонов LTR [27, 28], не существует известного целевого механизма вырезания L1, и, таким образом, L1 сохраняются в геноме, если они не удаляются неспецифическими механизмами. L1 обнаружены у всех плацентарных млекопитающих и сумчатых [2, 5, 29]. L1 млекопитающих развиваются как главные линии, так что одна или несколько линий ответственны за полную ретротранспозицию в коротком временном окне [30,31,32,33]. Новые основные элементы заменяют старые, в конечном итоге преобладая над ретротранспозицией, и этот процесс замены происходит периодически. B1 моложе L1, возникли незадолго до расхождения общего предка грызунов, ~ 65 млн лет назад [34], и специфичны для грызунов. Другие SINE, включая элементы B2, B4 и ID, также присутствуют в геномах грызунов [19].]. Семейства SINE взаимодействовали с L1 более 100 млн лет назад, а ископаемые остатки вымерших семейств SINE обнаруживаются в хорошо охарактеризованных геномах млекопитающих [18, 35]. Несмотря на то, что на них нацелено множество рестрикционных механизмов хозяина, L1 и B1 составляют примерно четверть типичного генома грызунов [4, 7].
В отличие от некоторых ретротранспозонов LTR [27, 28], не существует известного целевого механизма вырезания L1, и, таким образом, L1 сохраняются в геноме, если они не удаляются неспецифическими механизмами. L1 обнаружены у всех плацентарных млекопитающих и сумчатых [2, 5, 29]. L1 млекопитающих развиваются как главные линии, так что одна или несколько линий ответственны за полную ретротранспозицию в коротком временном окне [30,31,32,33]. Новые основные элементы заменяют старые, в конечном итоге преобладая над ретротранспозицией, и этот процесс замены происходит периодически. B1 моложе L1, возникли незадолго до расхождения общего предка грызунов, ~ 65 млн лет назад [34], и специфичны для грызунов. Другие SINE, включая элементы B2, B4 и ID, также присутствуют в геномах грызунов [19].]. Семейства SINE взаимодействовали с L1 более 100 млн лет назад, а ископаемые остатки вымерших семейств SINE обнаруживаются в хорошо охарактеризованных геномах млекопитающих [18, 35]. Несмотря на то, что на них нацелено множество рестрикционных механизмов хозяина, L1 и B1 составляют примерно четверть типичного генома грызунов [4, 7]. Например, в геноме мыши всего ~ 599 000 копий L1, отвечающих за ~ 19% генома [4], из которых ~ 3000 копий являются потенциально функциональными [36], и ~ 564 000 копий B1, ответственных за ~ 3% генома [4].
Например, в геноме мыши всего ~ 599 000 копий L1, отвечающих за ~ 19% генома [4], из которых ~ 3000 копий являются потенциально функциональными [36], и ~ 564 000 копий B1, ответственных за ~ 3% генома [4].
LINE и SINE оказывают значительное влияние на геном млекопитающих, хотя традиционно их рассматривали как «мусорную ДНК». Поскольку LINE и SINE, включая L1 и B1, ретротранспонируют и рекомбинируют, они привносят нестабильность генома [37] и вызывают заболевание [38]. Эти элементы могут иногда кооптироваться хозяином для выполнения определенных функций хозяина, таких как их предполагаемая роль в нейропластичности [39, 40], инактивации Х-хромосомы [41, 42], регуляторных функциях [43, 44] и Репарация разрывов ДНК [45]. Наша текущая картина эффектов ретротранспозиции не принимает во внимание, какой эффект может иметь замалчивание LINE и SINE на эти предполагаемые функции.
Поскольку ретротранспозиция L1 находится под строгим контролем множественных защитных механизмов хозяина [46], для хозяина может показаться разумным время от времени побеждать в эволюционной гонке вооружений с помощью L1, что приводит к потере активности L1 (вымирание L1).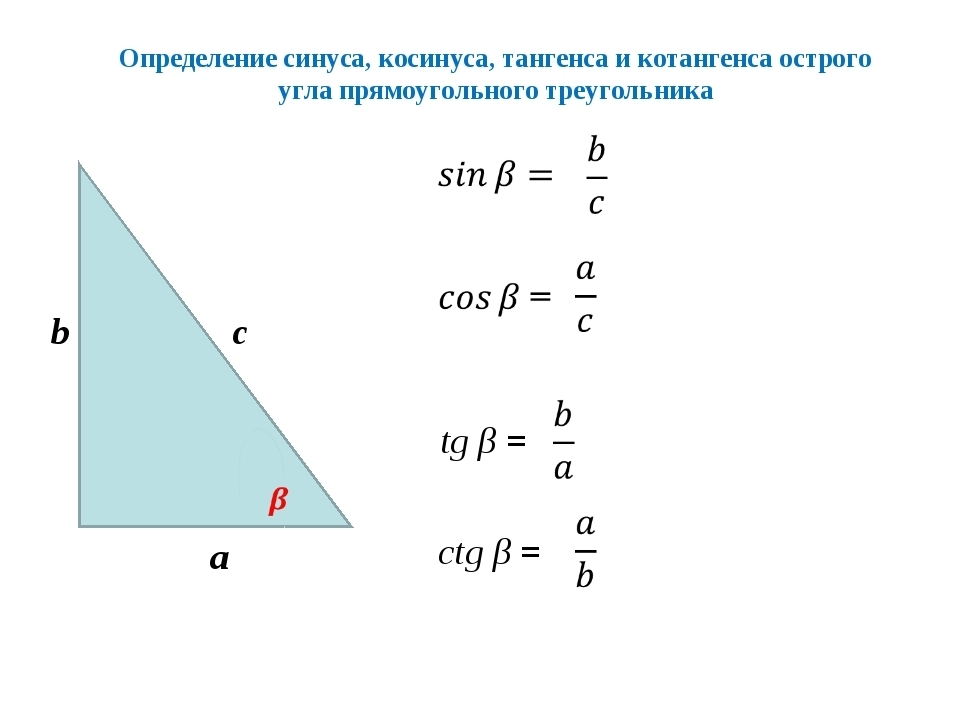 Тем не менее, вымирания L1 относительно редки. Факторы, способствующие редкости вымирания L1, могут быть, помимо прочего, кооптацией L1 для определенных важных функций хозяина. Неизвестно, что L1 млекопитающих двигаются горизонтально, хотя древние L1, возможно, были способны делать это, учитывая наличие других не-LTR ретротранспозонов [47]. Маловероятная замена L1 горизонтальным движением будет очевидна как несоответствие между филогении L1 и филогении хозяина. Следовательно, вымирание млекопитающих L1 затронет все производные виды-хозяева. Здесь следует отметить два фактора. Во-первых, клады с ранним вымиранием L1 могли дать начало крупным линиям млекопитающих без активности L1 и быть легко обнаруженными как из-за количества затронутых видов, так и из-за ухудшения остаточных последовательностей в геноме. Во-вторых, недавние вымирания будет трудно отличить от периодов покоя L1. Чтобы уточнить термины, связанные с потерей активности L1 в этой работе, мы называем период низкой активности L1 «затишьем», а полную потерю активности L1 — «угасанием».
Тем не менее, вымирания L1 относительно редки. Факторы, способствующие редкости вымирания L1, могут быть, помимо прочего, кооптацией L1 для определенных важных функций хозяина. Неизвестно, что L1 млекопитающих двигаются горизонтально, хотя древние L1, возможно, были способны делать это, учитывая наличие других не-LTR ретротранспозонов [47]. Маловероятная замена L1 горизонтальным движением будет очевидна как несоответствие между филогении L1 и филогении хозяина. Следовательно, вымирание млекопитающих L1 затронет все производные виды-хозяева. Здесь следует отметить два фактора. Во-первых, клады с ранним вымиранием L1 могли дать начало крупным линиям млекопитающих без активности L1 и быть легко обнаруженными как из-за количества затронутых видов, так и из-за ухудшения остаточных последовательностей в геноме. Во-вторых, недавние вымирания будет трудно отличить от периодов покоя L1. Чтобы уточнить термины, связанные с потерей активности L1 в этой работе, мы называем период низкой активности L1 «затишьем», а полную потерю активности L1 — «угасанием». Учитывая большое филогенетическое влияние ранних вымираний, можно ожидать, что L1 в конечном итоге вымрут в геномах большинства млекопитающих, и все же L1 сохранялись на протяжении всей истории эволюции их плацентарных млекопитающих и сумчатых хозяев. Таким образом, либо большинство вымираний L1 являются либо недавними, либо редкими, либо линии млекопитающих, подвергшиеся древним вымираниям L1, не сохраняются, либо они дают начало нескольким новым видам. Понимание динамики вымирания L1 будет так же важно, как и понимание динамики активности L1, для выяснения влияния L1 на эволюцию генома млекопитающих.
Учитывая большое филогенетическое влияние ранних вымираний, можно ожидать, что L1 в конечном итоге вымрут в геномах большинства млекопитающих, и все же L1 сохранялись на протяжении всей истории эволюции их плацентарных млекопитающих и сумчатых хозяев. Таким образом, либо большинство вымираний L1 являются либо недавними, либо редкими, либо линии млекопитающих, подвергшиеся древним вымираниям L1, не сохраняются, либо они дают начало нескольким новым видам. Понимание динамики вымирания L1 будет так же важно, как и понимание динамики активности L1, для выяснения влияния L1 на эволюцию генома млекопитающих.
В литературе было предложено несколько случаев вымирания L1 [48, 49, 50, 51, 52, 53, 54, 55, 56], и два из них являются событиями глубокого вымирания, которые охватывают основные группы млекопитающих [50, 51]. ,52]. Одно из крупных вымираний L1 произошло в большой группе южноамериканских грызунов и включает большинство видов Sigmodontinae. Sigmodontinae — это подсемейство семейства Cricetidae, включающее примерно 377 видов, классифицированных в 74 рода в девяти трибах (рис. 1) [58], и, таким образом, включает 7–8% от предполагаемых 5000 видов млекопитающих [59].]. Учитывая, что ретротранспозиция B1 зависит от ретротранспозиции L1, ожидается, что B1 должны терять свою активность одновременно с L1. Однако вымирание B1 у Sigmodontinae, по-видимому, предшествовало исчезновению L1, основываясь на выборках из 14 родов в пяти трибах [50, 51, 52], где базальный род Sigmodon несет неактивный B1 и активный L1, а потомки родов несут оба неактивны L1 и B1 (рис. 1). Также было показано, что потеря активности L1 и B1 следует за экспансией группы эндогенных ретровирусов [60, 61].
1) [58], и, таким образом, включает 7–8% от предполагаемых 5000 видов млекопитающих [59].]. Учитывая, что ретротранспозиция B1 зависит от ретротранспозиции L1, ожидается, что B1 должны терять свою активность одновременно с L1. Однако вымирание B1 у Sigmodontinae, по-видимому, предшествовало исчезновению L1, основываясь на выборках из 14 родов в пяти трибах [50, 51, 52], где базальный род Sigmodon несет неактивный B1 и активный L1, а потомки родов несут оба неактивны L1 и B1 (рис. 1). Также было показано, что потеря активности L1 и B1 следует за экспансией группы эндогенных ретровирусов [60, 61].
Филогения сигмодонтиновых грызунов. Активность L1 и B1, отображаемая в филогении, показывает, что исчезновение L1 предшествовало исчезновению B1. Филогения основана на Schenk et al. [57]. Таксоны — это роды, отобранные для выборки; трибы указаны справа от таксонов. Восемь из девяти триб и 12 из 14 родов, отобранных Rinehart et al. [52]. Активность L1 и B1 каждого таксона показана цветом: черный, активные L1 и B1; темно-синий, активный L1 и неактивный B1; темно-красный, неактивный L1 и неактивный B1; светло-синий, активность L1 не может быть установлена; и, светло-красный, L1 и B1 можно сделать вывод, что они неактивны. «+» соответствует активным L1 и B1, а «o» соответствует неактивным L1 и B1
[52]. Активность L1 и B1 каждого таксона показана цветом: черный, активные L1 и B1; темно-синий, активный L1 и неактивный B1; темно-красный, неактивный L1 и неактивный B1; светло-синий, активность L1 не может быть установлена; и, светло-красный, L1 и B1 можно сделать вывод, что они неактивны. «+» соответствует активным L1 и B1, а «o» соответствует неактивным L1 и B1
Изображение в натуральную величину
Ранее предполагалось, что L1 может находиться в длительном затишье в качестве «стелс-драйвера» [62], и что угасание B1 может произойти в этот период затишья L1 [52]. Поскольку B1 более склонны к мутациям, чем средняя последовательность, из-за обогащенного содержания CpG [24], Rinehart et al. [52] предположили, что B1 не может ретротранспонировать с достаточно высокой скоростью во время покоя L1, чтобы заменить свои активные копии, накапливая истощающие мутации быстрее, чем L1. Когда у Sigmodontini появилось более активное семейство L1, было высказано предположение, что B1 слишком деградировал для ретротранспозиции, что привело к исчезновению B1 даже при наличии высокой активности L1.
В этом исследовании мы изучаем историю эволюции L1 и B1, охватывающую время их исчезновения и излучения существующих видов Sigmodontinae (рис. 1). Поскольку группа, несущая вымершие L1 и B1 (Oryzomyalia, рис. 1), имеет общего предка, мы использовали болотную рисовую крысу Oryzomys palustris для представления этой группы, далее именуемой «L1-вымершая клада». Мы использовали хлопчатобумажную крысу Sigmodon hispidus , чтобы представить кладу, несущую активный L1, но неактивный B1, далее именуемую «базальной группой». Мы использовали мышь-олень Peromyscus maniculatus для представления близкородственной клады, несущей как активные L1, так и B1, далее именуемой «внешней группой».
Используя несобранные последовательности геномов видов, представляющих вымершую кладу L1 и базальную группу, мы показываем, что активность семейств L1 и B1, предшествовавшая расхождению клад, сравнима в современных геномах двух групп. Семейства L1 неуклонно заменялись до разделения на две группы и сохраняли активность после разделения базовой группы и вымершей клады L1. Вскоре после этого разделения активность L1 прекратилась в вымершей кладе L1, но стала очень активной в основной группе. У B1, с другой стороны, было очень большое увеличение активности до разделения между вымершей кладой L1 и базовой группой, и нет никаких свидетельств активности в двух группах после их расхождения.
Вскоре после этого разделения активность L1 прекратилась в вымершей кладе L1, но стала очень активной в основной группе. У B1, с другой стороны, было очень большое увеличение активности до разделения между вымершей кладой L1 и базовой группой, и нет никаких свидетельств активности в двух группах после их расхождения.
Результаты
Чтобы исследовать историю ретротранспозиции L1 у O. palustris и S. hispidus , мы использовали COSEG [63] для идентификации близкородственных групп L1 на основе общих, косегрегирующих сайтов, как описано в Методах. Мы следуем соглашению COSEG по обозначению этих групп как подсемейств . RepeatMasker [63] использовали для отнесения геномных копий L1 к подсемействам, генерируемым COSEG, и семь подсемейств без назначенных последовательностей были исключены из дальнейшего рассмотрения, оставив 47 подсемейств для дальнейшего анализа.
Чтобы изучить активность L1 в O. palustris и S. hispidus , мы провели поиск в файлах трассировок обоих геномов отдельно с консенсусными последовательностями вышеупомянутых 47 подсемейств и идентифицировали 19 254 последовательности в O. palustris и 90 526 в S. hispidus . Относительный возраст каждой последовательности был аппроксимирован ее процентным отклонением от соответствующего консенсуса подсемейства — чем выше процентное отклонение, тем старше последовательность. Пик распределения использовался как приближение расхождения подсемейства (дополнительный файл 3, таблица S1). Учитывая возможные изменения скорости эволюции в обнаруживаемом диапазоне истории эволюции L1, глобальное преобразование процентного расхождения во время является сложной задачей. Однако из-за общей эволюционной истории O. palustris и S. hispidus , мы можем использовать процентную дивергенцию в качестве достаточно хорошего маркера для сравнения относительного возраста подсемейств L1 двух видов, как это типично в исследованиях подвижных элементов [63].
palustris и 90 526 в S. hispidus . Относительный возраст каждой последовательности был аппроксимирован ее процентным отклонением от соответствующего консенсуса подсемейства — чем выше процентное отклонение, тем старше последовательность. Пик распределения использовался как приближение расхождения подсемейства (дополнительный файл 3, таблица S1). Учитывая возможные изменения скорости эволюции в обнаруживаемом диапазоне истории эволюции L1, глобальное преобразование процентного расхождения во время является сложной задачей. Однако из-за общей эволюционной истории O. palustris и S. hispidus , мы можем использовать процентную дивергенцию в качестве достаточно хорошего маркера для сравнения относительного возраста подсемейств L1 двух видов, как это типично в исследованиях подвижных элементов [63].
Консенсусные последовательности подсемейства также использовались для вывода о филогенетических отношениях между подсемействами (дополнительный файл 1: рисунок S1).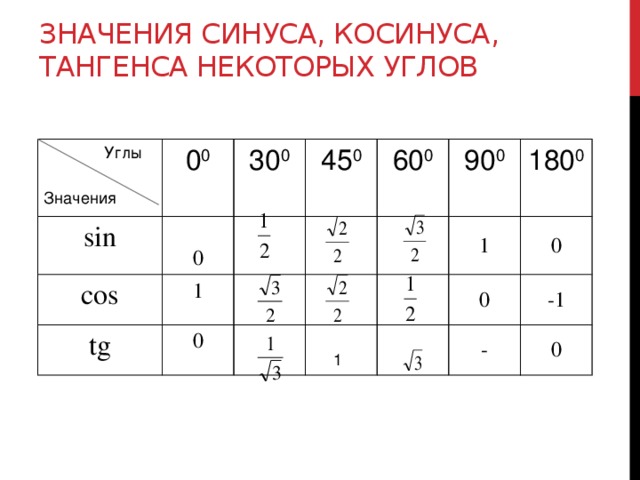 В дальнейшем филогенетические отношения и сходство последовательностей между подсемействами использовали для отнесения подсемейств к семействам с условием, что попарное расстояние между подсемействами внутри семейства не должно превышать 3,5%. Это расстояние было определено оперативно на основе расхождений между филогенетически сгруппированными подсемействами. Кластеры подсемейств, сходных на уровне последовательностей, но различающихся по дивергенции, были отнесены к разным семействам. Этот процесс выявил пять семей, характерных для S. hispidus (от L1-S1 до L1-S5), четыре семейства, общие для O. palustris и S. hispidus (от L1-OS1 до L1-OS4), и два общих для P. maniculatus , O. palustris и S. hispidus (L1-OSP1 и L1-OSP2, дополнительный файл 3: таблица S1). Филогения на основе расстояния, отражающая родство между семьями L1, представлена на рис. 2а. Отдельные последовательности были отнесены к семействам, к которым принадлежат их подсемейства; расхождение внутри семейства основано на расстоянии каждой последовательности от ее консенсуса подсемейства (рис.
В дальнейшем филогенетические отношения и сходство последовательностей между подсемействами использовали для отнесения подсемейств к семействам с условием, что попарное расстояние между подсемействами внутри семейства не должно превышать 3,5%. Это расстояние было определено оперативно на основе расхождений между филогенетически сгруппированными подсемействами. Кластеры подсемейств, сходных на уровне последовательностей, но различающихся по дивергенции, были отнесены к разным семействам. Этот процесс выявил пять семей, характерных для S. hispidus (от L1-S1 до L1-S5), четыре семейства, общие для O. palustris и S. hispidus (от L1-OS1 до L1-OS4), и два общих для P. maniculatus , O. palustris и S. hispidus (L1-OSP1 и L1-OSP2, дополнительный файл 3: таблица S1). Филогения на основе расстояния, отражающая родство между семьями L1, представлена на рис. 2а. Отдельные последовательности были отнесены к семействам, к которым принадлежат их подсемейства; расхождение внутри семейства основано на расстоянии каждой последовательности от ее консенсуса подсемейства (рис. 3).
3).
Филогении семейств L1 и B1. Филогенез показывает видовую специфичность семейств L1 и B1, и что существуют семейства L1, специфичные для S. hispidus , но не семейства B1. Панель a показывает дерево L1, а b показывает дерево B1. Чтобы отразить расхождение семейств, деревья были основаны на расстояниях между ними. Расстояние между любыми двумя семействами рассчитывали, взяв среднее попарное расстояние консенсусных последовательностей подсемейств, принадлежащих каждому семейству. Нумерация семейств L1 и B1 осуществляется в хронологическом порядке каждого элемента — семейства L1 и B1 с одинаковыми номерами не обязательно соответствуют одному и тому же периоду времени
Увеличенное изображение
Рис. 3 Дивергентное распределение L1-семейств. Семейства L1 были общими и имели одинаковые номера копий в O. palustris и S. hispidus вплоть до исчезновения L1. L1 испытал замедление (средняя левая панель) в S. hispidus , пока не восстановился (нижние панели — обратите внимание, что ось количества копий в 10 раз больше, чем на верхней и средней панелях). Семьи L1 в каждом ряду расположены в хронологическом порядке, самые молодые семьи слева. Виды, проанализированные в каждой строке, указаны справа. Имена семей отмечены в верхней части каждой панели. Число копий L1 отображается в виде процентного отклонения от соответствующего консенсуса подсемейства в 1% бинах. Расхождение каждого семейства аппроксимируется пиком распределения. Количество копий L1 нормализовано как количество копий на три Гб последовательности MiSeq, что приблизительно соответствует количеству копий на гаплоидный геном. Панель a показывает общие семейства, а панель b показывает семейства Sigmodon . Семейства L1 и B1 (на всех рисунках), которые соответствуют аналогичному расхождению, обозначены одинаковым цветом, и это цветовое кодирование одинаково на всех рисунках, показывающих расхождение L1 и B1: L1-OS2, красный; L1-OSP1, зеленый; и, L1-OSP2, синий
L1 испытал замедление (средняя левая панель) в S. hispidus , пока не восстановился (нижние панели — обратите внимание, что ось количества копий в 10 раз больше, чем на верхней и средней панелях). Семьи L1 в каждом ряду расположены в хронологическом порядке, самые молодые семьи слева. Виды, проанализированные в каждой строке, указаны справа. Имена семей отмечены в верхней части каждой панели. Число копий L1 отображается в виде процентного отклонения от соответствующего консенсуса подсемейства в 1% бинах. Расхождение каждого семейства аппроксимируется пиком распределения. Количество копий L1 нормализовано как количество копий на три Гб последовательности MiSeq, что приблизительно соответствует количеству копий на гаплоидный геном. Панель a показывает общие семейства, а панель b показывает семейства Sigmodon . Семейства L1 и B1 (на всех рисунках), которые соответствуют аналогичному расхождению, обозначены одинаковым цветом, и это цветовое кодирование одинаково на всех рисунках, показывающих расхождение L1 и B1: L1-OS2, красный; L1-OSP1, зеленый; и, L1-OSP2, синий
Полноразмерное изображение
Как и ожидалось, последовательности из семейств L1, общих для O. palustris и S. hispidus , присутствуют в обоих геномах, и эти общие семейства достаточно синхронизированы во времени и сопоставимы по числу копий (рис. 3а). L1-OS1 — это единственное совместно используемое семейство L1 между O. palustris и S. hispidus , которые показывают разницу: это самое молодое общее семейство L1, последнее активное семейство L1 перед вымиранием L1, и имеет примерно в 1,5 раза большее количество копий на Гб последовательности в O. palustris , чем у S. hispidus . Эта разница в отложении L1-OS1 между O. palustris и S. hispidus предполагает, что L1 оставались активными в вымершей кладе L1 после отделения этой группы от основной группы. Sigmodon -специфические семейства L1 (рис. 3b, семейства S1–5) претерпели значительную амплификацию после отделения от L1-вымершей клады, тогда как COSEG не идентифицировал Oryzomys -специфичных подсемейств. Подсемейства, специфичные для Sigmodon , имели несколько последовательностей из генома O.
palustris и S. hispidus , присутствуют в обоих геномах, и эти общие семейства достаточно синхронизированы во времени и сопоставимы по числу копий (рис. 3а). L1-OS1 — это единственное совместно используемое семейство L1 между O. palustris и S. hispidus , которые показывают разницу: это самое молодое общее семейство L1, последнее активное семейство L1 перед вымиранием L1, и имеет примерно в 1,5 раза большее количество копий на Гб последовательности в O. palustris , чем у S. hispidus . Эта разница в отложении L1-OS1 между O. palustris и S. hispidus предполагает, что L1 оставались активными в вымершей кладе L1 после отделения этой группы от основной группы. Sigmodon -специфические семейства L1 (рис. 3b, семейства S1–5) претерпели значительную амплификацию после отделения от L1-вымершей клады, тогда как COSEG не идентифицировал Oryzomys -специфичных подсемейств. Подсемейства, специфичные для Sigmodon , имели несколько последовательностей из генома O. palustris , назначенных им, но эти назначения кажутся аномальными, поскольку последовательности сильно расходятся с консенсусными последовательностями подсемейства (Дополнительный файл 3: Таблица S1). Таким образом, L1 испытал расширение (L1-OS1) в линии, ведущей к Oryzomyalia, непосредственно перед вымиранием L1, в то время как линия, ведущая к Sigmodontini, испытала отсроченное, но гораздо большее расширение L1.
palustris , назначенных им, но эти назначения кажутся аномальными, поскольку последовательности сильно расходятся с консенсусными последовательностями подсемейства (Дополнительный файл 3: Таблица S1). Таким образом, L1 испытал расширение (L1-OS1) в линии, ведущей к Oryzomyalia, непосредственно перед вымиранием L1, в то время как линия, ведущая к Sigmodontini, испытала отсроченное, но гораздо большее расширение L1.
Чтобы изучить динамику B1 у сигмодонтных грызунов, мы провели анализ B1 аналогично анализу L1. Из-за короткой длины и богатой CpG природы B1 нам потребовалось в два раза больше последовательностей для формирования подсемейства во втором цикле COSEG, как описано в разделе «Методы». Анализ выявил 30 подсемейств и пять семейств B1 у обоих видов (дополнительный файл 4: таблица S2). Филогения на основе расстояния, отражающая отношения между семействами B1, представлена на рис. 2б. Одно из семейств (B1-OS1) делится на O. palustris и S. hispidus и подсемейства внутри B1-OS1 образуют политомию по филогении, основанной на расстоянии (данные не показаны). Остальные четыре семейства B1 (B1-OSP1–4) являются общими для O. palustris , S. hispidus и P. maniculatus . Представленность этих семейств как в геномах O. palustris , так и в геномах S. hispidus достаточно синхронизирована во времени и сопоставима по количеству копий (рис. 4). Поскольку внешняя группа, представленная P. maniculatus , несет как активные L1, так и B1, мы знаем, что исчезновение B1 произошло после разделения внешней группы, однако необходимо определить точку, в которой B1 потерял активность в основной группе. Здесь мы показываем, что пик самого последнего семейства B1 находится при расхождении ~ 11,1% у O. palustris и ~ 10,7% у S. hispidus (дополнительный файл 4: таблица S2). Эти пики находятся в том же окне расхождения, что и L1-OS2 (~ 11,1% в O. palustris и ~ 10,3% в S. hispidus , Дополнительный файл 3: Таблица S1), предполагая, что B1-OS1 совпадает по времени с L1-OS2. Исключение сайтов CpG при расчете процентной дивергенции уменьшает вариацию между часами L1 и B1, но мы признаем, что другие незначительные вариации частоты мутаций между L1 и B1 все еще могут существовать.
Остальные четыре семейства B1 (B1-OSP1–4) являются общими для O. palustris , S. hispidus и P. maniculatus . Представленность этих семейств как в геномах O. palustris , так и в геномах S. hispidus достаточно синхронизирована во времени и сопоставима по количеству копий (рис. 4). Поскольку внешняя группа, представленная P. maniculatus , несет как активные L1, так и B1, мы знаем, что исчезновение B1 произошло после разделения внешней группы, однако необходимо определить точку, в которой B1 потерял активность в основной группе. Здесь мы показываем, что пик самого последнего семейства B1 находится при расхождении ~ 11,1% у O. palustris и ~ 10,7% у S. hispidus (дополнительный файл 4: таблица S2). Эти пики находятся в том же окне расхождения, что и L1-OS2 (~ 11,1% в O. palustris и ~ 10,3% в S. hispidus , Дополнительный файл 3: Таблица S1), предполагая, что B1-OS1 совпадает по времени с L1-OS2. Исключение сайтов CpG при расчете процентной дивергенции уменьшает вариацию между часами L1 и B1, но мы признаем, что другие незначительные вариации частоты мутаций между L1 и B1 все еще могут существовать. Поскольку L1-OS2 является самым молодым семейством L1 до разделения базальной группы и вымершей клады L1, последняя волна ретротранспозиции B1, вероятно, предшествовала исчезновению L1.
Поскольку L1-OS2 является самым молодым семейством L1 до разделения базальной группы и вымершей клады L1, последняя волна ретротранспозиции B1, вероятно, предшествовала исчезновению L1.
Дивергентное распределение семей B1. Семьи B1 в каждом ряду расположены в хронологическом порядке, самые молодые семьи слева. Виды, проанализированные в каждой строке, указаны справа. Имена семей отмечены в верхней части каждой панели. Количество копий B1 нанесено в виде процентного отклонения от соответствующего консенсуса подсемейства в 1% окнах. Расхождение каждого семейства аппроксимируется пиком распределения. Количество копий B1 нормализовано как количество копий на три Гб последовательности MiSeq, что приблизительно соответствует количеству копий на гаплоидный геном. Семейства L1 и B1 (на всех рисунках), которые соответствуют аналогичному расхождению, обозначены одинаковым цветом, и это цветовое кодирование одинаково на всех рисунках, показывающих расхождение L1 и B1: B1-OS1, красный; Б1-ОСП1, зеленый; и, B1-OSP3, синий
Изображение полного размера
Обсуждение
В этой статье мы исследуем темп активности L1 и B1, связанный с вымиранием обоих элементов, которое произошло у большинства видов подсемейства грызунов Sigmodontinae.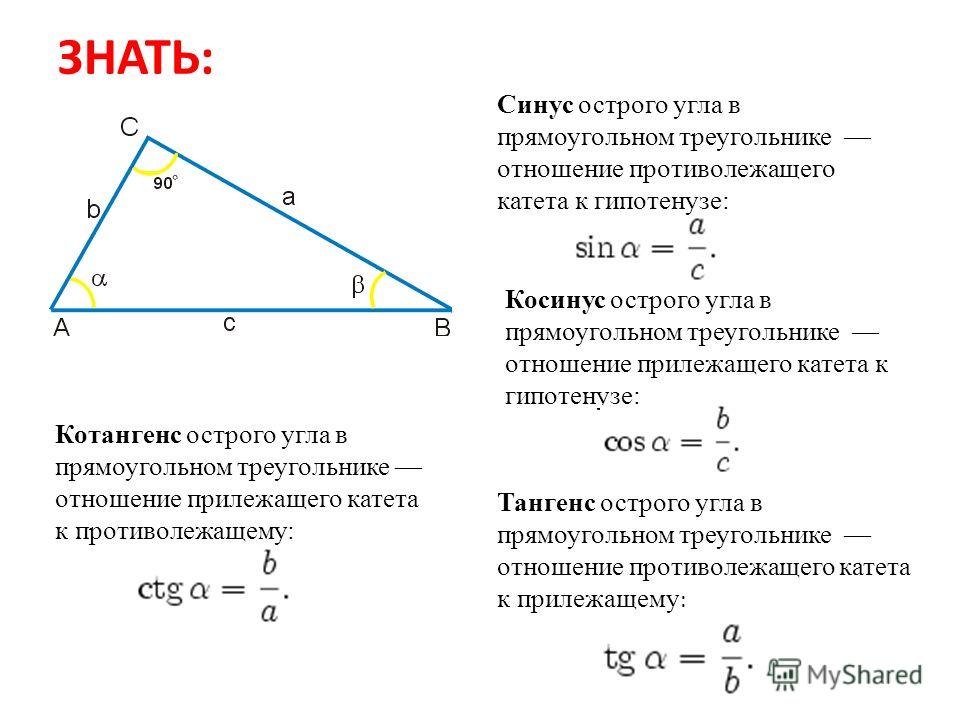 Эта работа стала возможной благодаря методам секвенирования, которые позволяют нам собирать большие объемы данных о последовательности, а также благодаря наличию надежной филогении видов для группы (рис. 1). Недавний филогенетический анализ мышевидных грызунов [57] показывает, что триба Sigmodontini является базальной в Sigmodontinae и сестрой трибе Ichthyomyini. Эти два племени являются сестрами большой политомической группы (оризомиалии), в которую входят остальные шесть племен. Подсемейство является результатом быстрой радиации грызунов в Южную Америку около 5 млн лет назад [64]. Предыдущая работа показала, что L1 вымерли у Oryzomyalia, но активны у Sigmodontini, состоящего из 14 видов одного рода, Сигмодон . Сводка по общему количеству L1 и B1 у S. hispidus и O. palustris (дополнительный файл 2: рисунок S2) согласуется с этой моделью исчезновения. Вымирание L1 в Oryzomyalia было зарегистрировано в 13 родах, распределенных по четырем трибам, охватывающим эту группу (рис.
Эта работа стала возможной благодаря методам секвенирования, которые позволяют нам собирать большие объемы данных о последовательности, а также благодаря наличию надежной филогении видов для группы (рис. 1). Недавний филогенетический анализ мышевидных грызунов [57] показывает, что триба Sigmodontini является базальной в Sigmodontinae и сестрой трибе Ichthyomyini. Эти два племени являются сестрами большой политомической группы (оризомиалии), в которую входят остальные шесть племен. Подсемейство является результатом быстрой радиации грызунов в Южную Америку около 5 млн лет назад [64]. Предыдущая работа показала, что L1 вымерли у Oryzomyalia, но активны у Sigmodontini, состоящего из 14 видов одного рода, Сигмодон . Сводка по общему количеству L1 и B1 у S. hispidus и O. palustris (дополнительный файл 2: рисунок S2) согласуется с этой моделью исчезновения. Вымирание L1 в Oryzomyalia было зарегистрировано в 13 родах, распределенных по четырем трибам, охватывающим эту группу (рис. 1). Доказательства этого вымирания L1 включали расхождение последовательностей между L1, клонированных с помощью метода, который обогащает недавно перемещенными элементами [65], и слабую гибридизацию наряду с отсутствием видо- или родоспецифичных полос в Саузерн-блоте 13 родов при зондировании с помощью Л1 [51].
1). Доказательства этого вымирания L1 включали расхождение последовательностей между L1, клонированных с помощью метода, который обогащает недавно перемещенными элементами [65], и слабую гибридизацию наряду с отсутствием видо- или родоспецифичных полос в Саузерн-блоте 13 родов при зондировании с помощью Л1 [51].
Мы реконструировали общую эволюционную историю L1 и B1 у Sigmodontinae в период до и после исчезновения этих элементов. Наши результаты показывают, что основные элементы L1 постоянно заменялись до исчезновения как L1, так и B1. Это отражено последовательным рядом семейств L1, общих для O. palustris и S. hispidus после их расхождения с Peromyscus . Элементы B1, по-видимому, не использовали каждую волну активности L1, но волна ретротранспозиции L1 (семейство L1-OS2, красный цвет на рисунках 3 и 5) соответствует пику ретротранспозиции B1 непосредственно перед исчезновением B1 (B1- OS1, красный цвет на рис. 4 и 5).
Рис.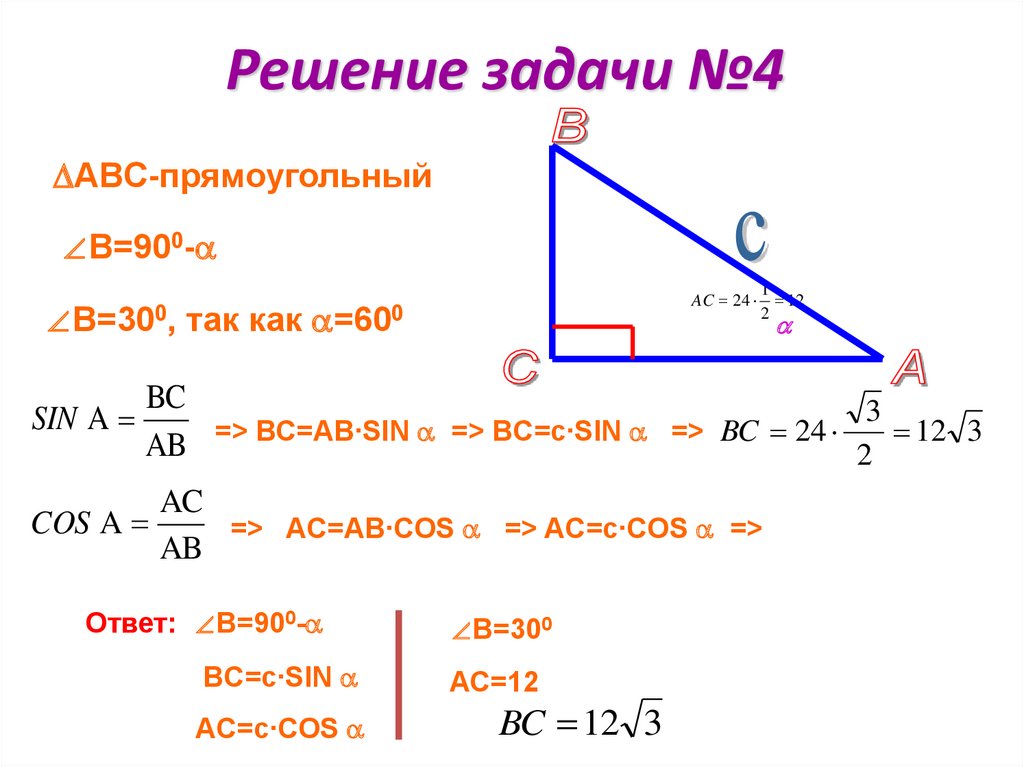 5
5 Сравнение числа расхождений и копий семейств L1 и B1. Кривые представляют распределение расхождений семейств L1 и B1 на основе сглаживания ядра гистограммы размера бина расхождения 1% семейства. Вертикальные пунктирные линии представляют пики распределений. Семействам L1 и B1 (на всех рисунках), которые соответствуют аналогичному расхождению, присвоен один и тот же цвет, и это цветовое кодирование одинаково на всех рисунках, показывающих расхождение L1 и B1: L1-OS2 и B1-OS1, красный; L1-OSP1 и B1-OSP1, зеленый; и L1-OSP2 и B1-OSP3, синие. Старшие два S. hispidus -специфические семейства L1 также показаны: L1-S5, черные; L1-S4, коричневый. Верхняя часть кривой L1-S5 (над горизонтальной черной пунктирной линией) усечена для наглядности, но ее пиковая координата показана вертикальной черной пунктирной линией и B1s из их консенсусов у видов, охватывающих 12 родов Sigmodontinae, Rinehart et al. [52] предположили, что вымирание L1 произошло после разделения между вымершей кладой L1 и Sigmodon , базальный род. Мы дополнительно уточняем этот временной интервал на сводной диаграмме, показывающей более высокий уровень активности L1-OS1 у O. palustris по сравнению с S. hispidus (рис. 5, пурпурный цвет). Это подтверждает вывод о том, что после разделения у обоих видов наблюдался некоторый уровень активности L1 и что события, приведшие к исчезновению L1, также произошли после разделения, а не через восстановление активности L1 у S. hispidus , как предполагалось ранее [1]. 51]. Эволюционная история B1 в O. palustris и S. hispidus сопоставимы. Новое отложение B1 в геноме было ничем не примечательным, за исключением большого всплеска у обоих видов в период, непосредственно предшествующий исчезновению B1 (B1-OS1, красный цвет на рис. 4 и 5). Учитывая короткую длину B1, идентифицировать кластеры подсемейств сложнее, поэтому наша оценка времени исчезновения B1 слабее, чем для L1. Однако две линии доказательств предполагают, что последний всплеск активности B1 произошел до разделения между L1-вымершими и базальными группами.
Мы дополнительно уточняем этот временной интервал на сводной диаграмме, показывающей более высокий уровень активности L1-OS1 у O. palustris по сравнению с S. hispidus (рис. 5, пурпурный цвет). Это подтверждает вывод о том, что после разделения у обоих видов наблюдался некоторый уровень активности L1 и что события, приведшие к исчезновению L1, также произошли после разделения, а не через восстановление активности L1 у S. hispidus , как предполагалось ранее [1]. 51]. Эволюционная история B1 в O. palustris и S. hispidus сопоставимы. Новое отложение B1 в геноме было ничем не примечательным, за исключением большого всплеска у обоих видов в период, непосредственно предшествующий исчезновению B1 (B1-OS1, красный цвет на рис. 4 и 5). Учитывая короткую длину B1, идентифицировать кластеры подсемейств сложнее, поэтому наша оценка времени исчезновения B1 слабее, чем для L1. Однако две линии доказательств предполагают, что последний всплеск активности B1 произошел до разделения между L1-вымершими и базальными группами. Во-первых, пиковая активность B1-OS1 наиболее точно соответствует пиковой активности L1-OS2, которая, по-видимому, предшествует расщеплению этих двух ветвей грызунов (рис. 5, красный цвет). Во-вторых, нет никаких указаний на различия в активности какого-либо из подсемейств B1 в O. palustris и S. hispidus (дополнительный файл 4: таблица S2), как и в случае с L1 (дополнительный файл 3: таблица S1).
Во-первых, пиковая активность B1-OS1 наиболее точно соответствует пиковой активности L1-OS2, которая, по-видимому, предшествует расщеплению этих двух ветвей грызунов (рис. 5, красный цвет). Во-вторых, нет никаких указаний на различия в активности какого-либо из подсемейств B1 в O. palustris и S. hispidus (дополнительный файл 4: таблица S2), как и в случае с L1 (дополнительный файл 3: таблица S1).
На оценку скорости ретротранспозиции, основанную на исторических количествах копий L1, может влиять скорость вырезания генома хозяина или предел обнаружения алгоритма. Хотя нет известного механизма, специально направленного на удаление L1 и B1, геномы млекопитающих постоянно удаляют последовательности с помощью различных механизмов, и скорость удаления варьирует в разных группах [66]. Более старые вставки подвергаются воздействию нецелевых механизмов вырезания в течение более длительного времени, поэтому ожидается, что в геноме будет представлено меньше копий старых семейств. Старые копии L1 и B1 также страдают от более ограниченного распознавания доступными алгоритмами. Последовательности, обнаруживаемые RepeatMasker, резко уменьшаются после 30% расхождения. Поскольку уровень мутаций в линии грызунов является одним из самых высоких среди всех млекопитающих, 30% расхождение в L1 и B1 восходит только к общему предку сигмодонтных грызунов и P. maniculatus , тогда как аналогичные исследования на летучих мышах [49] и приматах [67, 68] восходят к общему предку млекопитающих. К счастью, P. maniculatus несет как активные L1, так и B1 и находится достаточно близко, чтобы служить внешней группой в этом исследовании. Нам удалось идентифицировать два семейства L1, общих для O. palustris , S. hispidus и P. maniculatus , L1-OSP1 и L1-OSP2. Однако в этом типе эволюционного исследования есть преимущество изучения грызунов. Поскольку частота мутаций в линии грызунов выше, чем у других млекопитающих, эволюция подсемейств L1 и B1 за определенный период времени будет демонстрировать большую дивергенцию по сравнению с более медленно эволюционирующими видами.
Старые копии L1 и B1 также страдают от более ограниченного распознавания доступными алгоритмами. Последовательности, обнаруживаемые RepeatMasker, резко уменьшаются после 30% расхождения. Поскольку уровень мутаций в линии грызунов является одним из самых высоких среди всех млекопитающих, 30% расхождение в L1 и B1 восходит только к общему предку сигмодонтных грызунов и P. maniculatus , тогда как аналогичные исследования на летучих мышах [49] и приматах [67, 68] восходят к общему предку млекопитающих. К счастью, P. maniculatus несет как активные L1, так и B1 и находится достаточно близко, чтобы служить внешней группой в этом исследовании. Нам удалось идентифицировать два семейства L1, общих для O. palustris , S. hispidus и P. maniculatus , L1-OSP1 и L1-OSP2. Однако в этом типе эволюционного исследования есть преимущество изучения грызунов. Поскольку частота мутаций в линии грызунов выше, чем у других млекопитающих, эволюция подсемейств L1 и B1 за определенный период времени будет демонстрировать большую дивергенцию по сравнению с более медленно эволюционирующими видами. Это дает более высокое разрешение распределений дивергенции L1s и B1s и позволяет нам различать тонкие различия между дивергенцией подсемейств.
Это дает более высокое разрешение распределений дивергенции L1s и B1s и позволяет нам различать тонкие различия между дивергенцией подсемейств.
Выводы
Паттерны исторической активности L1 и B1 показывают критические временные рамки, при которых скорость ретротранспозиции расходилась у предковых хозяев. Очевидно, что L1 все еще были активны в то время, когда наследственные линии Oryzomyalia и Sigmodontinae разделились. Таким образом, вместо затишья L1 у общего предка с возрождением в линии sigmodontine, исчезновение L1, по-видимому, произошло после разделения и только в линии Oryzomyine. L1 вымерли у Oryzomyalia, но активны у Sigmodontini. B1 вымерли у Oryzomyalia и Sigmodontini. Однако статус как L1, так и B1 в промежуточной трибе Ichthyomyini неизвестен. Таким образом, вымирание L1 в результате этого единственного события, вероятно, затронет от 345 до 362 видов, или около 7% всех видов млекопитающих.
Наше исследование также показывает, что самая крупная и последняя волна экспансии B1 действительно произошла у общего предка линий-хозяев. Этот огромный всплеск активности B1 предполагает объяснение, отличное от покоя L1 и последующего дефицита белков L1, для его угасания. Учитывая, что мобильные элементы могут быть вовлечены в эволюционную гонку вооружений с факторами рестрикции хозяина [69], возможно, что радикальное расширение B1 вызвало более сильную защиту хозяина и в конечном итоге привело к исчезновению L1 и B1. Следовательно, реконструкция истории эволюции рестрикционных факторов хозяина L1 и B1 у соответствующих видов грызунов может стать ключом к раскрытию механизма вымирания L1 и B1.
Этот огромный всплеск активности B1 предполагает объяснение, отличное от покоя L1 и последующего дефицита белков L1, для его угасания. Учитывая, что мобильные элементы могут быть вовлечены в эволюционную гонку вооружений с факторами рестрикции хозяина [69], возможно, что радикальное расширение B1 вызвало более сильную защиту хозяина и в конечном итоге привело к исчезновению L1 и B1. Следовательно, реконструкция истории эволюции рестрикционных факторов хозяина L1 и B1 у соответствующих видов грызунов может стать ключом к раскрытию механизма вымирания L1 и B1.
Методы
ДНК O. palustris была получена из Исследовательской лаборатории естественных наук в Музее Техасского технического университета (идентификатор ткани: TK28621), а ДНК S. hispidus была получена из Техасской кооперативной коллекции дикой природы Техасского технического университета. Университет A&M (идентификатор ткани: MUR15). Секвенирование всего генома каждого вида проводили отдельными партиями с использованием MiSeq (Illumina, Inc. , Сан-Диего, Калифорния) в Центре геномных ресурсов IBEST (Университет Айдахо, Москва, штат Айдахо). Библиотеки с парными концами были созданы с размером вставки 450–550 bp; ~ 13 и 14 миллионов чтений было сгенерировано за O. palustris и S. hispidus соответственно. Последовательности обрабатывали с помощью SeqyClean (https://bitbucket.org/izhbannikov/seqyclean), а парные концы соединяли с помощью FLASH [70]. Охват генома был эквивалентен примерно 1,5X; 5,47 Гб последовательности были сгенерированы для O. palustris и 6,06 Гб для S. hispidus , но мы отмечаем, что размер генома у сигмодонтных грызунов варьируется. Хотя размер генома O. palustris , насколько нам известно, не задокументирован, размер генома сестринских видов в Oryzomys предполагают, что геномы Sigmodon на 11–16% больше, чем у Oryzomys [71].
, Сан-Диего, Калифорния) в Центре геномных ресурсов IBEST (Университет Айдахо, Москва, штат Айдахо). Библиотеки с парными концами были созданы с размером вставки 450–550 bp; ~ 13 и 14 миллионов чтений было сгенерировано за O. palustris и S. hispidus соответственно. Последовательности обрабатывали с помощью SeqyClean (https://bitbucket.org/izhbannikov/seqyclean), а парные концы соединяли с помощью FLASH [70]. Охват генома был эквивалентен примерно 1,5X; 5,47 Гб последовательности были сгенерированы для O. palustris и 6,06 Гб для S. hispidus , но мы отмечаем, что размер генома у сигмодонтных грызунов варьируется. Хотя размер генома O. palustris , насколько нам известно, не задокументирован, размер генома сестринских видов в Oryzomys предполагают, что геномы Sigmodon на 11–16% больше, чем у Oryzomys [71].
Реконструкции L1 для обоих видов были созданы на основе частичных геномных последовательностей, полученных с помощью 454 Pyrosequencing (Roche Applied Science, Penzberg, Germany) в Центре геномных ресурсов IBEST, 203 Мб последовательности для O. palustris и 214 Мб для S , гиспидус . Файлы трассировки генома P. maniculatus были получены с FTP-сайта NCBI (ftp://ftp.ncbi.nlm.nih.gov/pub/TraceDB/peromyscus_maniculatus/). Реконструкция 3′-концов O. palustris и S. hispidus L1 начинались с консенсусного семени размером 575 п.н. в 3′-половине ORF2 L1, полученного в соответствии с Cantrell et al. [65]. Биоинформатический конвейер для реконструкции полной длины L1 описан Yang et al. [49]. Вкратце, последовательности были получены из файлов трассировки генома на основе процентной идентичности [72, 73]. Выступы найденных последовательностей позволяли создавать новые начальные числа на обоих концах фрагмента L1 и использовались для инициации другого раунда запроса. В этом случае обход реконструкции повторялся в 3′-направлении до тех пор, пока не был достигнут 3′-конец ORF2. Процентное отсечение идентичности было установлено на уровне 92% для O. palustris для захвата самых последних активных последовательностей; для S.
palustris и 214 Мб для S , гиспидус . Файлы трассировки генома P. maniculatus были получены с FTP-сайта NCBI (ftp://ftp.ncbi.nlm.nih.gov/pub/TraceDB/peromyscus_maniculatus/). Реконструкция 3′-концов O. palustris и S. hispidus L1 начинались с консенсусного семени размером 575 п.н. в 3′-половине ORF2 L1, полученного в соответствии с Cantrell et al. [65]. Биоинформатический конвейер для реконструкции полной длины L1 описан Yang et al. [49]. Вкратце, последовательности были получены из файлов трассировки генома на основе процентной идентичности [72, 73]. Выступы найденных последовательностей позволяли создавать новые начальные числа на обоих концах фрагмента L1 и использовались для инициации другого раунда запроса. В этом случае обход реконструкции повторялся в 3′-направлении до тех пор, пока не был достигнут 3′-конец ORF2. Процентное отсечение идентичности было установлено на уровне 92% для O. palustris для захвата самых последних активных последовательностей; для S. hispidus использовалась более высокая процентная идентичность (97–99%) и длина выступа не менее 100 п.н. Это обеспечило удовлетворительный консенсус и исключение более старых элементов L1 для каждого вида. Затем 3’300 bp реконструированных L1 использовали в качестве эталонных последовательностей для анализа COSEG, описанного ниже.
hispidus использовалась более высокая процентная идентичность (97–99%) и длина выступа не менее 100 п.н. Это обеспечило удовлетворительный консенсус и исключение более старых элементов L1 для каждого вида. Затем 3’300 bp реконструированных L1 использовали в качестве эталонных последовательностей для анализа COSEG, описанного ниже.
Последовательности B1 из Rinehart et al. [52] использовали в качестве исходных семян для анализа B1. ПЦР-амплифицированные B1 из O. palustris и S. hispidus были выровнены с помощью Lasergene MegAlign (DNASTAR, Madison, WI), и консенсусная последовательность (146 bp) использовалась в качестве эталонной последовательности для анализа COSEG.
Подсемейства L1 и B1 в O. palustris и S. hispidus были идентифицированы и охарактеризованы аналогичным образом, как описано ниже, и обобщены в дополнительном файле 3: таблица S1 и дополнительном файле 4: таблица S2. Выравнивание последовательностей подсемейств L1 и B1 доступно в дополнительном файле 5 и дополнительном файле 6.
Каждая из реконструированных последовательностей длиной 300 п.н. с 3′-конца O. palustris и S. hispidus L1 ORF2 использовалась в качестве начальных последовательностей запроса L1, а полноразмерные консенсусы B1 от каждого вида, основанные на Rinehart et al. др. [52], использовались в качестве начальных последовательностей запросов B1. Библиотеки геномной ДНК O. palustris и S. hispidus MiSeq (обработанные чтения без сборки) запрашивали с помощью RepeatMasker [63] с параметрами по умолчанию. Этот начальный шаг был проведен для идентификации любой последовательности, которая потенциально гомологична L1/B1, что экономит время вычислений следующих шагов, избегая необходимости запрашивать все считывания последовательности напрямую. Обращения из запросов RepeatMasker были отфильтрованы по > 90% покрытие последовательности запроса и впоследствии использовалось для первого запуска COSEG [63] (http://www.repeatmasker.org/COSEGDownload.html) для идентификации подсемейств на основе общих, ко-сегрегирующих вариантов последовательностей. Все прогоны COSEG проводились с параметрами по умолчанию, за исключением отмеченных. Параметры были установлены таким образом, что для образования подсемейства L1 требовалось не менее 250 последовательностей, а для формирования подсемейства B1 требовалось 1000 последовательностей. Чтобы идентифицировать более старые подсемейства, консенсусные последовательности подсемейств, идентифицированные при первом запуске COSEG, использовались в качестве запросов для повторного поиска Библиотеки O. palustris и S. hispidus MiSeq с использованием RepeatMasker. Идентифицированные последовательности из второго запуска RepeatMasker были отфильтрованы для охвата > 90% и извлечены. Последовательности O. palustris и S. hispidus объединяют, и для объединенных последовательностей проводят второй цикл COSEG. Во избежание возможного образования случайных подсемейств из-за малой длины В1 и высокой копийности обнаруженных последовательностей количество последовательностей, необходимых для формирования подсемейства, было увеличено с 1000 (для прежнего отдельного запуска) до 2000, тогда как это число для L1 остался неизменным на уровне 250.
Все прогоны COSEG проводились с параметрами по умолчанию, за исключением отмеченных. Параметры были установлены таким образом, что для образования подсемейства L1 требовалось не менее 250 последовательностей, а для формирования подсемейства B1 требовалось 1000 последовательностей. Чтобы идентифицировать более старые подсемейства, консенсусные последовательности подсемейств, идентифицированные при первом запуске COSEG, использовались в качестве запросов для повторного поиска Библиотеки O. palustris и S. hispidus MiSeq с использованием RepeatMasker. Идентифицированные последовательности из второго запуска RepeatMasker были отфильтрованы для охвата > 90% и извлечены. Последовательности O. palustris и S. hispidus объединяют, и для объединенных последовательностей проводят второй цикл COSEG. Во избежание возможного образования случайных подсемейств из-за малой длины В1 и высокой копийности обнаруженных последовательностей количество последовательностей, необходимых для формирования подсемейства, было увеличено с 1000 (для прежнего отдельного запуска) до 2000, тогда как это число для L1 остался неизменным на уровне 250. Консенсусные последовательности полученных подсемейств COSEG были обрезаны, чтобы исключить концы, которые не были общими для всех подсемейств, а сайты CpG были удалены и, таким образом, обработаны RepeatMasker как пробелы и не учитывались при расчете расхождения. Эти модифицированные консенсусные последовательности подсемейства использовали для окончательного запроса индивидуальных Библиотеки O. palustris и S. hispidus MiSeq с использованием RepeatMasker. Последовательности из этого третьего запуска были отнесены к подсемействам на основе процентного расхождения, и эта информация была сохранена для дальнейшего анализа.
Консенсусные последовательности полученных подсемейств COSEG были обрезаны, чтобы исключить концы, которые не были общими для всех подсемейств, а сайты CpG были удалены и, таким образом, обработаны RepeatMasker как пробелы и не учитывались при расчете расхождения. Эти модифицированные консенсусные последовательности подсемейства использовали для окончательного запроса индивидуальных Библиотеки O. palustris и S. hispidus MiSeq с использованием RepeatMasker. Последовательности из этого третьего запуска были отнесены к подсемействам на основе процентного расхождения, и эта информация была сохранена для дальнейшего анализа.
Файлы трассировки генома P. maniculatus были получены аналогичным образом с помощью одного цикла RepeatMasker и COSEG. Описанные выше последовательности O. palustris L1 и B1 использовали в качестве начальных начальных значений запроса для этого прогона. Выбрано подсемейств P. maniculatus использовали для разграничения дивергенции подсемейств, идентифицированных в геномах O. palustris и S. hispidus (рис. 3).
palustris и S. hispidus (рис. 3).
Консенсусные последовательности подсемейства, полученные в результате второго запуска COSEG библиотек O. palustris и S. hispidus , были объединены и выровнены с помощью MegAlign с использованием метода Clustal W для L1 или метода Clustal V для B1, и была рассчитана матрица расстояний на основе выравнивания. Из-за использования консенсусов подсемейства пробелы в выравнивании редки и, следовательно, сохраняются. Сайты CpG были вручную удалены из выравниваний. На основе выравниваний были построены деревья максимального правдоподобия с использованием PhyML [74] с моделью GTR + I + G и 100 повторами начальной загрузки (дополнительный файл 1: рисунок S1). Затем последовательности L1 и B1 были отнесены к семействам на основе топологии дерева и попарного расстояния внутри семейства не более 3,5% от их консенсусов подсемейства для L1 и 4,4% для B1. Учитывая, что хозяева L1 и B1 постоянно заменяются в процессе эволюции, идеальное обозначение больших семейств невозможно.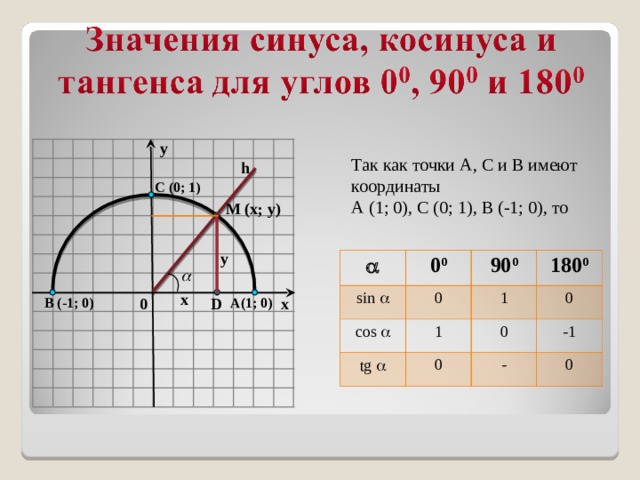 Порог в 3,5% был выбран таким образом, чтобы сгруппировать близкородственные подсемейства без увеличения количества семейств. Семейства названы в соответствии с их видовой специфичностью и дивергенцией: «S» означает Sigmodon , специфичные для семейства, «OS» для семейств, общих для Sigmodon и Oryzomys , и «OSP» для семейств, общих для Sigmodon , Oryzomys и Peromyscus ; цифры в фамилиях указывают на расхождение семьи в семейной группе, где «1» означает самый младший. Консенсусные последовательности семейства были созданы в программе MegAlign (DNAStar, Мэдисон, Висконсин) с использованием консенсусных последовательностей подсемейств, принадлежащих каждому семейству. Выравнивание и филогения семейств были созданы, как описано выше для подсемейств. Гистограммы распределений дивергенции L1 и B1 были сгенерированы функцией гистограммы R [75] с использованием размера ячейки 1% (рис. 3 и 4). Процентная дивергенция, соответствующая пикам ретротранспозиции отдельных семейств и подсемейств, определялась R с использованием функции сглаживания ядра с пропускной способностью 0,4% (дополнительный файл 3: таблица S1 и дополнительный файл 4: таблица S2).
Порог в 3,5% был выбран таким образом, чтобы сгруппировать близкородственные подсемейства без увеличения количества семейств. Семейства названы в соответствии с их видовой специфичностью и дивергенцией: «S» означает Sigmodon , специфичные для семейства, «OS» для семейств, общих для Sigmodon и Oryzomys , и «OSP» для семейств, общих для Sigmodon , Oryzomys и Peromyscus ; цифры в фамилиях указывают на расхождение семьи в семейной группе, где «1» означает самый младший. Консенсусные последовательности семейства были созданы в программе MegAlign (DNAStar, Мэдисон, Висконсин) с использованием консенсусных последовательностей подсемейств, принадлежащих каждому семейству. Выравнивание и филогения семейств были созданы, как описано выше для подсемейств. Гистограммы распределений дивергенции L1 и B1 были сгенерированы функцией гистограммы R [75] с использованием размера ячейки 1% (рис. 3 и 4). Процентная дивергенция, соответствующая пикам ретротранспозиции отдельных семейств и подсемейств, определялась R с использованием функции сглаживания ядра с пропускной способностью 0,4% (дополнительный файл 3: таблица S1 и дополнительный файл 4: таблица S2).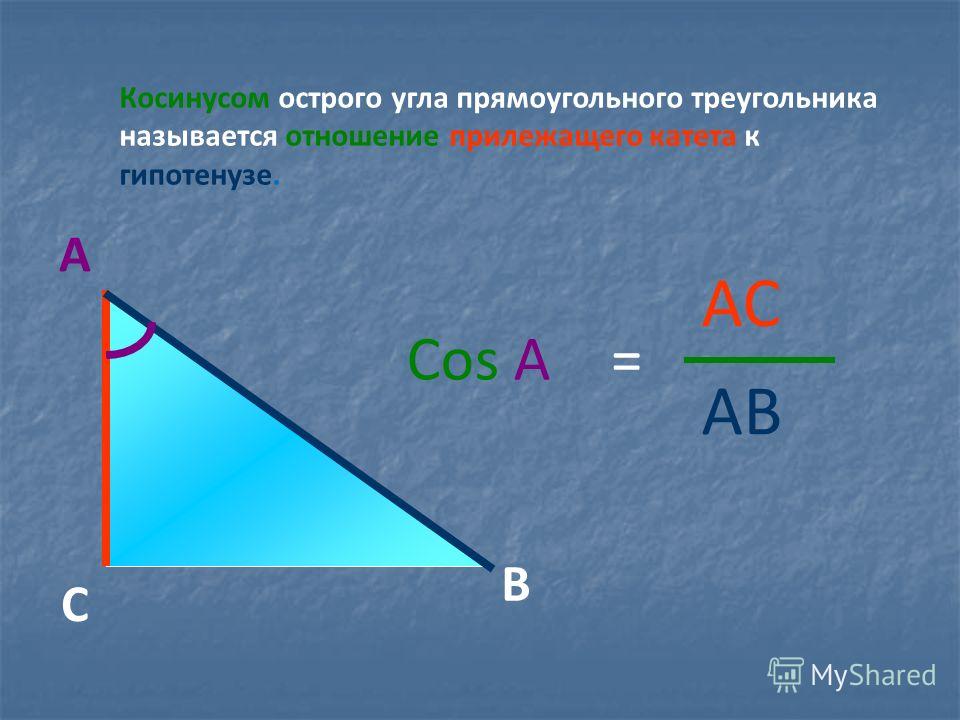
Чтобы избежать какой-либо систематической ошибки, вызванной использованием основанных на консенсусе семян, мы провели аналогичный анализ L1 в S. hispidus с использованием RepeatScout [76], который представляет собой метод идентификации повторов de novo, который не использует никаких априорных известные повторы. RepeatScout запускали с параметрами по умолчанию в обработанной библиотеке MiSeq S. hispidus для поиска повторяющихся последовательностей. Затем идентифицированные повторы были аннотированы с помощью RepeatMasker. Все L1-подобные последовательности, которые перекрываются с 3′ 300 п.н. S. hispidus L1 ORF2 использовали в качестве затравки для выполнения COSEG-анализа L1 в S. hispidus , следуя подходу, описанному выше. Все подсемейства L1 на основе RepeatScout (дополнительный файл 7) в S. hispidus находились в пределах 3,5% расхождения от подсемейства, определенного COSEG, как описано выше, и, следовательно, могут быть отнесены к одному и тому же семейству L1.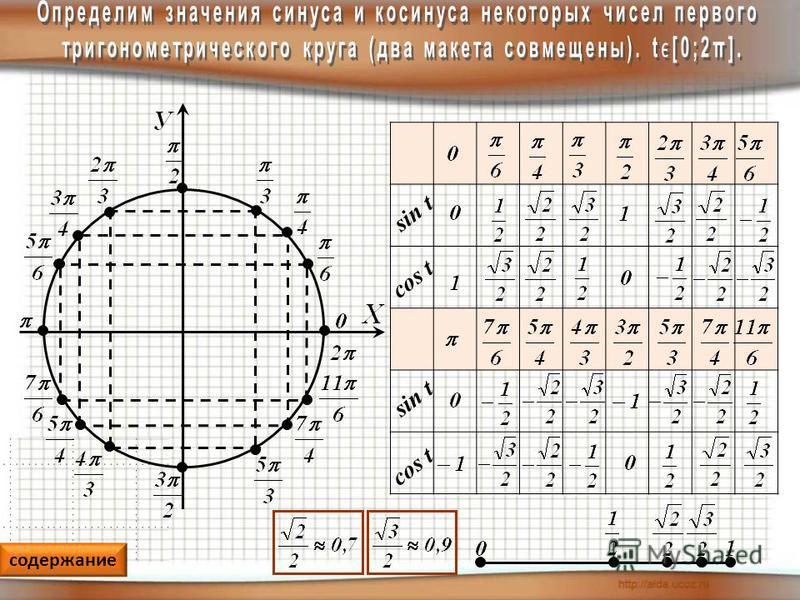
Сокращения
- ЛИНИЯ:
Длинный перемежающийся элемент
- млн лет назад:
Миллион лет назад
- О. болотная :
Оризомис болотный
- ORF:
Открытая рамка считывания
- P. maniculatus :
Peromyscus maniculatus
- S. hispidus :
Сигмодон гиспидус
- СИНУС:
Короткий вкрапленный элемент
Литература
- «>
Смит А.Ф. Происхождение вкрапленных повторов в геноме человека. Curr Opin Genet Dev. 1996;6(6):743–8.
КАС пабмед Статья Google ученый
Ландер Э.С., Линтон Л.М., Биррен Б., Нусбаум С., Зоди М.С., Болдуин Дж. и др. Начальная последовательность и анализ человеческого генома. Природа. 2001; 409 (6822): 860–921.
КАС пабмед Статья Google ученый
Секвенирование генома мыши C, Waterston RH, Lindblad-Toh K, Birney E, Rogers J, Abril JF, et al. Первоначальное секвенирование и сравнительный анализ генома мыши. Природа. 2002;420(6915): 520–62.

Артикул Google ученый
Boissinot S, Sookdeo A. Эволюция LINE-1 у позвоночных. Геном Биол Эвол. 2016;8(12):3485–507.
КАС пабмед ПабМед Центральный Google ученый
де Конинг А.П., Гу В., Кастоу Т.А., Батцер М.А., Поллок Д.Д. Повторяющиеся элементы могут составлять более двух третей генома человека. Генетика PLoS. 2011;7(12):e1002384.
ПабМед ПабМед Центральный Статья Google ученый
Platt RN 2nd, Vandewege MW, Ray DA. Мобильные элементы млекопитающих и их влияние на эволюцию генома. Хромосом Рез. 2018;26(1–2):25–43.
КАС Статья Google ученый
Фурано А.В. Биологические свойства и эволюционная динамика ретротранспозонов LINE-1 млекопитающих.
 Prog Nucleic Acid Res Mol Biol. 2000; 64: 255–9.4.
Prog Nucleic Acid Res Mol Biol. 2000; 64: 255–9.4.КАС пабмед Статья Google ученый
Вэй В., Гилберт Н., Оои С.Л., Лоулер Дж.Ф., Остертаг Э.М., Казазян Х.Х. и др. Ретротранспозиция L1 человека: цис-предпочтение по сравнению с транс-комплементацией. Мол Селл Биол. 2001;21(4):1429–39.
КАС пабмед ПабМед Центральный Статья Google ученый
Кулпа Д.А., Моран СП. Цис-предпочтительная активность обратной транскриптазы LINE-1 в частицах рибонуклеопротеина. Nat Struct Mol Biol. 2006;13(7):655–60.
КАС пабмед Статья Google ученый
Хан Дж.С., Боке Дж.Д. Высокоактивный синтетический ретротранспозон млекопитающих. Природа. 2004;429(6989):314–8.
КАС пабмед Статья Google ученый
«>Хан Дж.С., Сзак С.Т., Боке Дж.Д. Нарушение транскрипции ретротранспозоном L1 и последствия для транскриптомов млекопитающих. Природа. 2004; 429 (6989): 268–74.
КАС пабмед Статья Google ученый
Белансио В.П., Хеджес Д.Дж., Дейнингер П. Сплайсинг РНК LINE-1 и влияние на экспрессию генов млекопитающих. Нуклеиновые Кислоты Res. 2006;34(5):1512–21.
КАС пабмед ПабМед Центральный Статья Google ученый
Dewannieux M, Esnault C, Heidmann T. Опосредованная LINE ретротранспозиция маркированных последовательностей Alu.
 Нат Жене. 2003;35(1):41–8.
Нат Жене. 2003;35(1):41–8.КАС пабмед Статья Google ученый
Dewannieux M, Heidmann T. L1-опосредованная ретротранспозиция мышиных B1 и B2 SINE, рекапитулированных в культивируемых клетках. Дж Мол Биол. 2005;349(2): 241–7.
КАС пабмед Статья Google ученый
Уоллес Н., Вагстафф Б.Дж., Дайнингер П.Л., Рой-Энгель А.М. Белок LINE-1 ORF1 усиливает ретротранспозицию Alu SINE. Ген. 2008; 419(1–2):1–6.
КАС пабмед ПабМед Центральный Статья Google ученый
Васецкий Н.С., Крамеров Д.А. SINEBase: база данных и инструмент для анализа SINE. Нуклеиновые Кислоты Res. 2013; 41 (выпуск базы данных): D83–9.
КАС пабмед Статья Google ученый
«>Вайнер А.М. Обильная цитоплазматическая 7S РНК комплементарна доминирующему семейству вкрапленных средних повторяющихся последовательностей ДНК в геноме человека. Клетка. 1980;22(1 Пт 1):209–18.
КАС пабмед Статья Google ученый
Ullu E, Tschudi C. Последовательности Alu представляют собой процессированные гены РНК 7SL. Природа. 1984;312(5990):171–2.
КАС пабмед Статья Google ученый
Гейдушек Е.П., Кассаветис Г.А. Аппарат транскрипции РНК-полимеразы III.
 Дж Мол Биол. 2001;310(1):1–26.
Дж Мол Биол. 2001;310(1):1–26.КАС пабмед Статья Google ученый
Шрамм Л., Эрнандес Н. Рекрутирование РНК-полимеразы III на промоторы-мишени. Гены Дев. 2002;16(20):2593–620.
КАС пабмед Статья Google ученый
Птица АП. Метилирование ДНК и частота CpG в ДНК животных. Нуклеиновые Кислоты Res. 1980;8(7):1499–504.
КАС пабмед ПабМед Центральный Статья Google ученый
Ходжкинсон А., Эйр-Уокер А. Изменение частоты мутаций в геномах млекопитающих. Нат Рев Жене. 2011;12(11):756–66.
КАС пабмед Статья Google ученый
Хванг Д.Г., Грин П. Байесовская цепь Маркова Анализ последовательности Монте-Карло выявляет различные модели нейтрального замещения в эволюции млекопитающих.
 Proc Natl Acad Sci U S A. 2004;101(39):13994–4001.
Proc Natl Acad Sci U S A. 2004;101(39):13994–4001.КАС пабмед ПабМед Центральный Статья Google ученый
Магиоркинис Г., Белшоу Р., Кацуракис А. Туда и обратно»: пересмотр патофизиологических ролей эндогенных ретровирусов человека в постгеномную эпоху. Philos Trans R Soc Lond Ser B Biol Sci. 2013;368(1626):20120504.
Артикул Google ученый
Стое Ж.П. Эндогенные ретровирусы: все еще активны после стольких лет? Карр Биол. 2001; 11 (22): R914–6.
КАС пабмед Статья Google ученый
Luo ZX, Yuan CX, Meng QJ, Ji Q. Юрское плацентарное млекопитающее и дивергенция сумчатых и плацентарных. Природа. 2011;476(7361):442–5.
КАС пабмед Статья Google ученый
«>Касавант, Северная Каролина, Хардис, Южная Каролина. Динамика амплификации мышиного подсемейства LINE-1. Дж Мол Биол. 1994;241(3):390–7.
КАС пабмед Статья Google ученый
Adey NB, Schichman SA, Graham DK, Peterson SN, Edgell MH, Hutchison CA 3rd. Эволюция L1 грызунов управлялась единственной доминантной линией, которая неоднократно приобретала новые регуляторные последовательности транскрипции. Мол Биол Эвол. 1994; 11 (5): 778–89..
КАС пабмед Google ученый
Паскаль Э., Лю С., Валье Э., Усдин К.
 , Фурано А.В. Эволюция длинных вкраплений повторяющейся ДНК (L1, LINE 1), выявленная в результате анализа семейства ДНК L1 древних грызунов. Дж Мол Эвол. 1993;36(1):9–20.
, Фурано А.В. Эволюция длинных вкраплений повторяющейся ДНК (L1, LINE 1), выявленная в результате анализа семейства ДНК L1 древних грызунов. Дж Мол Эвол. 1993;36(1):9–20.КАС пабмед Статья Google ученый
Крамеров Д.А., Васецкий Н.С. Короткие ретропозоны в геномах эукариот. Int Rev Cytol. 2005; 247:165–221.
КАС пабмед Статья Google ученый
Огивара И., Мия М., Ошима К., Окада Н. Ретропозиционный паразитизм СИНУСОВ на ЛИНИЯХ: идентификация СИНУСОВ и ЛИНИЙ у пластиножаберных. Мол Биол Эвол. 1999;16(9):1238–50.
КАС пабмед Статья Google ученый
Goodier JL, Ostertag EM, Du K, Kazazian HH Jr. Новое подсемейство активных ретротранспозонов L1 у мышей. Геном Res. 2001;11(10):1677–85.
КАС пабмед ПабМед Центральный Статья Google ученый
«>Белансио В.П., Хеджес Д.Дж., Дейнингер П. Ретротранспозоны млекопитающих, не относящиеся к LTR: к лучшему или к худшему, в болезни и в здравии. Геном Res. 2008;18(3):343–58.
КАС пабмед Статья Google ученый
Муотри А.Р., Чу В.Т., Марчетто М.С., Дэн В., Моран Дж.В., Гейдж Ф.Х. Соматический мозаицизм в клетках-предшественниках нейронов, опосредованный ретротранспозицией L1. Природа. 2005; 435 (7044): 903–10.
КАС пабмед Статья Google ученый
Coufal NG, Garcia-Perez JL, Peng GE, Yeo GW, Mu Y, Lovci MT, et al.
 Ретротранспозиция L1 в нейральных клетках-предшественниках человека. Природа. 2009 г.;460(7259):1127–31.
Ретротранспозиция L1 в нейральных клетках-предшественниках человека. Природа. 2009 г.;460(7259):1127–31.КАС пабмед ПабМед Центральный Статья Google ученый
Кантрелл Массачусетс, Карстенс Британская Колумбия, Вичман ХА. Инактивация Х-хромосомы и эволюция Xist у грызуна, лишенного активности LINE-1. ПЛОС Один. 2009;4(7):e6252.
ПабМед ПабМед Центральный Статья Google ученый
Chow JC, Ciaudo C, Fazzari MJ, Mise N, Servant N, Glass JL и др. Активность LINE-1 в формировании факультативного гетерохроматина при инактивации Х-хромосомы. Клетка. 2010;141(6):956–69.
КАС пабмед Статья Google ученый
Сасаки Т., Нисихара Х., Хиракава М., Фуджимура К., Танака М., Кокубо Н. и др. Возможное участие SINE в формировании головного мозга у млекопитающих.
 Proc Natl Acad Sci U S A. 2008;105(11):4220–5.
Proc Natl Acad Sci U S A. 2008;105(11):4220–5.КАС пабмед ПабМед Центральный Статья Google ученый
Kunarso G, Chia NY, Jeyakani J, Hwang C, Lu X, Chan YS и другие. Мобильные элементы перепрограммировали основную регуляторную сеть эмбриональных стволовых клеток человека. Нат Жене. 2010;42(7):631–4.
КАС пабмед Статья Google ученый
Morrish TA, Gilbert N, Myers JS, Vincent BJ, Stamato TD, Taccioli GE, et al. Репарация ДНК, опосредованная эндонуклеазо-независимой ретротранспозицией LINE-1. Нат Жене. 2002;31(2):159–65.
КАС пабмед Статья Google ученый
Ариуми Ю. Хранитель генома человека: механизмы защиты хозяина от ретротранспозиции LINE-1. Фронт хим. 2016;4:28.
ПабМед ПабМед Центральный Статья Google ученый
«>Кантрелл М.А., Скотт Л., Браун С.Дж., Мартинес А.Р., Вичман Х.А. Потеря активности LINE-1 у мегабатов. Генетика. 2008;178(1):393–404.
КАС пабмед ПабМед Центральный Статья Google ученый
Ян Л., Брансфельд Дж., Скотт Л., Вичман Х. Оживление мертвых: история и реактивация вымершего L1. Генетика PLoS. 2014;10(6):e1004395.
ПабМед ПабМед Центральный Статья Google ученый
Casavant NC, Scott L, Cantrell MA, Wiggins LE, Baker RJ, Wichman HA. Конец ЛИНИИ?: отсутствие недавней активности L1 в группе южноамериканских грызунов.
 Генетика. 2000;154(4):1809–17.
Генетика. 2000;154(4):1809–17.КАС пабмед ПабМед Центральный Google ученый
Гран Р.А., Райнхарт Т.А., Кантрелл М.А., Вичман Х.А. Угасание активности LINE-1 совпало с основным облучением млекопитающих у грызунов. Цитогенет Геном Res. 2005; 110 (1–4): 407–15.
КАС пабмед Статья Google ученый
Райнхарт Т.А., Гран Р.А., Вичман Х.А. Вымирание SINE предшествовало вымиранию LINE у сигмодонтных грызунов: значение для ретротранспозиционной динамики и механизмов. Цитогенет Геном Res. 2005;110(1–4):416–25.
КАС пабмед Статья Google ученый
Платт Р.Н. 2-й, Рэй Д.А. Вымирание ретроэлемента без LTR у Spermophilus tridecemlineatus. Ген. 2012; 500(1):47–53.
КАС пабмед Статья Google ученый
«>Уотерс П.Д., Добиньи Г., Пардини А.Т., Робинсон Т.Дж. Распределение LINE-1 в Afrotheria и Xenarthra: последствия для понимания эволюции LINE-1 в геномах плацентарных. Хромосома. 2004;113(3):137–44.
КАС пабмед Статья Google ученый
Галлус С., Халлстром Б.М., Кумар В., Додт В.Г., Янке А., Шуманн Г.Г. и др. Эволюционные истории мобильных элементов в геноме самого крупного из ныне живущих сумчатых хищников, тасманийского дьявола. Мол Биол Эвол. 2015;32(5):1268–83.
КАС пабмед ПабМед Центральный Статья Google ученый
«>Смит М.Ф., Паттон Дж.Л. Филогенетические отношения и радиация сигмодонтиновых грызунов в Южной Америке: данные цитохрома b. Дж Мамм Эвол. 1999;6(2):89–128.
Артикул Google ученый
Уилсон, DE. Виды млекопитающих мира: таксономический и географический справочник. Балтимор: Издательство Университета Джона Хопкинса; 2005.
Google ученый
Кантрелл М.А., Эдерер М.М., Эриксон И.К., Свир В.Дж., Бейкер Р.Дж., Вичман Х.А. MysTR: семейство эндогенных ретровирусов у млекопитающих, которое недавно подверглось амплификации до беспрецедентного числа копий.
 Дж Вирол. 2005;79(23): 14698–707.
Дж Вирол. 2005;79(23): 14698–707.КАС пабмед ПабМед Центральный Статья Google ученый
Эриксон И.К., Кантрелл М.А., Скотт Л., Вичман Х.А. Модернизация генома: вымирание L1 следует за эндогенной ретровирусной экспансией в группе мышевидных грызунов. Дж Вирол. 2011;85(23):12315–23.
ПабМед ПабМед Центральный Статья Google ученый
Кордо Р., Батцер, Массачусетс. Влияние ретротранспозонов на эволюцию генома человека. Нат Рев Жене. 2009 г.;10(10):691–703.
КАС пабмед ПабМед Центральный Статья Google ученый
Смит А., Хабли Р. RepeatMasker Open-3.0; 1996-2010 гг.
Google ученый
Маршалл Л.Г., Батлер Р.Ф., Дрейк Р.Э., Кертис Г.
 Х., Тедфорд Р.Х. Калибровка большой американской развязки. Наука. 1979; 204 (4390): 272–9.
Х., Тедфорд Р.Х. Калибровка большой американской развязки. Наука. 1979; 204 (4390): 272–9.КАС пабмед Статья Google ученый
Кантрелл М.А., Гран Р.А., Скотт Л., Вичман Х.А. Выделение маркеров из недавно транспонированных ретротранспозонов LINE-1. Биотехнологии. 2000;29(6):1310–6.
КАС пабмед Статья Google ученый
Григорий ТР. Смещения вставки-удаления и эволюция размера генома. Ген. 2004; 324:15–34.
КАС пабмед Статья Google ученый
Смит А.Ф., Тот Г., Риггс А.Д., Юрка Дж. Предки, охватывающие все млекопитающие подсемейства повторяющихся последовательностей LINE-1. Дж Мол Биол. 1995;246(3):401–17.
КАС пабмед Статья Google ученый
«>Джейкобс Ф.М., Гринберг Д., Нгуен Н., Хеусслер М., Юинг А.Д., Кацман С. и др. Эволюционная гонка вооружений между генами цинковых пальцев KRAB ZNF91/93 и ретротранспозонами SVA/L1. Природа. 2014;516(7530):242–5.
КАС пабмед ПабМед Центральный Статья Google ученый
Магок Т., Зальцберг С.Л. FLASH: быстрая корректировка длины коротких ридов для улучшения сборки генома. Биоинформатика. 2011;27(21):2957–63.
КАС пабмед ПабМед Центральный Статья Google ученый
Григорий ТР. База данных размеров генома животных.
 2014. http://www.genomesize.com.
2014. http://www.genomesize.com.Google ученый
Альтшул С.Ф., Гиш В., Миллер В., Майерс Э.В., Липман Д.Дж. Базовый инструмент локального поиска выравнивания. Дж Мол Биол. 1990;215(3):403–10.
КАС Статья Google ученый
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, et al. BLAST+: архитектура и приложения. БМК Биоинф. 2009;10:421.
Артикул Google ученый
Guindon S, Dufayard JF, Lefort V, Anisimova M, Hordijk W, Gascuel O. Новые алгоритмы и методы для оценки филогении с максимальным правдоподобием: оценка производительности PhyML 3.0. Сист биол. 2010;59(3):307–21.
КАС Статья Google ученый
R Основная команда. R: язык и среда для статистических вычислений.
 Вена: R Foundation for Statistical Computing; 2013.
Вена: R Foundation for Statistical Computing; 2013.Прайс А.Л., Джонс Н.С., Певзнер П.А. Идентификация de novo повторяющихся семей в больших геномах. Биоинформатика. 2005; 21 (Приложение 1): i351–8.
КАС пабмед Статья Google ученый
Капитонов В.В., Юрка Ю. Универсальная классификация мобильных эукариотических элементов, реализованная в Repbase. Нат Рев Жене. 2008;9(5):411–2.
ПабМед Статья Google ученый
An W, Dai L, Niewiadomska AM, Yetil A, O’Donnell KA, Han JS, et al. Характеристика синтетического человеческого ретротранспозона LINE-1 ORFeus-Hs. ДНК мафии. 2011;2(1):2.
КАС пабмед ПабМед Центральный Статья Google ученый
Deininger PL, Tiedge H, Kim J, Brosius J. Эволюция, экспрессия и возможная функция мастер-гена для амплификации семейства перемежающихся повторяющихся ДНК у грызунов. Prog Nucleic Acid Res Mol Biol. 1996; 52: 67–88.
КАС пабмед Статья Google ученый
Клаф Дж.Э., Фостер Дж.А., Барнетт М., Вичман Х.А. Компьютерное моделирование эволюции мобильных элементов: случайный шаблон и строгие эталонные модели. Дж Мол Эвол. 1996;42(1):52–8.
КАС пабмед Статья Google ученый
Hedges DJ, Deininger PL. Приглашение нестабильности: мобильные элементы, двухцепочечные разрывы и поддержание целостности генома. Мутат рез. 2007; 616(1–2):46–59.
КАС пабмед Статья Google ученый
Gilbert C, Feschotte C. Горизонтальное приобретение мобильных элементов и вирусных последовательностей: закономерности и последствия. Curr Opin Genet Dev. 2018;49:15–24.
КАС пабмед ПабМед Центральный Статья Google ученый
Буассино С., Роос С., Фурано А.В. Различные скорости амплификации и эволюции ретротранспозона LINE-1 (L1) у обезьян Нового Света. Дж Мол Эвол. 2004;58(1):122–30.
КАС пабмед Статья Google ученый
Шенк Дж.Дж., Роу К.С., Степпан С.Дж. Экологическая возможность и необходимость диверсификации повторяющихся континентальных колоний мышевидными грызунами. Сист биол. 2013;62(6):837–64.
ПабМед Статья Google ученый
Хан Х., Смит А., Буассино С. Молекулярная эволюция и темпы амплификации ретротранспозонов LINE-1 человека с момента возникновения приматов. Геном Res. 2006;16(1):78–87.
КАС пабмед ПабМед Центральный Статья Google ученый
Загрузить ссылки
Благодарности
Мы благодарим д-ра Ежи Юрку из Института исследований генетической информации за обучение биоинформатике, Джона Брунсфельда и д-ра Селесту Браун за полезные идеи по проектированию трубопровода реконструкции L1 и д-ра. Wenfeng An, Celeste Brown и James Foster за полезные комментарии и обсуждения. Материалы для этого исследования были предоставлены Исследовательской лабораторией естественных наук в Музее Техасского технологического университета и Техасской кооперативной коллекцией дикой природы Техасского университета A&M. IBEST Genomics Resources Core помог с высокопроизводительным секвенированием, а IBEST Computer Resources Core предоставил ресурсы для биоинформатического анализа.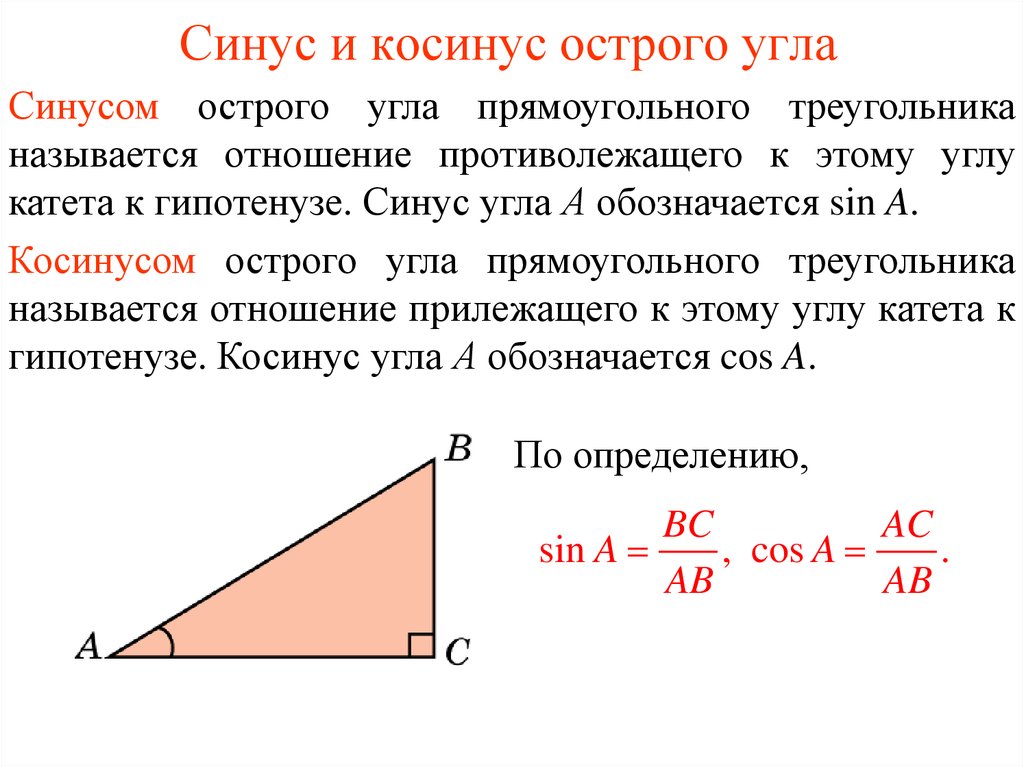
Финансирование
Эта работа финансировалась Национальным институтом здравоохранения R01-GM38737 для HAW и Национальным научным фондом DDIG-1210694 для HAW и LY. Аналитические ресурсы были предоставлены Национальным институтом здравоохранения P20GM103408 и P30GM103324. Поддержка для завершения анализа и подготовки рукописи была предоставлена P20GM102420. Спонсоры не участвовали в разработке исследования, сборе и анализе данных, принятии решения о публикации или подготовке рукописи.
Наличие данных и материалов
Данные, полученные или проанализированные в ходе этого исследования, включены в эту опубликованную статью и ее дополнительные файлы.
Информация об авторе
Авторы и организации
Факультет биологических наук, Университет Айдахо, Москва, штат Айдахо, США
Лей Янг, ЛуЭнн Скотт и Холли А. Вичман
Институт биоинформационных исследований и исследований Университет Айдахо, Москва, штат Айдахо, США
Лей Ян, ЛуЭнн Скотт и Холли А. Вичман
Вичман
Авторы
- Лей Ян
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- LuAnn Scott
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Holly A. Wichman
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
Вклады
LY и HAW задумали и разработали эксперимент, проанализировали данные и написали рукопись. LAS получила материалы для этого исследования, способствовала контролю качества данных и помогла с подготовкой рукописи. LY подготовила библиотеку ДНК для высокопроизводительного секвенирования и провела биоинформатический анализ. Все авторы прочитали и одобрили окончательный вариант рукописи.
Автор, ответственный за переписку
Холли А. Вичман.
Вичман.
Декларация этики
Одобрение этики и согласие на участие
Неприменимо.
Согласие на публикацию
Неприменимо.
Конкурирующие интересы
Авторы заявляют об отсутствии конкурирующих интересов.
Примечание издателя
Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и институциональной принадлежности.
Дополнительные файлы
Дополнительные файлы 1:
Рисунок S1. Филогения максимального правдоподобия обнаруженных подсемейств L1. Реконструированные O. palustris и S. hispidus L1, помеченные как «семена», и P. maniculatus подсемейств 5 и 6 включены в качестве маркеров. Дерево было построено с использованием PhyML [74] с моделью GTR + I + G и 100 бутстрепными повторами. Показаны значения начальной загрузки > 80%. (PDF, 39 КБ)
Дополнительный файл 2:
Рисунок S2. Распределение расхождений всех обнаруженных последовательностей L1 и B1. Процентное расхождение с соответствующими консенсусными последовательностями подсемейства представлено в ячейках 1%. Названия видов и ретротранспозонов указаны вверху каждой панели. (PDF 36 кб)
Процентное расхождение с соответствующими консенсусными последовательностями подсемейства представлено в ячейках 1%. Названия видов и ретротранспозонов указаны вверху каждой панели. (PDF 36 кб)
Дополнительный файл 3:
Таблица S1. Статистика и обозначение подсемейств и семейств L1. «Ory» означает O. palustris , а «Sig» означает S. hispidus . «Пик» указывает пик распределения дивергенции L1 подсемейства или семейства, идентифицированного ядерным сглаживанием. Количество копий нормировано как количество копий на три Гб последовательности MiSeq, используемой для поиска, что приблизительно соответствует количеству копий на гаплоидный геном. Обозначение семейств показано только после первого принадлежащего ему подсемейства; все последующие подсемейства принадлежат этому семейству до выделения следующего семейства. Символы в фамилиях: «S» представляет S. hispidus — специфичный, «OS» для общих с O. palustris и S. hispidus и «OSP» для общих с O. palustris , S. hispidus и P. maniculatus 90. Цифры в фамилиях отражают их расхождение в семейной группе, где «1» является самым младшим. Числа копий семейств представляют собой округленные суммы чисел копий подсемейств на три Гб последовательностей и, таким образом, иногда отклоняются на единицу. (XLSX 14 кб)
palustris , S. hispidus и P. maniculatus 90. Цифры в фамилиях отражают их расхождение в семейной группе, где «1» является самым младшим. Числа копий семейств представляют собой округленные суммы чисел копий подсемейств на три Гб последовательностей и, таким образом, иногда отклоняются на единицу. (XLSX 14 кб)
Доп.файл 4:
Таблица S2. Статистика и обозначение подсемейств и семейств В1. «Ory» означает O. palustris , а «Sig» означает S. hispidus . «Пик» указывает пик распределения расхождения B1 подсемейства или семейства, идентифицированного сглаживанием ядра. Количество копий нормировано как количество копий последовательности MiSeq, используемой для поиска, на три Гбит/с. Обозначение семейств показано только после первого принадлежащего ему подсемейства; все последующие подсемейства принадлежат этому семейству до выделения следующего семейства. Символы в фамилиях: «ОС» представляет семьи, разделенные O. palustris и S. hispidus и «OSP» для семейств, разделяемых O. palustris , S. hispidus и P. maniculatus . Цифры в фамилиях отражают их расхождение внутри семейной группы, где «1» является самым младшим. Число копий семейств представляет собой округленную сумму числа копий подсемейства на три Гб последовательностей и генома S. hispidus . Сайты CpG с признаками мутаций, вызванных метилированием, были удалены из выравнивания. (XLSX 13 КБ)
hispidus и «OSP» для семейств, разделяемых O. palustris , S. hispidus и P. maniculatus . Цифры в фамилиях отражают их расхождение внутри семейной группы, где «1» является самым младшим. Число копий семейств представляет собой округленную сумму числа копий подсемейства на три Гб последовательностей и генома S. hispidus . Сайты CpG с признаками мутаций, вызванных метилированием, были удалены из выравнивания. (XLSX 13 КБ)
Дополнительный файл 5:
Выравнивание последовательностей подсемейства L1, используемых в таблицах и рисунках. Сайты CpG с признаками мутаций, вызванных метилированием, были удалены из выравнивания. (FA 15 kb)
Дополнительный файл 6:
Выравнивание последовательностей подсемейства B1, используемых в таблицах и рисунках. Сайты CpG с признаками мутаций, вызванных метилированием, были удалены из выравнивания. (FA 7 kb)
Дополнительный файл 7:
Выравнивание последовательностей подсемейства L1 на основе сгенерированных RepeatScout семян и Геном S.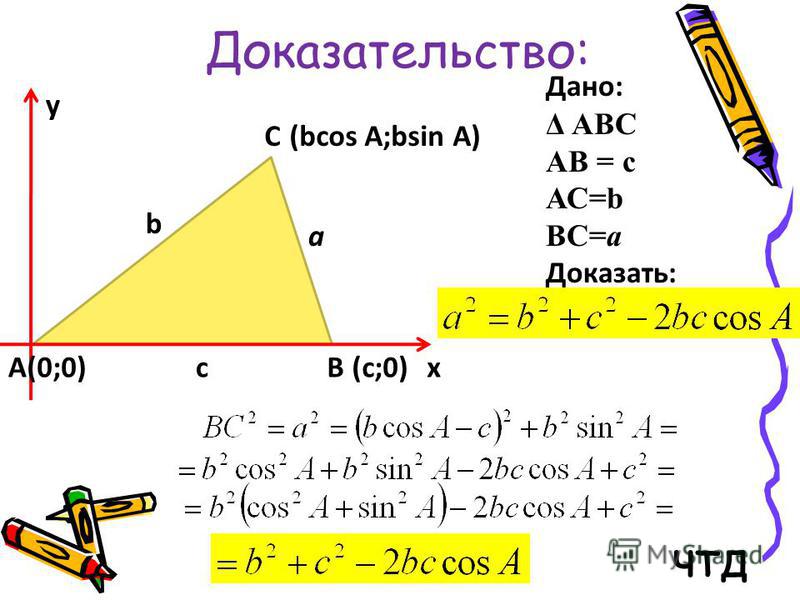 hispidus . Сайты CpG с признаками мутаций, вызванных метилированием, были удалены из выравнивания. (FA 21 kb)
hispidus . Сайты CpG с признаками мутаций, вызванных метилированием, были удалены из выравнивания. (FA 21 kb)
Права и разрешения
Открытый доступ Эта статья распространяется на условиях международной лицензии Creative Commons Attribution 4.0 (http://creativecommons.org/licenses/by/4.0/), которая разрешает неограниченное использование, распространение и воспроизведение на любом носителе при условии, что вы укажете первоначальных авторов и источник, предоставите ссылку на лицензию Creative Commons и укажете, были ли внесены изменения. Отказ от права Creative Commons на общественное достояние (http://creativecommons.org/publicdomain/zero/1.0/) применяется к данным, представленным в этой статье, если не указано иное.
Перепечатки и разрешения
Об этой статье
Графики синуса и косинуса | Brilliant Math & Science Wiki
Пи Хан Го, Мэй Ли, Рагхав Вайдьянатан, а также
способствовал
Содержимое
- Графики синуса и косинуса
- Характеристики
- Связь между графиками синусов и косинусов
- Растяжка и движение
- Решение проблем
На графике функции синуса ось xxx представляет значения θ\thetaθ, а ось yyy представляет значения sinθ\sin \thetasinθ. Например, sin0=0,\sin 0=0,sin0=0, подразумевая, что точка (0,0)(0,0)(0,0) является точкой на синусоидальном графике. Если мы нанесем значения функции синуса для большого числа углов θ\thetaθ, мы увидим, что точки образуют кривую, называемую синусоида :
Например, sin0=0,\sin 0=0,sin0=0, подразумевая, что точка (0,0)(0,0)(0,0) является точкой на синусоидальном графике. Если мы нанесем значения функции синуса для большого числа углов θ\thetaθ, мы увидим, что точки образуют кривую, называемую синусоида :
Аналогичным образом, построение значений функции косинуса для большого количества углов образует кривую, называемую косинусной кривой :
Мы можем визуализировать взаимосвязь между этими графиками и определением косинуса и синуса из единичного круга следующим образом:
Анимация предоставлена commons.wikimedia.org
Сколько точек пересечения есть между графиками sinx\sin xsinx и cosx\cos xcosx в интервале [0,2π][0,2\pi][0,2π]?
Из графиков синуса и косинуса видно, что количество точек пересечения в заданном диапазоне равно 222.
□_\square□
Диаграммы синуса и косинуса имеют диапазон [−1,1] [-1,1][−1,1] и повторяют значения каждые 2π2\pi2π (называемые амплитудой и периодом). Однако графики различаются по другим параметрам, например, по интервалам возрастания и убывания. Ниже приведены свойства каждого графа:
Свойства синуса:
- yyy-intercept: 000
- xxx-intercept: nπ, n \pi,nπ, где nnn — целое число здесь на
- возрастающих на интервалах [(4n−1)π2,(4n+1)π2]\left[\frac{(4n-1)\pi}{2},\frac{(4n+1)\pi}{2 } \right][2(4n−1)π,2(4n+1)π]
- убывающих на интервалах [(4n+1)π2,(4n+3)π2]\left[\frac{(4n+1)\pi}{2},\frac{(4n+3)\pi}{2 } \right][2(4n+1)π,2(4n+3)π]
- Симметрия: функция симметрична относительно начала координат.
- Максимум sinθ\sin\thetasinθ достигается при θ=(4n+1)π2\theta=\frac{(4n+1)\pi}{2}θ=2(4n+1)π.
Свойства косинуса:
- yyy-intercept: 1
- xxx-intercept: (n+12)π, \left(n + \frac{1}{2} \right) \pi, (n+21)π, где nnn — целое число здесь на
- возрастающих на интервалах [(2n−1)π,2nπ]\влево[(2n-1)\pi,2n\pi\вправо][(2n−1)π,2nπ]
- убывающих на интервалах [2nπ,(2n+1)π]\left[2n\pi,(2n+1)\pi \right][2nπ,(2n+1)π]
- Симметрия: функция симметрична относительно оси yyy.

- Максимум cosθ\cos\thetacosθ достигается при θ=2nπ\theta=2n\piθ=2nπ.
Найдите все xxx-перехваты для f(x)=2cos3x+2f(x) = 2\cos 3x +2 f(x)=2cos3x+2 в интервале 0≤x≤2π0\leq x\leq 2\ пи 0≤x≤2π.
Когда кривая f(x)f(x) f(x) пересекает ось xxx, f(x)=0f(x) = 0 f(x)=0. Таким образом, 2cos3x+2=0⇒cos3x=−12\cos3x + 2 = 0 \стрелка вправо \cos3x = -1 2cos3x+2=0⇒cos3x=-1.
Так как 0≤x≤2π⇒0≤3x≤6π0 \leq x \leq 2\pi \Rightarrow 0\leq 3x \leq 6\pi 0≤x≤2π⇒0≤3x≤6π и функция косинуса имеет период 2π2\pi2π, имеем 3x=π, π+2π, π+4π⇒x=π3, π, 5π3,3x = \pi, ~\pi+2\pi, ~\pi+4\pi \Rightarrow x=\frac{\pi} {3}, ~\pi, ~\frac{5\pi}{3},3x=π, π+2π, π+4π⇒x=3π, π, 35π, которые являются тремя xxx-перехватами, которые мы ищем. □_\квадрат□
Каково количество точек пересечения между двумя кривыми f(x)=5cosx+7f(x) = 5\cos x + 7 f(x)=5cosx+7 и g(x)=−6sinx− 10g(x) = -6\sin x — 10 g(x)=−6sinx−10 в интервале 0≤x≤2π0 \leq x \leq 2\pi0≤x≤2π?
Поскольку и f(x)f(x) f(x), и g(x)g(x) g(x) являются растянутыми графами со сдвигом по вертикали, мы сначала пытаемся выяснить, существует ли общее пересечение этих двух кривых.
Поскольку −1≤cosx≤1⇒−5≤5cosx≤5⇒2≤5cosx+7≤12-1 \leq \cos x \leq 1 \Rightarrow -5 \leq 5\cos x \ leq 5 \Rightarrow 2 \leq 5\cos x + 7 \leq 12 −1≤cosx≤1⇒−5≤5cosx≤5⇒2≤5cosx+7≤12, диапазон f(x)f(x) f (x) равно [2,12][2,12][2,12].
Аналогично, поскольку −1≤−sinx≤1⇒−6≤−6sinx≤6⇒−16≤−6sinx−10≤−4-1 \leq -\sin x \leq 1 \Rightarrow — 6\leq -6\sin x \leq 6 \Rightarrow -16 \leq -6\sin x -10 \leq -4 −1≤−sinx≤1⇒−6≤−6sinx≤6⇒−16≤−6sinx− 10≤−4, диапазон g(x)g(x) g(x) равен [−16,−4][-16,-4][−16,−4].
Поскольку f(x)f(x)f(x) строго положительна, а g(x)g(x) g(x) строго отрицательна, между этими двумя кривыми нет точки пересечения. В частности, у этих кривых нет точки пересечения в интервале 0≤x≤2π. □ 0 \leq x \leq 2\pi. \ _\квадрат 0≤x≤2π. □
График синуса имеет ту же форму, что и график косинуса. Действительно, график синуса можно получить, сдвинув график косинуса на (4n+1)π2\frac{(4n+1)\pi}{2}2(4n+1)π единиц вдоль положительных xxx- ось (nnn — целое число).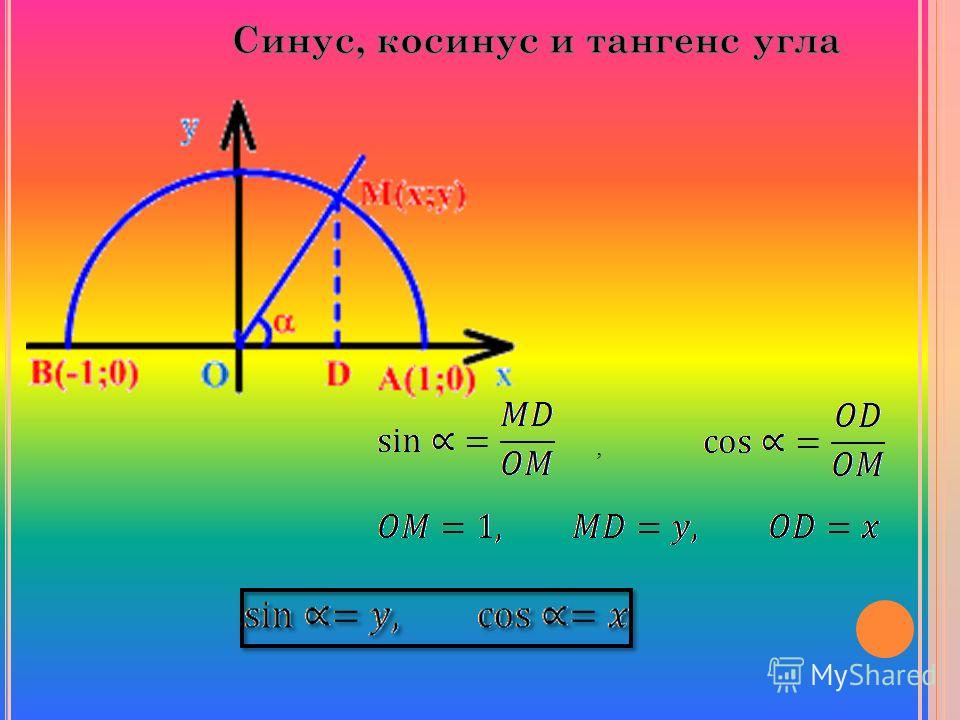 Также график косинуса можно получить, сдвинув график синуса на (4n+1)π2\frac{(4n+1)\pi}{2}2(4n+1)π единиц вдоль отрицательной xxx- ось. Другими словами:
Также график косинуса можно получить, сдвинув график синуса на (4n+1)π2\frac{(4n+1)\pi}{2}2(4n+1)π единиц вдоль отрицательной xxx- ось. Другими словами:
cosθ=sin((4n+1)π2−θ), sinθ=cos((4n+1)π2−θ).\cos{\theta}=\sin{ \left(\frac{(4n+1)\pi}{2}-\theta\right)}, ~\sin{\theta}=\cos{\left(\frac{(4n+1)\pi} {2}-\theta\right)}.cosθ=sin(2(4n+1)π−θ), sinθ=cos(2(4n+1)π−θ).
Упростить sin(9π2−θ) \sin \left( \frac{9 \pi }{ 2} — \theta \right) sin(29π−θ).
Поскольку функция sinθ \sin\theta sinθ имеет период 2π2\pi2π, sin(−θ)\sin(-\theta) sin(−θ) также имеет период 2π2\pi2π, поэтому
sin(9π2−θ)=sin(9π2−θ−2π−2π)=sin(π2−θ)=cosθ. \sin\left(\frac{9\pi}2 -\theta\right) = \sin\left(\frac{9\pi}2 -\theta — 2\pi — 2\pi \right) = \sin \left(\frac{\pi}2 -\theta\right) = \cos \theta. грех(29π−θ)=sin(29π−θ−2π−2π)=sin(2π−θ)=cosθ.
Альтернативно, мы распознаем это как форму sin((4n+1)π2−θ) \sin \left( \frac{(4n+1)\pi}{2} — \theta \right) sin(2 (4n+1)π−θ) при n=2 n = 2 n=2.
Следовательно, он равен cosθ \cos\theta cosθ. □_\квадрат□
Упростить cos(θ+π2) \cos \left ( \theta + \frac{\pi}{2} \right) cos(θ+2π).
Существует несколько подходов к использованию:
Используя перечисленные выше свойства, мы имеем
cos(θ+π2)=sin(−θ)=−sinθ. \cos\left (\theta + \frac{\pi}{2} \right) = \sin ( — \theta ) = — \sin \theta. потому что (θ+2π)=sin(-θ)=-sinθ.Нарисуйте график и сравните его с тем, что мы уже знаем.
Нарисовав график, мы можем визуально увидеть, что он равен −sinθ — \sin \theta −sinθ.Расширить, используя косинус — формулы суммы и разности, что дает нам
cos(θ+π2)=cosθcosπ2−sinθsinπ2=−sinθ. □ \cos \left ( \theta + \frac{\pi}{2} \right) = \cos \theta \cos \frac{\pi}{2} — \sin \theta \sin \frac{\pi} {2} = — \sin\theta.\ _\квадрат cos(θ+2π)=cosθcos2π−sinθsin2π=-sinθ. □
Полную информацию см.
в разделе Преобразование графика.
Мы можем манипулировать основным тригонометрическим графом, добавляя константы следующим образом:
y=asin(bx−c)+d.y = a \sin (b x — c) + d.y=asin(bx−c)+d. y=acos(bx−c)+d.y = a \cos(bx-c) + d.y=acos(bx−c)+d.
Рассмотрим, как каждая из этих констант изменяет график:
- Значение aaa растягивает график по вертикали.
- Значение bbb стягивает график по горизонтали
- Значение ccc переводит график по горизонтали
- Значение ddd переводит график вертикально.
Найдите амплитуду, период, сдвиг по горизонтали и сдвиг по вертикали функции
.у=2cos(π2x−π)+4. y = 2 \cos \left( \frac{\pi}{2} x — \pi \right) + 4.y=2cos(2πx−π)+4.
Итак, мы хотим найти значения a,b,ca,b,ca,b,c и ddd, сравнивая их с формой acos(bx−c)+da \cos(bx-c) + d acos(bx−c)+d.
Легко видеть, что a=2,b=π2,c=π,d=4a = 2, b = \frac\pi2, c =\pi, d = 4 a=2,b=2π,c= п,д=4.
Интерпретация значений показывает следующее:
- Период этой функции y=2cos(π2x−π)+4y = 2 \cos\left( \frac{\pi}{2} x — \pi \right) + 4 y=2cos(2πx −π)+4 равно 2π÷π2=4 2\pi \div \frac{\pi}2 = 4 2π÷2π=4.
- Амплитуда равна максимальному абсолютному значению скалярного кратного тригонометрической функции. В данном случае это ∣2cos(π2x−π)∣≤2 \left | 2 \cos \left( \frac{\pi}{2} x — \pi \right) \right | \leq 2 ∣∣2cos(2πx−π)∣∣≤2.
- Поскольку c=πc = \pic=π, функция была смещена по горизонтали на π\piπ единиц вправо.
- Аналогично, поскольку d=4d = 4d=4, функция была смещена по вертикали на 444 единицы вверх. □_\квадрат □
Учитывая функцию y=asin(bx−c)+dy = a \sin(bx-c) + dy=asin(bx−c)+d для констант a,b,c,a,b,c,a,b,c и ddd, каковы наибольшее и наименьшее возможные значения y?y?y?
Так как −1≤sinθ≤1 -1 \leq \sin \theta \leq 1 −1≤sinθ≤1, то −∣a∣≤asin(bx−c)≤∣a∣ -| а| \leq a \sin (bx -c) \leq |a| −∣a∣≤asin(bx−c)≤∣a∣.
Таким образом, наибольшее возможное значение равно ∣a∣+d |a| + d ∣a∣+d, а наименьшее возможное значение равно −∣a∣+d — |a| + d −∣a∣+d. □_\квадрат□
Положение пружины как функция времени представляется уравнением вида p(t)=acosbtp(t) = a \cosbtp(t)=acosbt. Если пружина начинается на 3 единицы выше точки покоя, отскакивает на 3 единицы ниже точки покоя, а затем возвращается на 3 единицы выше точки покоя всего за 2 секунды, найдите уравнение, описывающее это движение.
Из контекста «пружина начинается на 3 единицы выше точки покоя» мы можем интерпретировать это как p(0)=3p(0) = 3 p(0)=3, что подразумевает 3=acos(b× 0)⇒a=33 = a\cos(b\times0) \стрелка вправо a = 3 3=acos(b×0)⇒a=3.
Из контекста «а затем обратно на 3 единицы выше точки покоя всего за 2 секунды» мы можем интерпретировать это как фундаментальный период p(t)p(t) p(t) 22 2. Таким образом, 2π ÷b=2⇒b=π2 \pi \div b = 2 \Стрелка вправо b = \pi 2π÷b=2⇒b=π.
Следовательно, уравнение, описывающее это движение, имеет вид p(t)=3cosπt.
□p(t) = 3 \cos \pi t.\ _\square p(t)=3cosπt. □
Цитировать как: Графики синуса и косинуса. Brilliant.org . Извлекаются из https://brilliant.org/wiki/sine-and-cosine-graphs/
Влияние ретротранспозонов LINE-1 и SINE на геномы млекопитающих
1. Orgel LE, Crick FH, Sapienza C. Selfish DNA. Природа. 1980; 288: 645–646. [PubMed] [Google Scholar]
2. Doolittle WF, Sapienza C. Эгоистичные гены, парадигма фенотипа и эволюция генома. Природа. 1980; 284: 601–603. [PubMed] [Google Scholar]
3. Craig NL, Craigie R, Gellert M, Lambowitz AM. В: Мобильная ДНК II. Крейг Н.Л., Крейги Р., Геллерт М., Ламбовиц А.М., редакторы. Вашингтон, округ Колумбия: ASM Press; 2002. С. 1–1204. [Академия Google]
4. МакКлинток Б. Происхождение и поведение мутабельных локусов в кукурузе. Proc Natl Acad Sci U S A. 1950; 36:344–355. [Бесплатная статья PMC] [PubMed] [Google Scholar]
5. Fedoroff N, Wessler S, Shure M. Изоляция мобильных элементов управления кукурузой Ac и Ds. Клетка. 1983; 35: 235–242. [PubMed] [Google Scholar]
Fedoroff N, Wessler S, Shure M. Изоляция мобильных элементов управления кукурузой Ac и Ds. Клетка. 1983; 35: 235–242. [PubMed] [Google Scholar]
6. Boeke JD, Garfinkel DJ, Styles CA, Fink GR. Элементы Ty транспонируются через интермедиат РНК. Клетка. 1985; 40: 491–500. [PubMed] [Google Scholar]
7. Ландер Э.С., Линтон Л.М., Биррен Б., Нусбаум С., Зоди М.С., Болдуин Дж., Девон К., Дьюар К., Дойл М., ФитцХью В., Функе Р., Гейдж Д., Харрис К., Хифорд А., Хауленд Дж., Канн Л., Лехоцки Дж., ЛеВайн Р., Макьюэн П., МакКернан К., Мелдрим Дж., Месиров Дж.П., Миранда С., Моррис В., Нейлор Дж., Рэймонд С., Розетти М., Сантос Р., Шеридан А., Сугнез С. , Станге-Томанн Н., Стоянович Н., Субраманиан А., Вайман Д., Роджерс Дж., Салстон Дж., Эйнско Р., Бек С., Бентли Д., Бертон Дж., Кли С., Картер Н., Коулсон А., Дедман Р., Делоукас П., Данхэм А. , Данхэм И., Дурбин Р., Френч Л., Графхэм Д., Грегори С., Хаббард Т., Хамфри С., Хант А., Джонс М., Ллойд С., МакМюррей А., Мэтьюз Л., Мерсер С. , Милн С., Малликин Дж. К., Мангалл А., Пламб Р., Росс М., Шонкин Р., Симс С., Уотерстон Р.Х., Уилсон Р.К., Хиллиер Л.В., Макферсон Д.Д., Марра М.А., Мардис Э.Р., Фултон Л.А., Чинвалла А.Т., Пепин К.Х., Гиш В.Р., Чиссо С.Л., Вендл М.К., Делеханти К.Д., Майнер Т.Л., Делеханти А., Крамер Дж.Б., Кук Л.Л., Фултон Р.С., Джонсон Д.Л., Минкс П.Дж., Клифтон С.В., Хокинс Т., Бранско mb E, Predki P, Richardson P, Wenning S, Slezak T, Doggett N, Cheng JF, Olsen A, Lucas S, Elkin C, Uberbacher E, Frazier M, Gibbs RA, Muzny DM, Scherer SE, Bouck JB, Sodergren EJ , Уорли К.С., Ривз К.М., Горрелл Д.Х., Мецкер М.Л., Нейлор С.Л., Кучерлапати Р.С., Нельсон Д.Л., Вайнсток Г.М., Сакаки Ю., Фудзияма А., Хаттори М., Яда Т., Тойода А., Ито Т., Кавагоэ К., Ватанабэ Х., Тотоки Y, Taylor T, Weissenbach J, Heilig R, Saurin W, Artiguenave F, Brottier P, Bruls T, Pelletier E, Robert C, Wincker P, Smith DR, Doucette-Stamm L, Rubenfield M, Weinstock K, Lee HM, Dubois Дж., Розенталь А., Платцер М., Ньякатура Г., Таудиен С.
, Милн С., Малликин Дж. К., Мангалл А., Пламб Р., Росс М., Шонкин Р., Симс С., Уотерстон Р.Х., Уилсон Р.К., Хиллиер Л.В., Макферсон Д.Д., Марра М.А., Мардис Э.Р., Фултон Л.А., Чинвалла А.Т., Пепин К.Х., Гиш В.Р., Чиссо С.Л., Вендл М.К., Делеханти К.Д., Майнер Т.Л., Делеханти А., Крамер Дж.Б., Кук Л.Л., Фултон Р.С., Джонсон Д.Л., Минкс П.Дж., Клифтон С.В., Хокинс Т., Бранско mb E, Predki P, Richardson P, Wenning S, Slezak T, Doggett N, Cheng JF, Olsen A, Lucas S, Elkin C, Uberbacher E, Frazier M, Gibbs RA, Muzny DM, Scherer SE, Bouck JB, Sodergren EJ , Уорли К.С., Ривз К.М., Горрелл Д.Х., Мецкер М.Л., Нейлор С.Л., Кучерлапати Р.С., Нельсон Д.Л., Вайнсток Г.М., Сакаки Ю., Фудзияма А., Хаттори М., Яда Т., Тойода А., Ито Т., Кавагоэ К., Ватанабэ Х., Тотоки Y, Taylor T, Weissenbach J, Heilig R, Saurin W, Artiguenave F, Brottier P, Bruls T, Pelletier E, Robert C, Wincker P, Smith DR, Doucette-Stamm L, Rubenfield M, Weinstock K, Lee HM, Dubois Дж., Розенталь А., Платцер М., Ньякатура Г., Таудиен С. , Румп А., Ян Х., Ю Дж., Ван Дж., Хуанг Г., Гу Дж., Худ Л., Роуэн Л., Мадан А., Цинь С., Дэвис Р.В., Федершпиль Н.А., Абола А.П., Проктор М.Дж., Майерс Р.М., Шмутц Дж., Диксон М., Гримвуд Дж., Кокс Д.Р., Олсон М.В., Каул Р., Рэймонд С., Симидзу Н., Кавасаки К., Миношима С., Эванс Г.А., Атанасиу М., Шульц Р., Роу Б.А. , Чен Ф., Пан Х., Рамсер Дж., Лехрах Х., Рейнхардт Р., Маккомби В.Р., де ла Бастид М., Дедия Н., Блокер Х., Хорниш r K, Nordsiek G, Agarwala R, Aravind L, Bailey JA, Bateman A, Batzoglou S, Birney E, Bork P, Brown DG, Burge CB, Cerutti L, Chen HC, Church D, Clamp M, Copley RR, Doerks T , Эдди С.Р., Эйхлер Э.Е., Фьюри Т.С., Галаган Дж., Гилберт Дж.Г., Хармон С., Хаяшизаки Ю., Хаусслер Д., Хермякоб Х., Хокамп К., Ян В., Джонсон Л.С., Джонс Т.А., Касиф С., Каспризк А., Кеннеди С., Кент WJ, Kitts P, Koonin EV, Korf I, Kulp D, Lancet D, Lowe TM, McLysaght A, Mikkelsen T, Moran JV, Mulder N, Pollara VJ, Ponting CP, Schuler G, Schultz J, Slater G, Smit AF, Ступка Э., Шустаковски Дж.
, Румп А., Ян Х., Ю Дж., Ван Дж., Хуанг Г., Гу Дж., Худ Л., Роуэн Л., Мадан А., Цинь С., Дэвис Р.В., Федершпиль Н.А., Абола А.П., Проктор М.Дж., Майерс Р.М., Шмутц Дж., Диксон М., Гримвуд Дж., Кокс Д.Р., Олсон М.В., Каул Р., Рэймонд С., Симидзу Н., Кавасаки К., Миношима С., Эванс Г.А., Атанасиу М., Шульц Р., Роу Б.А. , Чен Ф., Пан Х., Рамсер Дж., Лехрах Х., Рейнхардт Р., Маккомби В.Р., де ла Бастид М., Дедия Н., Блокер Х., Хорниш r K, Nordsiek G, Agarwala R, Aravind L, Bailey JA, Bateman A, Batzoglou S, Birney E, Bork P, Brown DG, Burge CB, Cerutti L, Chen HC, Church D, Clamp M, Copley RR, Doerks T , Эдди С.Р., Эйхлер Э.Е., Фьюри Т.С., Галаган Дж., Гилберт Дж.Г., Хармон С., Хаяшизаки Ю., Хаусслер Д., Хермякоб Х., Хокамп К., Ян В., Джонсон Л.С., Джонс Т.А., Касиф С., Каспризк А., Кеннеди С., Кент WJ, Kitts P, Koonin EV, Korf I, Kulp D, Lancet D, Lowe TM, McLysaght A, Mikkelsen T, Moran JV, Mulder N, Pollara VJ, Ponting CP, Schuler G, Schultz J, Slater G, Smit AF, Ступка Э., Шустаковски Дж. , Тьерри-Миг Д., Тьерри-Миг Дж., Вагнер Л., Уоллис Дж., Уилер Р., Уильямс А., Вольф Й.И., Вулф К.Х., Ян С.П., Йе Р.Ф., Коллинз Ф., Гайер М.С., Петерсон Дж., Фельзенфельд А., Веттерстранд К.А., Патринос А., Морган М.Дж., де Йонг П., Катанесе Дж.Дж., Осоэгава К., Сидзуя Х., Чой С., Чен Ю.Дж. Начальная последовательность и анализ человеческого генома. Природа. 2001;409: 860–921. [PubMed] [Google Scholar]
, Тьерри-Миг Д., Тьерри-Миг Дж., Вагнер Л., Уоллис Дж., Уилер Р., Уильямс А., Вольф Й.И., Вулф К.Х., Ян С.П., Йе Р.Ф., Коллинз Ф., Гайер М.С., Петерсон Дж., Фельзенфельд А., Веттерстранд К.А., Патринос А., Морган М.Дж., де Йонг П., Катанесе Дж.Дж., Осоэгава К., Сидзуя Х., Чой С., Чен Ю.Дж. Начальная последовательность и анализ человеческого генома. Природа. 2001;409: 860–921. [PubMed] [Google Scholar]
8. Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, Smith HO, Yandell M, Evans CA, Holt RA, Gocayne JD, Amanatides P, Ballew RM, Хьюсон Д.Х., Вортман Дж.Р., Чжан К., Кодира К.Д., Чжэн Х.Х., Чен Л., Скупски М., Субраманиан Г., Томас П.Д., Чжан Дж., Габор Миклош Г.Л., Нельсон С., Бродер С., Кларк А.Г., Надо Дж., МакКьюсик В.А., Зиндер Н., Левин А.Дж., Робертс Р.Дж., Саймон М., Слейман С., Ханкапиллер М., Боланос Р., Делчер А., Дью И., Фасуло Д., Фланиган М., Флореа Л., Халперн А., Ханненхалли С., Кравиц С., Леви С., Мобарри С., Рейнерт К., Ремингтон К., Абу-Трейде Дж., Бизли Э. , Биддик К., Бонацци В., Брэндон Р., Каргилл М., Чандрамулисваран И., Чарлаб Р., Чатурведи К., Дэн З., Ди Франческо В., Данн П., Эйлбек К., Евангелиста С. , Габриэлян А.Э., Ган В., Гэ В., Гонг Ф., Гу З., Гуан П., Хейман Т.Дж., Хиггинс М.Е., Джи Р.Р., Ке З., Кетчум К.А., Лай З., Лей И., Ли З., Ли Дж., Лян И., Линь X, Лу Ф, Меркулов Г.В., Мильшина Н., Мур Х.М., Наик А.К., Нараян В.А., Нилам Б., Нускерн Д., Руш Д.Б., Зальцберг С., Шао В., Шу Б., Sun J, Wang Z, Wang A, Wang X, Wang J, Wei M, Wides R, Xiao C, Yan C, Yao A, Ye J, Zhan M, Zhang W, Zhang H, Zhao Q, Zheng L, Zhong F , Чжун В., Чжу С., Чжао С., Гилберт Д., Баумхутер С., Спир Дж., Картер С., Кравчик А., Вудейдж Т., Али Ф., Ан Х., Трепет А., Болдуин Д., Баден Х., Барнстед М., Бэрроу И., Бисон К., Бусам Д., Карвер А., Центр А., Ченг М.Л., Карри Л., Данахер С., Давенпорт Л., Десилетс Р., Дитц С., Додсон К., Доуп Л., Ферриера С., Гарг Н., Глюксманн А., Харт Б., Хейнс Дж., Хейнс С., Хайнер С., Хладун С., Хостин Д., Хоук Дж., Хауленд Т.
, Биддик К., Бонацци В., Брэндон Р., Каргилл М., Чандрамулисваран И., Чарлаб Р., Чатурведи К., Дэн З., Ди Франческо В., Данн П., Эйлбек К., Евангелиста С. , Габриэлян А.Э., Ган В., Гэ В., Гонг Ф., Гу З., Гуан П., Хейман Т.Дж., Хиггинс М.Е., Джи Р.Р., Ке З., Кетчум К.А., Лай З., Лей И., Ли З., Ли Дж., Лян И., Линь X, Лу Ф, Меркулов Г.В., Мильшина Н., Мур Х.М., Наик А.К., Нараян В.А., Нилам Б., Нускерн Д., Руш Д.Б., Зальцберг С., Шао В., Шу Б., Sun J, Wang Z, Wang A, Wang X, Wang J, Wei M, Wides R, Xiao C, Yan C, Yao A, Ye J, Zhan M, Zhang W, Zhang H, Zhao Q, Zheng L, Zhong F , Чжун В., Чжу С., Чжао С., Гилберт Д., Баумхутер С., Спир Дж., Картер С., Кравчик А., Вудейдж Т., Али Ф., Ан Х., Трепет А., Болдуин Д., Баден Х., Барнстед М., Бэрроу И., Бисон К., Бусам Д., Карвер А., Центр А., Ченг М.Л., Карри Л., Данахер С., Давенпорт Л., Десилетс Р., Дитц С., Додсон К., Доуп Л., Ферриера С., Гарг Н., Глюксманн А., Харт Б., Хейнс Дж., Хейнс С., Хайнер С., Хладун С., Хостин Д., Хоук Дж., Хауленд Т. , Ибегвам С., Джонсон Дж., Калуш Ф., Клайн Л., Кодуру С., Лав А., Манн Ф., Мэй Д., МакКоули С., Макинтош Т., Макмаллен И. , Мой М., Мой Л., Мерфи Б., Нельсон К., Пфаннкоч С., Праттс Э., Пури В., Куреши Х., Рирдон М., Родригес Р., Роджерс Ю.Х., Ромблад Д., Руфел Б., Скотт Р., Ситтер С., Смоллвуд М., Стюарт E, Strong R, Suh E, Thomas R, Tint NN, Tse S, Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J, Zaveri K, Abril Дж.Ф., Гиго Р., Кэмпбелл М.Дж., Шоландер К.В., Карлак Б., Кежаривал А., Ми Х., Лазарева Б., Хаттон Т., Наречания А., Димер К., Муругануджан А., Го Н., Сато С., Бафна В., Истраил С., Липперт Р., Шварц Р., Валенц Б., Юсеф С., Аллен Д. , Басу А., Баксендейл Дж., Блик Л., Каминья М., Карнес-Стайн Дж., Колк П., Чан Ю.Х., Койн М., Далке К., Мейс А., Домброски М., Доннелли М., Эли Д., Эспархам С., Фослер С., Гире Х. , Глановски С., Глассер К., Глодек А., Горохов М., Грэм К., Гропман Б., Харрис М., Хейл Дж., Хендерсон С., Гувер Дж., Дженнингс Д.
, Ибегвам С., Джонсон Дж., Калуш Ф., Клайн Л., Кодуру С., Лав А., Манн Ф., Мэй Д., МакКоули С., Макинтош Т., Макмаллен И. , Мой М., Мой Л., Мерфи Б., Нельсон К., Пфаннкоч С., Праттс Э., Пури В., Куреши Х., Рирдон М., Родригес Р., Роджерс Ю.Х., Ромблад Д., Руфел Б., Скотт Р., Ситтер С., Смоллвуд М., Стюарт E, Strong R, Suh E, Thomas R, Tint NN, Tse S, Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J, Zaveri K, Abril Дж.Ф., Гиго Р., Кэмпбелл М.Дж., Шоландер К.В., Карлак Б., Кежаривал А., Ми Х., Лазарева Б., Хаттон Т., Наречания А., Димер К., Муругануджан А., Го Н., Сато С., Бафна В., Истраил С., Липперт Р., Шварц Р., Валенц Б., Юсеф С., Аллен Д. , Басу А., Баксендейл Дж., Блик Л., Каминья М., Карнес-Стайн Дж., Колк П., Чан Ю.Х., Койн М., Далке К., Мейс А., Домброски М., Доннелли М., Эли Д., Эспархам С., Фослер С., Гире Х. , Глановски С., Глассер К., Глодек А., Горохов М., Грэм К., Гропман Б., Харрис М., Хейл Дж., Хендерсон С., Гувер Дж., Дженнингс Д. , Джордан С., Джордан Дж., Каша Дж., Каган Л., Крафт С., Левицкий А., Льюис М., Лю Х., Лопес Дж., Ма Д., Майорос В., Макдэниел Дж., Мерфи С., Ньюман М., Нгуен Т., Нгуен Н., Ноделл М., Пан С., Пек Дж., Петерсон М., Роу В., Сандерс Р., Скотт Дж., Симпсон М., Смит Т., Спраг А., Стоквелл Т., Тернер Р., Вентер Э., Ван М., Вен М., Ву Д., Ву М., Ся А., Занди А., Чжу Х. Последовательность генома человека. Наука. 2001;291: 1304–1351. [PubMed] [Google Scholar]
, Джордан С., Джордан Дж., Каша Дж., Каган Л., Крафт С., Левицкий А., Льюис М., Лю Х., Лопес Дж., Ма Д., Майорос В., Макдэниел Дж., Мерфи С., Ньюман М., Нгуен Т., Нгуен Н., Ноделл М., Пан С., Пек Дж., Петерсон М., Роу В., Сандерс Р., Скотт Дж., Симпсон М., Смит Т., Спраг А., Стоквелл Т., Тернер Р., Вентер Э., Ван М., Вен М., Ву Д., Ву М., Ся А., Занди А., Чжу Х. Последовательность генома человека. Наука. 2001;291: 1304–1351. [PubMed] [Google Scholar]
9. Waring M, Britten RJ. Повторение последовательности нуклеотидов: быстро реассоциирующая фракция мышиной ДНК. Наука. 1966; 154: 791–794. [PubMed] [Google Scholar]
10. Britten RJ, Kohne DE. Повторяющиеся последовательности в ДНК. Сотни тысяч копий последовательностей ДНК были включены в геномы высших организмов. Наука. 1968; 161: 529–540. [PubMed] [Google Scholar]
11. Beck CR, Garcia-Perez JL, Badge RM, Moran JV. Элементы LINE-1 в структурных изменениях и заболеваниях. Annu Rev Genomics Hum Genet. 2011;12:187–215. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
12. Белансио В.П., Рой-Энгель А.М., Дейнингер П.Л. Все, что вам нужно знать о ретроэлементах в раке. Семин Рак Биол. 2010;20:200–210. [Бесплатная статья PMC] [PubMed] [Google Scholar]
13. Левин Х.Л., Моран Дж.В. Динамические взаимодействия между подвижными элементами и их хостами. Нат Рев Жене. 2011;12:615–627. [Бесплатная статья PMC] [PubMed] [Google Scholar]
14. Hancks DC, Kazazian HH. Jr. Активные ретротранспозоны человека: вариации и болезни. Curr Opin Genet Dev. 2012;22:191–203. [Бесплатная статья PMC] [PubMed] [Google Scholar]
15. Ричардсон С.Р., Морелл С., Фолкнер Г.Дж. Ретротранспозоны L1 и соматический мозаицизм в головном мозге. Анну Рев Жене. 2014; 48:1–27. [PubMed] [Google Scholar]
16. Goodier JL, Kazazian HH, Jr Новый взгляд на ретротранспозоны: сдерживание и реабилитация паразитов. Клетка. 2008; 135:23–35. [PubMed] [Google Scholar]
17. Feschotte C. Мобильные элементы и эволюция регуляторных сетей. Нат Рев Жене. 2008;9: 397–405. [PMC free article] [PubMed] [Google Scholar]
Нат Рев Жене. 2008;9: 397–405. [PMC free article] [PubMed] [Google Scholar]
18. Бабушок Д.В., Казазян Х.Х., мл. Прогресс в понимании биологии человеческого мутагена LINE-1. Хум Мутат. 2007; 28: 527–539. [PubMed] [Google Scholar]
19. Burns KH, Boeke JD. Тектоника транспозонов человека. Клетка. 2012; 149:740–752. [Бесплатная статья PMC] [PubMed] [Google Scholar]
20. Lindblad-Toh K, Wade CM, Mikkelsen TS, Karlsson EK, Jaffe DB, Kamal M, Clamp M, Chang JL, Kulbokas EJ, 3rd, Zody MC, Маусели Э., Се Х, Брин М., Уэйн Р.К., Острандер Э.А., Понтинг С.П., Галиберт Ф., Смит Д.Р., ДеДжонг П.Дж., Киркнесс Э., Альварес П., Биаджи Т., Брокман В., Батлер Дж., Чин К.В., Кук А., Кафф Дж. , Дейли М.Дж., ДеКаприо Д., Гнерре С., Грабхерр М., Келлис М., Клебер М., Барделебен С., Гудштадт Л., Хегер А., Хитте С., Ким Л., Копфли К.П., Паркер Х.Г., Поллинджер Д.П., Сирл С.М., Саттер Н.Б., Томас Р., Уэббер С., Болдуин Дж., Абебе А., Абуэллей А., Афтук Л., Айт-Захра М., Олдридж Т., Аллен Н. , Ан П., Андерсон С., Антуан С., Арачи Х., Аслам А., Айотте Л., Бачанцанг П., Барри А, Баюл Т, Бенамара М, Берлин А, Бессет Д, Блитштейн Б, Блум Т, Блай Дж, Богуславский Л, Бонне С, Бухгалтер Б, Браун А, Кэхилл П, Каликсте Н, Камарата Дж, Чешацанг Ю, Чу Дж, Citroen M, Collymore A, Cooke P, Dawoe T, Daza R, Decktor K, DeGray S, Dhargay N, Dooley K, Dorje P, Dorjee K, Доррис Л., Даффи Н., Дупес А., Эгбиремолен О., Элонг Р., Фальк Дж., Фарина А., Фаро С., Фергюсон Д., Феррейра П., Фишер С., Фитцджеральд М., Фоули К., Фоли С., Франке А., Фридрих Д., Гейдж Д. , Гарбер М., Гиарин Г., Яннукос Г., Гуд Т., Гойетт А., Грэм Дж., Гранбуа Э., Гьялцен К., Хафез Н., Хагопян Д., Хагос Б., Холл Дж., Хили С., Хегарти Р., Хонан Т., Хорн А., Худе Н., Хьюз Л., Ханникатт Л., Хасби М., Джестер Б., Джонс С., Камат А., Канга Б., Келлс С., Хазанович Д., Кью А.С., Киснер П., Кумар М., Лэнс К., Ландерс Т., Лара М., Ли В., Леже Дж. П., Леннон Н., Леупер Л., ЛеВин С., Лю Дж., Лю Х, Локьитсанг Ю., Локьитсанг Т., Луи А.
, Ан П., Андерсон С., Антуан С., Арачи Х., Аслам А., Айотте Л., Бачанцанг П., Барри А, Баюл Т, Бенамара М, Берлин А, Бессет Д, Блитштейн Б, Блум Т, Блай Дж, Богуславский Л, Бонне С, Бухгалтер Б, Браун А, Кэхилл П, Каликсте Н, Камарата Дж, Чешацанг Ю, Чу Дж, Citroen M, Collymore A, Cooke P, Dawoe T, Daza R, Decktor K, DeGray S, Dhargay N, Dooley K, Dorje P, Dorjee K, Доррис Л., Даффи Н., Дупес А., Эгбиремолен О., Элонг Р., Фальк Дж., Фарина А., Фаро С., Фергюсон Д., Феррейра П., Фишер С., Фитцджеральд М., Фоули К., Фоли С., Франке А., Фридрих Д., Гейдж Д. , Гарбер М., Гиарин Г., Яннукос Г., Гуд Т., Гойетт А., Грэм Дж., Гранбуа Э., Гьялцен К., Хафез Н., Хагопян Д., Хагос Б., Холл Дж., Хили С., Хегарти Р., Хонан Т., Хорн А., Худе Н., Хьюз Л., Ханникатт Л., Хасби М., Джестер Б., Джонс С., Камат А., Канга Б., Келлс С., Хазанович Д., Кью А.С., Киснер П., Кумар М., Лэнс К., Ландерс Т., Лара М., Ли В., Леже Дж. П., Леннон Н., Леупер Л., ЛеВин С., Лю Дж., Лю Х, Локьитсанг Ю., Локьитсанг Т., Луи А. , Макдональд Дж., Мейджор Дж., Марабелла Р., Мару К., Мэтьюз С., Макдонаф С., Мехта Т., Мелдрим Дж. , Мельников А., Менеус Л., Михалев А., Михова Т., Миллер К., Миттельман Р., Мленга В., Малрейн Л., Мансон Г., Навиди А., Нейлор Дж., Нгуен Т., Нгуен Н., Нгуен С., Никол Р., Норбу Н., Норбу К., Новод Н., Ньима Т., Оландт П., О’Нил Б., О’Нил К., Осман С., Ойоно Л., Патти К., Перрин Д., Пхунханг П., Пьер Ф., Прист М., Рачупка А., Рагураман С. , Rameau R, Ray V, Raymond C, Rege F, Rise C, Rogers J, Rogov P, Sahalie J, Settipalli S, Sharpe T, Shea T, Sheehan M, Sherpa N, Shi J, Shih D, Sloan J, Smith К., Воробей Т., Сталкер Дж., Станге-Томанн Н., Ставропулос С., Стоун С., Стоун С., Сайкс С., Чуинга П., Тенцинг П., Тесфайе С., Тулутсанг Д., Тулутсанг Ю., Топхэм К., Топпинг И., Цамла Т., Васильев Х., Венкатараман В., Во А., Вангчук Т., Вангди Т., Вейанд М., Уилкинсон Дж., Уилсон А., Ядав С., Ян С., Ян Х., Янг Г., Ю К., Зайноун Дж., Зембек Л., Циммер А., Ландер Э.С. Последовательность генома, сравнительный анализ и структура гаплотипов домашней собаки.
, Макдональд Дж., Мейджор Дж., Марабелла Р., Мару К., Мэтьюз С., Макдонаф С., Мехта Т., Мелдрим Дж. , Мельников А., Менеус Л., Михалев А., Михова Т., Миллер К., Миттельман Р., Мленга В., Малрейн Л., Мансон Г., Навиди А., Нейлор Дж., Нгуен Т., Нгуен Н., Нгуен С., Никол Р., Норбу Н., Норбу К., Новод Н., Ньима Т., Оландт П., О’Нил Б., О’Нил К., Осман С., Ойоно Л., Патти К., Перрин Д., Пхунханг П., Пьер Ф., Прист М., Рачупка А., Рагураман С. , Rameau R, Ray V, Raymond C, Rege F, Rise C, Rogers J, Rogov P, Sahalie J, Settipalli S, Sharpe T, Shea T, Sheehan M, Sherpa N, Shi J, Shih D, Sloan J, Smith К., Воробей Т., Сталкер Дж., Станге-Томанн Н., Ставропулос С., Стоун С., Стоун С., Сайкс С., Чуинга П., Тенцинг П., Тесфайе С., Тулутсанг Д., Тулутсанг Ю., Топхэм К., Топпинг И., Цамла Т., Васильев Х., Венкатараман В., Во А., Вангчук Т., Вангди Т., Вейанд М., Уилкинсон Дж., Уилсон А., Ядав С., Ян С., Ян Х., Янг Г., Ю К., Зайноун Дж., Зембек Л., Циммер А., Ландер Э.С. Последовательность генома, сравнительный анализ и структура гаплотипов домашней собаки. Природа. 2005; 438:803–819. [PubMed] [Google Scholar]
Природа. 2005; 438:803–819. [PubMed] [Google Scholar]
21. Уотерстон Р. Х., Линдблад-Тох К., Бирни Э., Роджерс Дж., Абрил Дж. Ф., Агарвал П., Агарвала Р., Эйнско Р., Александрссон М., Ан П., Антонаракис С. Э., Эттвуд Дж., Бэрч Р., Бейли Дж., Барлоу К., Бек С., Берри Э., Биррен Б., Блум Т., Борк П., Ботчерби М., Брей Н., Брент М.Р., Браун Д.Г., Браун С.Д., Булт С., Бертон Дж., Батлер Дж., Кэмпбелл Р.Д., Карнинчи П., Коули С., Кьяромонте Ф., Чинвалла А.Т., Черч Д.М., Клэмп М., Кли С., Коллинз Ф.С., Кук Л.Л., Копли Р.Р., Коулсон А., Куронн О., Кафф Дж., Карвен В., Каттс Т., Дейли М., Дэвид Р. , Дэвис Дж., Делеханти К.Д., Дери Дж., Дермицакис Э.Т., Дьюи С., Диккенс Н.Дж., Диханс М., Додж С., Дубчак И., Данн Д.М., Эдди С.Р., Ельницки Л., Эмес Р.Д., Эсвара П., Эйрас Э., Фельзенфельд А., Фьюэлл GA, Flicek P, Foley K, Frankel WN, Fulton LA, Fulton RS, Furey TS, Gage D, Gibbs RA, Glusman G, Gnerre S, Goldman N, Goodstadt L, Grafham D, Graves TA, Green ED, Gregory S, Гиго Р., Гайер М., Хардисон Р. С., Хаусслер Д., Хаяшизаки Ю., Хиллиер Л.В., Хинрихс А., Хлавина В., Хольцер Т., Хсу Ф., Х. уа А, Хаббард Т, Хант А, Джексон И, Джаффе Д.Б., Джонсон Л.С., Джонс М., Джонс Т.А., Джой А., Камаль М., Карлссон Э.К., Карольчик Д., Каспршик А., Каваи Дж., Кейблер Э., Келлс С., Кент В.Дж. , Кирби А., Кольбе Д.Л., Корф И., Кучерлапати Р.С., Кулбокас Э.Дж., Кулп Д., Ландерс Т., Леже Д.П., Леонард С., Летуник И., Левин Р., Ли Дж., Ли М., Ллойд С., Лукас С., Ма Б., Маглотт DR, Mardis ER, Matthews L, Mauceli E, Mayer JH, McCarthy M, McCombie WR, McLaren S, McLay K, McPherson JD, Meldrim J, Meredith B, Mesirov JP, Miller W, Miner TL, Mongin E, Montgomery KT, Морган М., Мотт Р., Малликин Дж. К., Музны Д. М., Нэш В. Е., Нельсон Дж. О., Нхан М. Н., Никол Р., Нин З., Нусбаум С., О’Коннор М. Дж., Оказаки Ю., Оливер К., Овертон-Ларти Э., Пахтер Л., Парра Г., Пепин К.Х., Петерсон Дж., Певзнер П., Пламб Р., Пол К.С., Поляков А., Понсе Т.С., Понтинг С.П., Поттер С., Куэйл М., Реймонд А., Роу Б.А., Роскин К.М.
С., Хаусслер Д., Хаяшизаки Ю., Хиллиер Л.В., Хинрихс А., Хлавина В., Хольцер Т., Хсу Ф., Х. уа А, Хаббард Т, Хант А, Джексон И, Джаффе Д.Б., Джонсон Л.С., Джонс М., Джонс Т.А., Джой А., Камаль М., Карлссон Э.К., Карольчик Д., Каспршик А., Каваи Дж., Кейблер Э., Келлс С., Кент В.Дж. , Кирби А., Кольбе Д.Л., Корф И., Кучерлапати Р.С., Кулбокас Э.Дж., Кулп Д., Ландерс Т., Леже Д.П., Леонард С., Летуник И., Левин Р., Ли Дж., Ли М., Ллойд С., Лукас С., Ма Б., Маглотт DR, Mardis ER, Matthews L, Mauceli E, Mayer JH, McCarthy M, McCombie WR, McLaren S, McLay K, McPherson JD, Meldrim J, Meredith B, Mesirov JP, Miller W, Miner TL, Mongin E, Montgomery KT, Морган М., Мотт Р., Малликин Дж. К., Музны Д. М., Нэш В. Е., Нельсон Дж. О., Нхан М. Н., Никол Р., Нин З., Нусбаум С., О’Коннор М. Дж., Оказаки Ю., Оливер К., Овертон-Ларти Э., Пахтер Л., Парра Г., Пепин К.Х., Петерсон Дж., Певзнер П., Пламб Р., Пол К.С., Поляков А., Понсе Т.С., Понтинг С.П., Поттер С., Куэйл М., Реймонд А., Роу Б.А., Роскин К.М. , Рубин Э.М., Руст А.Г., Сантос Р., Сапожников В., Шульц Б., Шульц Дж., Шварц М.С., Шварц С., Скотт С., Симэн С., Сирл С., Шарп Т., Шеридан А., Шоункин Р., Симс С., Сингер Дж. Б., Слейтер Г., Смит А., Смит Д.Р., Спенсер Б., Стабенау А., Станге-Томанн Н., Сугнет С., Суяма М., Теслер Г., Томпсон Дж., Торрентс Д., Треваскис Э., Тромп Дж., Укла С., Урета-Видал А., Винсон Дж.П. , Von Niederhausern AC, Wade CM, Wall M, Weber RJ, Weiss RB, Wendl MC, West AP, Wetterstrand K, Wheeler R, Whelan S, Wierzbowski J, Willey D, Williams S, Wilson RK, Winter E, Worley KC, Вайман Д., Ян С., Ян С.П., Здобнов Э.М., Зоди М.С., Ландер Э.С. Первоначальное секвенирование и сравнительный анализ генома мыши. Природа. 2002; 420: 520–562. [PubMed] [Академия Google]
, Рубин Э.М., Руст А.Г., Сантос Р., Сапожников В., Шульц Б., Шульц Дж., Шварц М.С., Шварц С., Скотт С., Симэн С., Сирл С., Шарп Т., Шеридан А., Шоункин Р., Симс С., Сингер Дж. Б., Слейтер Г., Смит А., Смит Д.Р., Спенсер Б., Стабенау А., Станге-Томанн Н., Сугнет С., Суяма М., Теслер Г., Томпсон Дж., Торрентс Д., Треваскис Э., Тромп Дж., Укла С., Урета-Видал А., Винсон Дж.П. , Von Niederhausern AC, Wade CM, Wall M, Weber RJ, Weiss RB, Wendl MC, West AP, Wetterstrand K, Wheeler R, Whelan S, Wierzbowski J, Willey D, Williams S, Wilson RK, Winter E, Worley KC, Вайман Д., Ян С., Ян С.П., Здобнов Э.М., Зоди М.С., Ландер Э.С. Первоначальное секвенирование и сравнительный анализ генома мыши. Природа. 2002; 420: 520–562. [PubMed] [Академия Google]
22. де Конинг А.П., Гу В., Кастоу Т.А., Батцер М.А., Поллок Д.Д. Повторяющиеся элементы могут составлять более двух третей генома человека. Генетика PLoS. 2011;7:e1002384. [Бесплатная статья PMC] [PubMed] [Google Scholar]
23. Ivics Z, Hackett PB, Plasterk RH, Izsvak Z. Молекулярная реконструкция «Спящей красавицы», Tc1-подобного транспозона из рыб и его транспозиция в клетках человека. Клетка. 1997; 91: 501–510. [PubMed] [Google Scholar]
Молекулярная реконструкция «Спящей красавицы», Tc1-подобного транспозона из рыб и его транспозиция в клетках человека. Клетка. 1997; 91: 501–510. [PubMed] [Google Scholar]
24. Шапиро Дж.А. Молекулярная модель транспозиции и репликации бактериофага Mu и других мобильных элементов. Proc Natl Acad Sci U S A. 1979;76:1933–1937. [Бесплатная статья PMC] [PubMed] [Google Scholar]
25. Клекнер Н. Регуляция транспозиции у бактерий. Annu Rev Cell Biol. 1990; 6: 297–327. [PubMed] [Google Scholar]
26. Рио, округ Колумбия. Мобильные элементы P в Drosophila melanogaster . В: Craig NL, Craigie R, Gellert M, Lambowitz AM, редакторы. Мобильная ДНК II. Вашингтон, округ Колумбия: ASM Press; 2002. стр. 484–518. [Google Scholar]
27. Лазаров К., Долл М.Л., Кунце Р. Молекулярная биология элементов Ac/Ds кукурузы: обзор. Методы Мол Биол. 2013;1057:59–82. [PubMed] [Google Scholar]
28. Cary LC, Goebel M, Corsaro BG, Wang HG, Rosen E, Fraser MJ. Транспозонный мутагенез бакуловирусов: анализ инсерций транспозона IFP2 Trichoplusia ni в FP-локусе вирусов ядерного полиэдроза. Вирусология. 1989; 172: 156–169. [PubMed] [Google Scholar]
Вирусология. 1989; 172: 156–169. [PubMed] [Google Scholar]
29. Fraser MJ, Cary L, Boonvisudhi K, Wang HG. Анализ движения транспозона IFP2 чешуекрылых в клетках насекомых с использованием генома бакуловируса в качестве ДНК-мишени. Вирусология. 1995;211:397–407. [PubMed] [Google Scholar]
30. Ding S, Wu X, Li G, Han M, Zhuang Y, Xu T. Эффективная транспозиция транспозона piggyBac (PB) в клетках млекопитающих и мышей. Клетка. 2005; 122:473–483. [PubMed] [Google Scholar]
31. Rad R, Rad L, Wang W, Cadinanos J, Vassiliou G, Rice S, Campos LS, Yusa K, Banerjee R, Li MA, de la Rosa J, Strong A, Lu Д., Эллис П., Конте Н., Ян Ф.Т., Лю П., Брэдли А. Мутагенез транспозонов PiggyBac: инструмент для обнаружения генов рака у мышей. Наука. 2010; 330:1104–1107. [Бесплатная статья PMC] [PubMed] [Google Scholar]
32. Ivics Z, Li MA, Mates L, Boeke JD, Nagy A, Bradley A, Izsvak Z. Опосредованные транспозонами манипуляции с геномом у позвоночных. Нат Методы. 2009; 6: 415–422. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
33. Largaespada DA. Мутагенез транспозонов у мышей. Методы Мол Биол. 2009; 530:379–390. [Бесплатная статья PMC] [PubMed] [Google Scholar]
34. Ray DA, Feschotte C, Pagan HJ, Smith JD, Pritham EJ, Arensburger P, Atkinson PW, Craig NL. Множественные волны недавней активности ДНК-транспозонов у летучей мыши Myotis lucifugus. Геном Res. 2008; 18: 717–728. [Бесплатная статья PMC] [PubMed] [Google Scholar]
35. Рэй Д.А., Пэган Х.Дж., Томпсон М.Л., Стивенс Р.Д. Летучие мыши с hAT: свидетельство недавней активности ДНК-транспозонов у рода Myotis. Мол Биол Эвол. 2007; 24: 632–639. [PubMed] [Google Scholar]
36. Mitra R, Li X, Kapusta A, Mayhew D, Mitra RD, Feschotte C, Craig NL. Функциональная характеристика piggyBat летучей мыши Myotis lucifugus раскрывает активный транспозон ДНК млекопитающих. Proc Natl Acad Sci U S A. 2013;110:234–239. [Бесплатная статья PMC] [PubMed] [Google Scholar]
37. Пейс Дж. К., 2-й, Фешотт К. Эволюционная история транспозонов ДНК человека: свидетельство интенсивной активности в родословной приматов. Геном Res. 2007; 17: 422–432. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Эволюционная история транспозонов ДНК человека: свидетельство интенсивной активности в родословной приматов. Геном Res. 2007; 17: 422–432. [Бесплатная статья PMC] [PubMed] [Google Scholar]
38. Вольф Ю.Н. Превращение мусора в золото: одомашнивание мобильных элементов и создание новых генов у эукариот. Биоэссе. 2006; 28: 913–922. [PubMed] [Google Scholar]
39. Капитонов В.В., Юрка Дж. Сигнальные последовательности кора RAG1 и рекомбинации V(D)J получены из транспозонов Transib. PLoS биол. 2005;3:e181. [Бесплатная статья PMC] [PubMed] [Google Scholar]
40. Маджумдар С., Сингх А., Рио, округ Колумбия. Ген THAP9 человека кодирует активную ДНК-транспозазу Р-элемента. Наука. 2013;339: 446–448. [Бесплатная статья PMC] [PubMed] [Google Scholar]
41. Bannert N, Kurth R. Эволюционная динамика эндогенных ретровирусных семейств человека. Annu Rev Genomics Hum Genet. 2006; 7: 149–173. [PubMed] [Google Scholar]
42. Белшоу Р., Доусон А.Л., Вулвен-Аллен Дж., Реддинг Дж. , Берт А., Тристем М. Полногеномный скрининг выявил высокий уровень инсерционного полиморфизма в семействе эндогенных ретровирусов человека HERV-K(HML2). ): последствия для современной деятельности. Дж Вирол. 2005;79:12507–12514. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Берт А., Тристем М. Полногеномный скрининг выявил высокий уровень инсерционного полиморфизма в семействе эндогенных ретровирусов человека HERV-K(HML2). ): последствия для современной деятельности. Дж Вирол. 2005;79:12507–12514. [Бесплатная статья PMC] [PubMed] [Google Scholar]
43. Macfarlane C, Simmonds P. Аллельная изменчивость эндогенных ретровирусных элементов HERV-K(HML-2) в популяциях человека. Дж Мол Эвол. 2004; 59: 642–656. [PubMed] [Google Scholar]
44. Moyes D, Griffiths DJ, Venables PJ. Инсерционные полиморфизмы: новая жизнь для эндогенных ретровирусов при заболеваниях человека. Тенденции Жене. 2007; 23: 326–333. [PubMed] [Google Scholar]
45. Shin W, Lee J, Son SY, Ahn K, Kim HS, Han K. Вставка HERV-K, специфичная для человека, вызывает геномные вариации в геноме человека. ПЛОС Один. 2013;8:e60605. [Бесплатная статья PMC] [PubMed] [Google Scholar]
46. Хьюз Дж.Ф., Коффин Дж.М. Формирование соло-LTR эндогенного ретровируса К человека и инсерционные полиморфизмы: значение для эволюции человека и вирусов. Proc Natl Acad Sci U S A. 2004; 101:1668–1672. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Proc Natl Acad Sci U S A. 2004; 101:1668–1672. [Бесплатная статья PMC] [PubMed] [Google Scholar]
47. Yohn CT, Jiang Z, McGrath SD, Hayden KE, Khaitovich P, Johnson ME, Eichler MY, McPherson JD, Zhao S, Paabo S, Eichler EE. Специфичные для линии экспансии ретровирусных вставок в геномах африканских человекообразных обезьян, но не человека и орангутангов. PLoS биол. 2005;3:e110. [Бесплатная статья PMC] [PubMed] [Google Scholar]
48. Dewannieux M, Harper F, Richaud A, Letzelter C, Ribet D, Pierron G, Heidmann T. Идентификация инфекционного предшественника множественных копий эндогенных ретроэлементов HERV-K человека. Геном Res. 2006; 16: 1548–1556. [Бесплатная статья PMC] [PubMed] [Google Scholar]
49. Lee YN, Bieniasz PD. Восстановление инфекционного эндогенного ретровируса человека. PLoS Патог. 2007;3:e10. [Бесплатная статья PMC] [PubMed] [Google Scholar]
50. Stocking C, Kozak CA. Эндогенные ретровирусы мышей. Cell Mol Life Sci. 2008; 65: 3383–339.8. [Бесплатная статья PMC] [PubMed] [Google Scholar]
51. Максакова И.А., Романиш М.Т., Ганье Л., Данн К.А., ван де Лагемаат Л.Н., Магер Д.Л. Ретровирусные элементы и их хозяева: инсерционный мутагенез в зародышевой линии мышей. Генетика PLoS. 2006;2:е2. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Максакова И.А., Романиш М.Т., Ганье Л., Данн К.А., ван де Лагемаат Л.Н., Магер Д.Л. Ретровирусные элементы и их хозяева: инсерционный мутагенез в зародышевой линии мышей. Генетика PLoS. 2006;2:е2. [Бесплатная статья PMC] [PubMed] [Google Scholar]
52. Burton FH, Loeb DD, Voliva CF, Martin SL, Edgell MH, Hutchison CA., 3-я консервация у млекопитающих и обширная способность кодирования белков высоко повторяющейся ДНК длинная перемежающаяся последовательность один. Дж Мол Биол. 1986;187:291–304. [PubMed] [Google Scholar]
53. Смит А.Ф., Тот Г., Риггс А.Д., Юрка Дж. Предки, распространенные среди млекопитающих подсемейства повторяющихся последовательностей LINE-1. Дж Мол Биол. 1995; 246:401–417. [PubMed] [Google Scholar]
54. Ян Л., Брансфельд Дж., Скотт Л., Вичман Х. Оживление мертвых: история и реактивация вымершего l1. Генетика PLoS. 2014;10:e1004395. [Бесплатная статья PMC] [PubMed] [Google Scholar]
55. Хан Х., Смит А., Буассино С. Молекулярная эволюция и темп амплификации ретротранспозонов LINE-1 человека с момента возникновения приматов. Геном Res. 2006; 16:78–87. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Геном Res. 2006; 16:78–87. [Бесплатная статья PMC] [PubMed] [Google Scholar]
56. Джейкобс Ф.М., Гринберг Д., Нгуен Н., Хеусслер М., Эвинг А.Д., Кацман С., Патен Б., Салама С.Р., Хаусслер Д. Эволюционная гонка вооружений между генами цинковых пальцев KRAB ZNF91/93 и ретротранспозонами SVA/L1. Природа. 2014 [Epub перед печатью]. [Бесплатная статья PMC] [PubMed] [Google Scholar]
57. Castro-Diaz N, Ecco G, Coluccio A, Kapopoulou A, Yazdanpanah B, Friedli M, Duc J, Jang SM, Turelli P, Trono D. Эволюционно динамично Регуляция L1 в эмбриональных стволовых клетках. Гены Дев. 2014;28:1397–1409. [Бесплатная статья PMC] [PubMed] [Google Scholar]
58. Beck CR, Collier P, Macfarlane C, Malig M, Kidd JM, Eichler EE, Badge RM, Moran JV. Ретротранспозиционная активность LINE-1 в геномах человека. Клетка. 2010; 141:1159–1170. [Бесплатная статья PMC] [PubMed] [Google Scholar]
59. Brouha B, Schustak J, Badge RM, Lutz-Prigge S, Farley AH, Moran JV, Kazazian HH., Jr На горячие L1 приходится основная часть ретротранспозиции в человеческая популяция. Proc Natl Acad Sci U S A. 2003;100:5280–5285. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Proc Natl Acad Sci U S A. 2003;100:5280–5285. [Бесплатная статья PMC] [PubMed] [Google Scholar]
60. Moran JV, Holmes SE, Naas TP, DeBerardinis RJ, Boeke JD, Kazazian HH., Jr Высокочастотная ретротранспозиция в культивируемых клетках млекопитающих. Клетка. 1996; 87: 917–927. [PubMed] [Google Scholar]
61. Sassaman DM, Dombroski BA, Moran JV, Kimberland ML, Naas TP, DeBerardinis RJ, Gabriel A, Swergold GD, Kazazian HH., Jr Многие элементы L1 человека способны к ретротранспозиции. Нат Жене. 1997; 16:37–43. [PubMed] [Google Scholar]
62. Skowronski J, Fanning TG, Singer MF. Транскрипты линии-1 единичной длины в клетках тератокарциномы человека. Мол Селл Биол. 1988;8:1385–1397. [Бесплатная статья PMC] [PubMed] [Google Scholar]
63. Boissinot S, Chevret P, Furano AV. Эволюция и амплификация ретротранспозона L1 (LINE-1) в недавней истории человечества. Мол Биол Эвол. 2000;17:915–928. [PubMed] [Google Scholar]
64. Домброски Б.А., Матиас С.Л., Нантакумар Э. , Скотт А.Ф., Казазян Х.Х., мл. Изоляция активного мобильного элемента человека. Наука. 1991; 254:1805–1808. [PubMed] [Google Scholar]
, Скотт А.Ф., Казазян Х.Х., мл. Изоляция активного мобильного элемента человека. Наука. 1991; 254:1805–1808. [PubMed] [Google Scholar]
65. Овчинников И., Троксель А.Б., Свергольд Г.Д. Геномная характеристика недавних вставок LINE-1 человека: доказательства, подтверждающие случайную вставку. Геном Res. 2001; 11:2050–2058. [Бесплатная статья PMC] [PubMed] [Google Scholar]
66. Шин Ф.М., Шерри С.Т., Риш Г.М., Робишо М., Насидзе И., Стоункинг М., Батцер М.А., Свергольд Г.Д. Чтение между LINE: геномная вариация человека, вызванная ретротранспозицией LINE-1. Геном Res. 2000; 10:1496–1508. [Бесплатная статья PMC] [PubMed] [Google Scholar]
67. Badge RM, Alisch RS, Moran JV. ATLAS: система для выборочной идентификации специфичных для человека вставок L1. Am J Hum Genet. 2003; 72: 823–838. [Бесплатная статья PMC] [PubMed] [Google Scholar]
68. Ewing AD, Kazazian HH. Jr Высокопроизводительное секвенирование выявляет обширные различия в специфическом для человека содержании L1 в индивидуальных геномах человека. Геном Res. 2010;20:1262–1270. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Геном Res. 2010;20:1262–1270. [Бесплатная статья PMC] [PubMed] [Google Scholar]
69. Iskow RC, McCabe MT, Mills RE, Torene S, Pittard WS, Neuwald AF, Van Meir EG, Vertino PM, Devine SE. Естественный мутагенез геномов человека эндогенными ретротранспозонами. Клетка. 2010; 141:1253–1261. [Бесплатная статья PMC] [PubMed] [Google Scholar]
70. Boissinot S, Entezam A, Young L, Munson PJ, Furano AV. Инсерционная история активного семейства ретротранспозонов L1 у людей. Геном Res. 2004; 14:1221–1231. [Бесплатная статья PMC] [PubMed] [Google Scholar]
71. Myers JS, Vincent BJ, Udall H, Watkins WS, Morrish TA, Kilroy GE, Swergold GD, Henke J, Henke L, Moran JV, Jorde LB, Batzer Массачусетс Комплексный анализ недавно интегрированных элементов Ta L1 человека. Am J Hum Genet. 2002; 71: 312–326. [Бесплатная статья PMC] [PubMed] [Google Scholar]
72. Дурбин Р.М., Абекасис Г.Р., Альтшулер Д.Л., Аутон А., Брукс Л.Д., Гиббс Р.А., Херлс М.Е., Маквин Г.А. Карта вариаций генома человека на основе секвенирования в популяционном масштабе. Природа. 2010; 467:1061–1073. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Природа. 2010; 467:1061–1073. [Бесплатная статья PMC] [PubMed] [Google Scholar]
73. Grimaldi G, Skowronski J, Singer MF. Определение начала и конца сегментов семейства KpnI. EMBO J. 1984; 3: 1753–1759. [Бесплатная статья PMC] [PubMed] [Google Scholar]
74. Scott AF, Schmeckpeper BJ, Abdelrazik M, Comey CT, O’Hara B, Rossiter JP, Cooley T, Heath P, Smith KD, Margolet L. Origin of элементы L1 человека: предполагаемые гены-предшественники, полученные из консенсусной последовательности ДНК. Геномика. 1987;1:113–125. [Бесплатная статья PMC] [PubMed] [Google Scholar]
75. Swergold GD. Идентификация, характеристика и клеточная специфичность промотора LINE-1 человека. Мол Селл Биол. 1990; 10:6718–6729. [Бесплатная статья PMC] [PubMed] [Google Scholar]
76. Спик М. Антисмысловой промотор ретротранспозона L1 человека управляет транскрипцией соседних клеточных генов. Мол Селл Биол. 2001; 21:1973–1985. [Бесплатная статья PMC] [PubMed] [Google Scholar]
77. Holmes SE, Singer MF, Swergold GD. Исследования p40, белка, содержащего мотив лейциновой молнии, кодируемого первой открытой рамкой считывания активного мобильного элемента LINE-1 человека. Дж. Биол. Хим. 1992;267:19765–19768. [PubMed] [Google Scholar]
Исследования p40, белка, содержащего мотив лейциновой молнии, кодируемого первой открытой рамкой считывания активного мобильного элемента LINE-1 человека. Дж. Биол. Хим. 1992;267:19765–19768. [PubMed] [Google Scholar]
78. Hohjoh H, Singer MF. Цитоплазматические рибонуклеопротеиновые комплексы, содержащие человеческий белок LINE-1 и РНК. Эмбо Дж. 1996; 15: 630–639. [Бесплатная статья PMC] [PubMed] [Google Scholar]
79. Хазина Э., Трюффо В., Бюттнер Р., Шмидт С., Коулз М., Вейхенридер О. Тримерная структура и гибкость белка L1ORF1 при ретротранспозиции L1 человека. Nat Struct Mol Biol. 2011;18:1006–1014. [PubMed] [Google Scholar]
80. Мартин С.Л., Бушмен Ф.Д. Шаперонная активность нуклеиновой кислоты белка ORF1 из мышиного ретротранспозона LINE-1. Мол Селл Биол. 2001; 21: 467–475. [Бесплатная статья PMC] [PubMed] [Google Scholar]
81. Ergun S, Buschmann C, Heukeshoven J, Dammann K, Schnieders F, Lauke H, Chalajour F, Kilic N, Stratling WH, Schumann GG. Специфическая для типа клеток экспрессия открытых рамок считывания 1 и 2 LINE-1 в тканях плода и взрослого человека. Дж. Биол. Хим. 2004; 279:27753–27763. [PubMed] [Google Scholar]
Дж. Биол. Хим. 2004; 279:27753–27763. [PubMed] [Google Scholar]
82. Doucet AJ, Hulme AE, Sahinovic E, Kulpa DA, Moldovan JB, Kopera HC, Athanikar JN, Hasnaoui M, Bucheton A, Moran JV, Gilbert N. Характеристика рибонуклеопротеина LINE-1 частицы. Генетика PLoS. 2010;6:e1001150. [Бесплатная статья PMC] [PubMed] [Google Scholar]
83. Фэн К., Моран Дж., Казазян Х., Боке Д.Д. Ретротранспозон L1 человека кодирует консервативную эндонуклеазу, необходимую для ретротранспозиции. Клетка. 1996; 87: 905–916. [PubMed] [Google Scholar]
84. Mathias SL, Scott AF, Kazazian HH, Jr, Boeke JD, Gabriel A. Обратная транскриптаза, кодируемая мобильным элементом человека. Наука. 1991; 254:1808–1810. [PubMed] [Google Scholar]
85. Fanning T, Singer M. Последовательности ДНК LINE-1 в четырех отрядах млекопитающих предсказывают белки, сохраняющие гомологию с белками ретровируса. Нуклеиновые Кислоты Res. 1987;15:2251–2260. [Бесплатная статья PMC] [PubMed] [Google Scholar]
86. Goodier JL, Ostertag EM, Du K, Kazazian HH., Jr Новое активное подсемейство ретротранспозонов L1 у мышей. Геном Res. 2001; 11: 1677–1685. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Goodier JL, Ostertag EM, Du K, Kazazian HH., Jr Новое активное подсемейство ретротранспозонов L1 у мышей. Геном Res. 2001; 11: 1677–1685. [Бесплатная статья PMC] [PubMed] [Google Scholar]
87. Фурано А.В. Биологические свойства и эволюционная динамика ретротранспозонов LINE-1 млекопитающих. Prog Nucleic Acid Res Mol Biol. 2000; 64: 255–294. [PubMed] [Google Scholar]
88. Sookdeo A, Hepp CM, McClure MA, Boissinot S. Пересмотр эволюции мышиной LINE-1 в геномную эру. ДНК мафии. 2013;4:3. [Бесплатная статья PMC] [PubMed] [Google Scholar]
89. Adey NB, Tollefsbol TO, Sparks AB, Edgell MH, Hutchison CA., 3-е молекулярное восстановление вымершего предкового промотора для мыши L1. Proc Natl Acad Sci U S A. 1994; 91:1569–1573. [Бесплатная статья PMC] [PubMed] [Google Scholar]
90. Naas TP, DeBerardinis RJ, Moran JV, Ostertag EM, Kingsmore SF, Seldin MF, Hayashizaki Y, Martin SL, Kazazian HH. Активно ретротранспонирующее новое подсемейство мышиных элементов L1. EMBO J. 1998; 17: 590–597. [Бесплатная статья PMC] [PubMed] [Google Scholar]
1998; 17: 590–597. [Бесплатная статья PMC] [PubMed] [Google Scholar]
91. DeBerardinis RJ, Goodier JL, Ostertag EM, Kazazian HH. Jr Быстрая амплификация подсемейства ретротранспозонов приводит к эволюции генома мыши. Нат Жене. 1998; 20: 288–290. [PubMed] [Google Scholar]
92. Dewannieux M, Esnault C, Heidmann T. Опосредованная LINE ретротранспозиция маркированных последовательностей Alu. Нат Жене. 2003; 35:41–48. [PubMed] [Google Scholar]
93. Dewannieux M, Heidmann T. L1-опосредованная ретротранспозиция мышиных B1 и B2 SINE, рекапитулированных в культивируемых клетках. Дж Мол Биол. 2005;349: 241–247. [PubMed] [Google Scholar]
94. Raiz J, Damert A, Chira S, Held U, Klawitter S, Hamdorf M, Lower J, Stratling WH, Lower R, Schumann GG. Неавтономный ретротранспозон SVA трансмобилизуется с помощью белкового механизма LINE-1 человека. Нуклеиновые Кислоты Res. 2012;40:1666–1683. [Статья бесплатно PMC] [PubMed] [Google Scholar]
95. Hancks DC, Goodier JL, Mandal PK, Cheung LE, Kazazian HH. , Jr Ретротранспозиция меченых элементов SVA человеческими L1 в культивируемых клетках. Хум Мол Жене. 2011;20:3386–3400. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Jr Ретротранспозиция меченых элементов SVA человеческими L1 в культивируемых клетках. Хум Мол Жене. 2011;20:3386–3400. [Бесплатная статья PMC] [PubMed] [Google Scholar]
96. Hancks DC, Mandal PK, Cheung LE, Kazazian HH., Jr Минимально активный ретротранспозон SVA человека требует только 5′-гексамера и Alu-подобных доменов. Мол Селл Биол. 2012;32:4718–4726. [Бесплатная статья PMC] [PubMed] [Google Scholar]
97. Esnault C, Maestre J, Heidmann T. Ретротранспозоны человека LINE генерируют обработанные псевдогены. Нат Жене. 2000; 24: 363–367. [PubMed] [Google Scholar]
98. Wei W, Gilbert N, Ooi SL, Lawler JF, Ostertag EM, Kazazian HH, Boeke JD, Moran JV. Ретротранспозиция L1 человека: цис-предпочтение по сравнению с транс-комплементацией. Мол Селл Биол. 2001;21:1429–1439. [Бесплатная статья PMC] [PubMed] [Google Scholar]
99. Юрка Дж. Паттерны последовательности указывают на ферментативное участие в интеграции ретропозонов млекопитающих. Proc Natl Acad Sci U S A. 1997; 94:1872–1877. [Бесплатная статья PMC] [PubMed] [Google Scholar]
1997; 94:1872–1877. [Бесплатная статья PMC] [PubMed] [Google Scholar]
100. Rubin CM, Houck CM, Deininger PL, Friedmann T, Schmid CW. Частичная нуклеотидная последовательность из 300 нуклеотидов перемежается повторяющимися последовательностями ДНК человека. Природа. 1980; 284: 372–374. [PubMed] [Google Scholar]
101. Ullu E, Tschudi C. Последовательности Alu представляют собой процессированные гены РНК 7SL. Природа. 1984;312:171–172. [PubMed] [Google Scholar]
102. Чу В.М., Лю В.М., Шмид К.В. Элементы промотора и терминатора РНК-полимеразы III влияют на экспрессию Alu РНК. Нуклеиновые Кислоты Res. 1995; 23:1750–1757. [Бесплатная статья PMC] [PubMed] [Google Scholar]
103. Batzer MA, Deininger PL. Повторы Alu и геномное разнообразие человека. Нат Рев Жене. 2002; 3: 370–379. [PubMed] [Google Scholar]
104. Чесноков И., Шмид К.В. Фланкирующие последовательности источника Alu стимулируют транскрипцию in vitro путем взаимодействия со специфическими для последовательности факторами транскрипции. Дж Мол Эвол. 1996;42:30–36. [PubMed] [Google Scholar]
Дж Мол Эвол. 1996;42:30–36. [PubMed] [Google Scholar]
105. Comeaux MS, Roy-Engel AM, Hedges DJ, Deininger PL. Различные цис-факторы, контролирующие ретротранспозицию Alu: что вызывает гибель элементов Alu? Геном Res. 2009; 19: 545–555. [Бесплатная статья PMC] [PubMed] [Google Scholar]
106. Dewannieux M, Heidmann T. Роль длины хвоста поли(A) в ретротранспозиции Alu. Геномика. 2005; 86: 378–381. [PubMed] [Google Scholar]
107. Goodier JL, Maraia RJ. Специфичная для терминатора рециркуляция транскрипционного комплекса B1-Alu с помощью РНК-полимеразы III опосредуется РНК-концевым связывающим белком La. J Biol Chem. 1998;273:26110–26116. [PubMed] [Google Scholar]
108. Liu WM, Schmid CW. Предполагаемая роль метилирования ДНК в репрессии транскрипции Alu и мутационной инактивации. Нуклеиновые Кислоты Res. 1993; 21:1351–1359. [Бесплатная статья PMC] [PubMed] [Google Scholar]
109. Ullu E, Weiner AM. Восходящие последовательности модулируют внутренний промотор гена РНК 7SL человека. Природа. 1985; 318: 371–374. [PubMed] [Google Scholar]
Природа. 1985; 318: 371–374. [PubMed] [Google Scholar]
110. Юрка Дж., Смит Т. Фундаментальное подразделение в семействе повторяющихся последовательностей Alu. Proc Natl Acad Sci U S A. 1988;85:4775–4778. [Бесплатная статья PMC] [PubMed] [Google Scholar]
111. Slagel V, Flemington E, Traina-Dorge V, Bradshaw H, Deininger P. Кластеризация и подсемейные отношения семейства Alu в геноме человека. Мол Биол Эвол. 1987; 4: 19–29. [PubMed] [Google Scholar]
112. Britten RJ, Baron WF, Stout DB, Davidson EH. Источники и эволюция повторяющихся последовательностей Alu человека. Proc Natl Acad Sci U S A. 1988; 85:4770–4774. [Бесплатная статья PMC] [PubMed] [Google Scholar]
113. Willard C, Nguyen HT, Schmid CW. Существование как минимум трех различных подсемейств Alu. Дж Мол Эвол. 1987;26:180–186. [PubMed] [Google Scholar]
114. Batzer MA, Deininger PL, Hellmann-Blumberg U, Jurka J, Labuda D, Rubin CM, Schmid CW, Zietkiewicz E, Zuckerkandl E. Стандартизированная номенклатура для повторов Alu. Дж Мол Эвол. 1996; 42:3–6. [PubMed] [Google Scholar]
Дж Мол Эвол. 1996; 42:3–6. [PubMed] [Google Scholar]
115. Batzer MA, Schmid CW, Deininger PL. Эволюционный анализ повторяющихся последовательностей ДНК. Методы Энзимол. 1993; 224: 213–232. [PubMed] [Google Scholar]
116. Кэрролл М.Л., Рой-Энгель А.М., Нгуен С.В., Салем А.Х., Фогель Э., Винсент Б., Майерс Дж., Ахмад З., Нгуен Л., Саммарко М., Уоткинс В.С., Хенке Дж., Макаловский W, Jorde LB, Deininger PL, Batzer MA. Крупномасштабный анализ подсемейств Alu Ya5 и Yb8 и их вклад в геномное разнообразие человека. Дж Мол Биол. 2001; 311:17–40. [PubMed] [Академия Google]
117. Уизерспун Д.Дж., Чжан И., Син Дж., Уоткинс В.С., Ха Х., Батцер М.А., Джорд Л.Б. Мобильное сканирование элементов (ME-Scan) идентифицирует тысячи новых вставок Alu в различных человеческих популяциях. Геном Res. 2013;23:1170–1181. [Бесплатная статья PMC] [PubMed] [Google Scholar]
118. Мингетти П.П., Дугайчик А. Появление новых повторов ДНК и дивергенция приматов. Proc Natl Acad Sci U S A. 1993; 90:1872–1876. [Бесплатная статья PMC] [PubMed] [Google Scholar]
1993; 90:1872–1876. [Бесплатная статья PMC] [PubMed] [Google Scholar]
119. Batzer MA, Stoneking M, Alegria-Hartman M, Bazan H, Kass DH, Shaikh TH, Novick GE, Ioannou PA, Scheer WD, Herrera RJ, et al . Африканское происхождение специфичных для человека полиморфных вставок Alu. Proc Natl Acad Sci U S A. 1994;91:12288–12292. [Бесплатная статья PMC] [PubMed] [Google Scholar]
120. Никайдо М., Руни А.П., Окада Н. Филогенетические отношения между цетартиодактилями, основанные на вставках коротких и длинных вставок: гиппопотамы — ближайшие из ныне живущих родственников китов. Proc Natl Acad Sci U S A. 1999; 96:10261–10266. [Бесплатная статья PMC] [PubMed] [Google Scholar]
121. Окада Н. СИНЕС. Curr Opin Genet Dev. 1991; 1: 498–504. [PubMed] [Google Scholar]
122. Ray DA, Xing J, Salem AH, Batzer MA. СИНУСы почти идеального характера. Сист биол. 2006;55:928–935. [PubMed] [Google Scholar]
123. Уизерспун Д.Дж., Марчани Э.Е., Уоткинс В.С., Остлер К.Т., Вудинг С. П., Андерс Б.А., Фаулкс Д.Д., Буассино С., Фурано А.В., Рэй Д.А., Роджерс А.Р., Батцер М.А., Джорде Л.Б. Генетическая структура и разнообразие популяции человека, полученные на основе полиморфных вставок L1 (LINE-1) и Alu. Хам Херед. 2006; 62:30–46. [PubMed] [Google Scholar]
П., Андерс Б.А., Фаулкс Д.Д., Буассино С., Фурано А.В., Рэй Д.А., Роджерс А.Р., Батцер М.А., Джорде Л.Б. Генетическая структура и разнообразие популяции человека, полученные на основе полиморфных вставок L1 (LINE-1) и Alu. Хам Херед. 2006; 62:30–46. [PubMed] [Google Scholar]
124. Konkel MK, Walker JA, Batzer MA. ЛИНИИ и СИНУСЫ эволюции приматов. Эвол Антропол. 2010; 19: 236–249. [Бесплатная статья PMC] [PubMed] [Google Scholar]
125. Bennett EA, Keller H, Mills RE, Schmidt S, Moran JV, Weichenrieder O, Devine SE. Активные ретротранспозоны Alu в геноме человека. Геном Res. 2008; 18:1875–1883. [Бесплатная статья PMC] [PubMed] [Google Scholar]
126. Sarrowa J, Chang DY, Maraia RJ. Снижение ретропозиции Alu человека сопровождалось асимметричным снижением связывания SRP9/14 с димерной Alu РНК и повышенной экспрессией малой цитоплазматической Alu РНК. Мол Селл Биол. 1997; 17:1144–1151. [Бесплатная статья PMC] [PubMed] [Google Scholar]
127. Ostertag EM, Goodier JL, Zhang Y, Kazazian HH. , Jr Элементы SVA представляют собой неавтономные ретротранспозоны, вызывающие заболевания у людей. Am J Hum Genet. 2003; 73: 1444–1451. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Jr Элементы SVA представляют собой неавтономные ретротранспозоны, вызывающие заболевания у людей. Am J Hum Genet. 2003; 73: 1444–1451. [Бесплатная статья PMC] [PubMed] [Google Scholar]
128. Wang H, Xing J, Grover D, Hedges DJ, Han K, Walker JA, Batzer MA. Элементы SVA: специфичное для гоминидов семейство ретропозонов. Дж Мол Биол. 2005; 354: 994–1007. [PubMed] [Google Scholar]
129. Hancks DC, Kazazian HH., Jr Ретротранспозоны SVA: эволюция и генетическая нестабильность. Семин Рак Биол. 2010;20:234–245. [Бесплатная статья PMC] [PubMed] [Google Scholar]
130. Сэвидж А.Л., Бабб В.Дж., Брин Г., Куинн Дж.П. Характеристика потенциальной функции ретротранспозонов SVA для модуляции паттернов экспрессии генов. БМС Эвол Биол. 2013;13:101. [Бесплатная статья PMC] [PubMed] [Google Scholar]
131. Hara T, Hirai Y, Baicharoen S, Hayakawa T, Hirai H, Koga A. Новый композитный ретротранспозон, полученный из SVA или созданный независимо от него (SINE/VNTR). /Alu) транспозон подвергся пролиферации в геномах гиббонов. Гены Генет Сист. 2012; 87: 181–190. [PubMed] [Google Scholar]
Гены Генет Сист. 2012; 87: 181–190. [PubMed] [Google Scholar]
132. Ianc B, Ochis C, Persch R, Popescu O, Damert A. Hominoid Composite Non-LTR Retrotransposons — разнообразие, сборка, эволюция и структурные детерминанты мобилизации. Мол Биол Эвол. 2014;31:2847–2864. [PubMed] [Google Scholar]
133. Carbone L, Harris RA, Mootnick AR, Milosavljevic A, Martin DI, Rocchi M, Capozzi O, Archidiacono N, Konkel MK, Walker JA, Batzer MA, de Jong PJ. Ремоделирование центромеры у Hoolock leuconedys (Hylobatidae) с помощью нового подвижного элемента, уникального для гиббонов. Геном Биол Эвол. 2012; 4: 648–658. [Бесплатная статья PMC] [PubMed] [Google Scholar]
134. Карбоне Л., Харрис Р.А., Гнерре С., Вирама К.Р., Лоренте-Галдос Б., Хаддлстон Дж., Мейер Т.Дж., Эрреро Дж., Рус С., Акен Б., Анаклерио Ф., Архидиаконо Н., Бейкер С., Баррелл Д., Батцер М.А. , Бил К., Бланчер А., Борсон С.Л., Брамейер М., Кэмпбелл М.С., Капоцци О., Касола С., Чиатанте Г., Кри А., Дамерт А., де Йонг П. Дж., Дюма Л., Фернандес-Каллехо М., Фличек П., Фукс Н.В., Гут Я, Гут М., Хан М.В., Эрнандес-Родригес Дж., Хиллиер Л.В., Хабли Р., Янк Б., Изсвак З., Яблонски Н.Г., Джонстон Л.М., Каримпур-Фард А., Конкель М.К., Костка Д., Лазар Н.Х., Ли С.Л., Льюис Л.Р. , Лю Ю., Локк Д.П., Маллик С., Мендес Ф.Л., Муффато М., Назарет Л.В., Невонен К.А., О’Бленесс М., Очис С., Одом Д.Т., Поллард К.С., Килез Дж., Райх Д., Рокки М., Шуман Г.Г., Сирл С. , Сикела Дж. М., Сколлар Дж., Смит А., Сонмез К., Тен Халлерс Б., Терхун Э., Томас Г.В., Уллмер Б., Вентура М., Уокер Дж.А., Уолл Дж.Д., Уолтер Л., Уорд М.С., Уилан С.Дж., Уилан К.В., Уайт С., Вильгельм Л.Дж., Вернер А.Е., Янделл М., Чжу Б., Хаммер М.Ф., Маркес-Боне Т., Эйхлер Э.Е., Фултон Л., Фроник С., Музный Д.М., Варре n WC, Уорли К.С., Роджерс Дж., Уилсон Р.К., Гиббс Р.А. Геном гиббона и быстрая эволюция кариотипа мелких обезьян. Природа. 2014;513:195–201. [PMC free article] [PubMed] [Google Scholar]
Дж., Дюма Л., Фернандес-Каллехо М., Фличек П., Фукс Н.В., Гут Я, Гут М., Хан М.В., Эрнандес-Родригес Дж., Хиллиер Л.В., Хабли Р., Янк Б., Изсвак З., Яблонски Н.Г., Джонстон Л.М., Каримпур-Фард А., Конкель М.К., Костка Д., Лазар Н.Х., Ли С.Л., Льюис Л.Р. , Лю Ю., Локк Д.П., Маллик С., Мендес Ф.Л., Муффато М., Назарет Л.В., Невонен К.А., О’Бленесс М., Очис С., Одом Д.Т., Поллард К.С., Килез Дж., Райх Д., Рокки М., Шуман Г.Г., Сирл С. , Сикела Дж. М., Сколлар Дж., Смит А., Сонмез К., Тен Халлерс Б., Терхун Э., Томас Г.В., Уллмер Б., Вентура М., Уокер Дж.А., Уолл Дж.Д., Уолтер Л., Уорд М.С., Уилан С.Дж., Уилан К.В., Уайт С., Вильгельм Л.Дж., Вернер А.Е., Янделл М., Чжу Б., Хаммер М.Ф., Маркес-Боне Т., Эйхлер Э.Е., Фултон Л., Фроник С., Музный Д.М., Варре n WC, Уорли К.С., Роджерс Дж., Уилсон Р.К., Гиббс Р.А. Геном гиббона и быстрая эволюция кариотипа мелких обезьян. Природа. 2014;513:195–201. [PMC free article] [PubMed] [Google Scholar]
135. Краев А.С., Крамеров Д.А., Скрябин К.Г., Рысков А. П., Баев А.А., Георгиев Г.П. Нуклеотидная последовательность вездесущей повторяющейся последовательности ДНК B1 комплементарна наиболее распространенному классу мышиных фолд-бэк РНК. Нуклеиновые Кислоты Res. 1980; 8: 1201–1215. [Бесплатная статья PMC] [PubMed] [Google Scholar]
П., Баев А.А., Георгиев Г.П. Нуклеотидная последовательность вездесущей повторяющейся последовательности ДНК B1 комплементарна наиболее распространенному классу мышиных фолд-бэк РНК. Нуклеиновые Кислоты Res. 1980; 8: 1201–1215. [Бесплатная статья PMC] [PubMed] [Google Scholar]
136. Квентин Ю. Мастер-последовательность, связанная со свободным левым мономером Alu (FLAM) в происхождении семейства B1 в геномах грызунов. Нуклеиновые Кислоты Res. 1994;22:2222–2227. [Бесплатная статья PMC] [PubMed] [Google Scholar]
137. Daniels GR, Deininger PL. Семейства повторяющихся последовательностей, полученные из генов тРНК млекопитающих. Природа. 1985; 317: 819–822. [PubMed] [Google Scholar]
138. DeChiara TM, Brosius J. Neural BC1 RNA: клоны кДНК обнаруживают неповторяющиеся последовательности. Proc Natl Acad Sci U S A. 1987; 84:2624–2628. [Бесплатная статья PMC] [PubMed] [Google Scholar]
139. Lee IY, Westaway D, Smit AF, Wang K, Seto J, Chen L, Acharya C, Ankener M, Baskin D, Cooper C, Yao H, Prusiner СБ, Худ ЛЭ. Полная геномная последовательность и анализ области гена прионного белка трех видов млекопитающих. Геном Res. 1998;8:1022–1037. [PubMed] [Google Scholar]
Полная геномная последовательность и анализ области гена прионного белка трех видов млекопитающих. Геном Res. 1998;8:1022–1037. [PubMed] [Google Scholar]
140. Сердобова И.М., Крамеров Д.А. Короткие ретропозоны надсемейства В2: эволюция и применение для изучения филогении грызунов. Дж Мол Эвол. 1998; 46: 202–214. [PubMed] [Google Scholar]
141. Вайнер А.М., Дейнингер П.Л., Эфстратиадис А. Невирусные ретропозоны: гены, псевдогены и мобильные элементы, генерируемые обратным потоком генетической информации. Анну Рев Биохим. 1986; 55: 631–661. [PubMed] [Google Scholar]
142. Ванин Е.Ф. Процессированные псевдогены: характеристики и эволюция. Анну Рев Жене. 1985;19:253–272. [PubMed] [Google Scholar]
143. Торрентс Д., Суяма М., Здобнов Э., Борк П. Полногеномный обзор псевдогенов человека. Геном Res. 2003; 13: 2559–2567. [Бесплатная статья PMC] [PubMed] [Google Scholar]
144. Чжан З., Харрисон П., Герштейн М. Идентификация и анализ более 2000 псевдогенов рибосомных белков в геноме человека. Геном Res. 2002; 12:1466–1482. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Геном Res. 2002; 12:1466–1482. [Бесплатная статья PMC] [PubMed] [Google Scholar]
145. Zhang Z, Harrison PM, Liu Y, Gerstein M. Миллионы лет эволюции сохранены: полный каталог обработанных псевдогенов в геноме человека. Геном Res. 2003; 13: 2541–2558. [Бесплатная статья PMC] [PubMed] [Google Scholar]
146. Ewing AD, Ballinger TJ, Earl D, Harris CC, Ding L, Wilson RK, Haussler D. Ретротранспозиция транскриптов генов приводит к структурным изменениям в геномах млекопитающих. Геном биол. 2013;14:R22. [Бесплатная статья PMC] [PubMed] [Google Scholar]
147. Schrider DR, Navarro FC, Galante PA, Parmigiani RB, Camargo AA, Hahn MW, de Souza SJ. Полиморфизм числа копий гена, вызванный ретротранспозицией у человека. Генетика PLoS. 2013;9:e1003242. [Бесплатная статья PMC] [PubMed] [Google Scholar]
148. Абызов А., Исков Р., Гоккумен О., Радке Д.В., Баласубраманян С., Пей Б., Хабеггер Л., Ли С., Герштейн М. Анализ различных ретродупликаций в популяциях человека предполагает связь ретротранспозиции с делением клеток. Геном Res. 2013;23:2042–2052. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Геном Res. 2013;23:2042–2052. [Бесплатная статья PMC] [PubMed] [Google Scholar]
149. Richardson SR, Salvador-Palomeque C, Faulkner GJ. Разнообразие за счет дублирования: полногеномное секвенирование выявляет новые ретрокопии генов в человеческой популяции. Биоэссе. 2014; 36: 475–481. [Бесплатная статья PMC] [PubMed] [Google Scholar]
150. Kazazian HH., Jr Процессированные псевдогенные вставки в соматических клетках. ДНК мафии. 2014; 5:20. [Бесплатная статья PMC] [PubMed] [Google Scholar]
151. Hulme AE, Kulpa DA, Garcia-Perez JL, Moran JV. В: Влияние ретротранспозиции LINE-1 на геном человека. Лупски Дж. Р., Станкевич П., редакторы. Тотова, Нью-Джерси: Humana Press; 2006. С. 35–72. [Google Scholar]
152. Sayah DM, Sokolskaja E, Berthoux L, Luban J. Ретротранспозиция циклофилина A в TRIM5 объясняет резистентность совиных обезьян к ВИЧ-1. Природа. 2004;430:569–573. [PubMed] [Google Scholar]
153. Малфавон-Борха Р., Ву Л.И., Эмерман М., Малик Х.С. Рождение, распад и реконструкция древнего слияния генов TRIMCyp в геномах приматов. Proc Natl Acad Sci U S A. 2013;110:E583–E592. [PMC free article] [PubMed] [Google Scholar]
Proc Natl Acad Sci U S A. 2013;110:E583–E592. [PMC free article] [PubMed] [Google Scholar]
154. Буздин А., Гогвадзе Е., Ковальская Е., Волчков П., Устюгова С., Илларионова А., Фушан А., Виноградова Т., Свердлов Е. Геном человека содержит множество типов химерные ретрогены, генерируемые в результате рекомбинации РНК in vivo. Нуклеиновые Кислоты Res. 2003; 31: 4385–439.0. [PMC free article] [PubMed] [Google Scholar]
155. Буздин А., Устюгова С., Гогвадзе Е., Виноградова Т., Лебедев Ю., Свердлов Е. Новое семейство химерных ретротранскриптов, образованных полной копией U6 small ядерная РНК, слитая с 3′-концом l1. Геномика. 2002; 80: 402–406. [PubMed] [Google Scholar]
156. Гилберт Н., Лутц С., Морриш Т.А., Моран Дж.В. Множественные судьбы интермедиатов ретротранспозиции L1 в культивируемых клетках человека. Мол Селл Биол. 2005; 25:7780–7795. [Бесплатная статья PMC] [PubMed] [Google Scholar]
157. Garcia-Perez JL, Doucet AJ, Bucheton A, Moran JV, Gilbert N. Различные механизмы транс-опосредованной мобилизации клеточных РНК обратной транскриптазой LINE-1. Геном Res. 2007; 17: 602–611. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Геном Res. 2007; 17: 602–611. [Бесплатная статья PMC] [PubMed] [Google Scholar]
158. Weber MJ. Малые ядрышковые РНК млекопитающих являются мобильными генетическими элементами. Генетика PLoS. 2006;2:e205. [Статья бесплатно PMC] [PubMed] [Google Scholar]
159. Hasnaoui M, Doucet AJ, Meziane O, Gilbert N. Последовательность древних повторов, полученная из snRNA U6 в геномах приматов. Ген. 2009 г.;448:139–144. [PubMed] [Google Scholar]
160. Kazazian HH, Jr, Wong C, Youssoufian H, Scott AF, Phillips DG, Antonarakis SE. Гемофилия А, возникающая в результате вставки de novo последовательностей L1, представляет собой новый механизм мутации у человека. Природа. 1988; 332: 164–166. [PubMed] [Google Scholar]
161. Домброски Б.А., Скотт А.Ф., Казазян Х.Х., мл. Два дополнительных потенциальных ретротранспозона, выделенных из подсемейства L1 человека, которые содержат активный ретротранспозонный элемент. Proc Natl Acad Sci U S A. 1993;90:6513–6517. [Бесплатная статья PMC] [PubMed] [Google Scholar]
162. Лутц С.М., Винсент Б.Дж., Казазян Х.Х., младший, Батцер М.А., Моран Дж.В. Аллельная гетерогенность в ретротранспозиционной активности LINE-1. Am J Hum Genet. 2003; 73: 1431–1437. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Лутц С.М., Винсент Б.Дж., Казазян Х.Х., младший, Батцер М.А., Моран Дж.В. Аллельная гетерогенность в ретротранспозиционной активности LINE-1. Am J Hum Genet. 2003; 73: 1431–1437. [Бесплатная статья PMC] [PubMed] [Google Scholar]
163. Skowronski J, Singer MF. Экспрессия цитоплазматического транскрипта LINE-1 регулируется в клеточной линии тератокарциномы человека. Proc Natl Acad Sci U S A. 1985; 82:6050–6054. [Бесплатная статья PMC] [PubMed] [Google Scholar]
164. Уоллес М.Р., Андерсен Л.Б., Саулино А.М., Грегори П.Е., Гловер Т.В., Коллинз Ф.С. Вставка Alu de novo приводит к нейрофиброматозу 1 типа. Природа. 1991; 353: 864–866. [PubMed] [Google Scholar]
165. Hassoun H, Coetzer TL, Vassiliadis JN, Sahr KE, Maalouf GJ, Saad ST, Catanzariti L, Palek J. Новый мобильный элемент, встроенный в ген альфа-спектрина: спектрин дайтон. Усеченный альфа-спектрин связан с наследственным эллиптоцитозом. Джей Клин Инвест. 1994; 94: 643–648. [Бесплатная статья PMC] [PubMed] [Google Scholar]
166. Ван дер Клифт Х.М., Топс К.М., Хес Ф.Дж., Девили П., Вийнен Дж.Т. Вставка элемента SVA, неавтономного ретротранспозона, в интрон 7 PMS2 как новая причина синдрома Линча. Хум Мутат. 2012;33:1051–1055. [PubMed] [Google Scholar]
Ван дер Клифт Х.М., Топс К.М., Хес Ф.Дж., Девили П., Вийнен Дж.Т. Вставка элемента SVA, неавтономного ретротранспозона, в интрон 7 PMS2 как новая причина синдрома Линча. Хум Мутат. 2012;33:1051–1055. [PubMed] [Google Scholar]
167. Vogt J, Bengesser K, Claes KB, Wimmer K, Mautner VF, van Minkelen R, Legius E, Brems H, Upadhyaya M, Hogel J, Lazaro C, Rosenbaum T, Bammert S , Messiaen L, Cooper DN, Kehrer-Sawatzki H. Делеция, связанная со вставкой ретротранспозона SVA, представляет собой новый мутационный механизм, лежащий в основе больших изменений числа копий генома с неповторяющимися точками останова. Геном биол. 2014;15:R80. [Бесплатная статья PMC] [PubMed] [Google Scholar]
168. де Бур М., ван Леувен К., Гейсслер Дж., Вимаес К.М., ван ден Берг Т.К., Куиджперс Т.В., Варрис А., Роос Д. Первичный иммунодефицит, вызванный экзонизированной ретропозированной копией гена, вставленной в ген CYBB. Хум Мутат. 2014; 35: 486–496. [PubMed] [Google Scholar]
169. Кук С.Л., Шлиен А. , Маршалл Дж., Пипиникас С.П., Мартинкорена И., Тубио Дж.М., Ли И., Мензис А., Муди Л., Рамакришна М., Йейтс Л., Дэвис Х., Болли Н., Бигнелл Г.Р., Тарпи П.С., Беджати С., Ник-Зайнал С., Папаэммануил Э., Тейшейра В.Х., Рейн К., О’Мира С., Додоран М.С., Тиг Дж.В., Батлер А.П., Якобуцио-Донахью С., Сантариус Т., Гранди Р.Г., Малкин Д. , Гривз М., Мунши Н., Фланаган А.М., Боутелл Д., Мартин С., Ларсимонт Д., Рейс-Фильо Дж.С., Буссиутас А., Тейлор Дж.А., Хейс Н.Д., Джейнс С.М., Футреал П.А., Стрэттон М.Р., Макдермотт У., Кэмпбелл П.Дж. Процессированные псевдогены, приобретенные соматически при развитии рака. Нац коммун. 2014;5:3644. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Маршалл Дж., Пипиникас С.П., Мартинкорена И., Тубио Дж.М., Ли И., Мензис А., Муди Л., Рамакришна М., Йейтс Л., Дэвис Х., Болли Н., Бигнелл Г.Р., Тарпи П.С., Беджати С., Ник-Зайнал С., Папаэммануил Э., Тейшейра В.Х., Рейн К., О’Мира С., Додоран М.С., Тиг Дж.В., Батлер А.П., Якобуцио-Донахью С., Сантариус Т., Гранди Р.Г., Малкин Д. , Гривз М., Мунши Н., Фланаган А.М., Боутелл Д., Мартин С., Ларсимонт Д., Рейс-Фильо Дж.С., Буссиутас А., Тейлор Дж.А., Хейс Н.Д., Джейнс С.М., Футреал П.А., Стрэттон М.Р., Макдермотт У., Кэмпбелл П.Дж. Процессированные псевдогены, приобретенные соматически при развитии рака. Нац коммун. 2014;5:3644. [Бесплатная статья PMC] [PubMed] [Google Scholar]
170. Wimmer K, Callens T, Wernstedt A, Messiaen L. Ген NF1 содержит горячие точки для зависимой от эндонуклеазы L1 вставки de novo. Генетика PLoS. 2011;7:e1002371. [Бесплатная статья PMC] [PubMed] [Google Scholar]
171. Kingsmore SF, Giros B, Suh D, Bieniarz M, Caron MG, Seldin MF. Мутация гена бета-субъединицы рецептора глицина у спастической мыши, связанная со вставкой элемента LINE-1. Нат Жене. 1994; 7: 136–141. [PubMed] [Google Scholar]
Нат Жене. 1994; 7: 136–141. [PubMed] [Google Scholar]
172. Mulhardt C, Fischer M, Gass P, Simon-Chazottes D, Guenet JL, Kuhse J, Betz H, Becker CM. Спастическая мышь: аберрантный сплайсинг мРНК бета-субъединицы рецептора глицина, вызванный интронной вставкой элемента L1. Нейрон. 1994;13:1003–1015. [PubMed] [Google Scholar]
173. Такахара Т., Осуми Т., Куромицу Дж., Сибата К., Сасаки Н., Оказаки Ю., Сибата Х., Сато С., Йошики А., Кусакабе М., Мурамацу М., Уэки М., Окуда К., Hayashizaki Y. Дисфункция орлеанского рилера, возникающая из-за пропуска экзона из-за транспозиции полноразмерной копии активной последовательности L1 в пропущенный экзон. Хум Мол Жене. 1996; 5: 989–993. [PubMed] [Google Scholar]
174. Кодзима Т., Накадзима К., Микошиба К. Неработающий ген 1 нарушен заменой фрагментом L1 у мышей йотари. Мозг Res Мол Мозг Res. 2000; 75: 121–127. [PubMed] [Академия Google]
175. Perou CM, Pryor RJ, Naas TP, Kaplan J. Мутация аллеля bg происходит из-за ретротранспозиции элемента LINE1. Геномика. 1997; 42: 366–368. [PubMed] [Google Scholar]
Геномика. 1997; 42: 366–368. [PubMed] [Google Scholar]
176. Yajima I, Sato S, Kimura T, Yasumoto K, Shibahara S, Goding CR, Yamamoto H. Интронная вставка элемента L1 в черноглазый белый (Mitf[mi-bw] ) ген: потеря единственной изоформы Mitf, ответственная за пигментный дефект и глухоту внутреннего уха. Хум Мол Жене. 1999; 8: 1431–1441. [PubMed] [Академия Google]
177. Корман Д.С., Харрис Дж.Б., Мейслер М.Х. Обнаружение мутаций в аллелях med и medJ натриевого канала Scn8a. Необычный сплайсинг из-за минорного интрона класса AT-AC. Дж. Биол. Хим. 1996; 271:17576–17581. [PubMed] [Google Scholar]
178. Bomar JM, Benke PJ, Slattery EL, Puttagunta R, Taylor LP, Seong E, Nystuen A, Chen W, Albin RL, Patel PD, Kittles RA, Sheffield VC, Burmeister M. Мутации в новом гене, кодирующем домен CRAL-TRIO, вызывают атаксию Каймановых островов у человека и атаксию/дистонию у нервных мышей. Нат Жене. 2003; 35: 264–269.. [PubMed] [Google Scholar]
179. Gilbert N, Bomar JM, Burmeister M, Moran JV. Характеристика мутагенной вставки ретротранспозона B1 у нервных мышей. Хум Мутат. 2004; 24:9–13. [PubMed] [Google Scholar]
Характеристика мутагенной вставки ретротранспозона B1 у нервных мышей. Хум Мутат. 2004; 24:9–13. [PubMed] [Google Scholar]
180. Katzir N, Rechavi G, Cohen JB, Unger T, Simoni F, Segal S, Cohen D, Givol D. Вставка «ретропозон» в клеточный онкоген c-myc при трансмиссивных венерических заболеваниях собак. опухоль. Proc Natl Acad Sci U S A. 1985; 82:1054–1058. [Бесплатная статья PMC] [PubMed] [Google Scholar]
181. Choi Y, Ishiguro N, Shinagawa M, Kim CJ, Okamoto Y, Minami S, Ogihara K. Молекулярная структура собачьих элементов LINE-1 в собачьей трансмиссивной венерической опухоли. Аним Жене. 1999; 30:51–53. [PubMed] [Google Scholar]
182. Брукс М.Б., Гу В., Барнас Дж.Л., Рэй Дж., Рэй К. Вставка линии 1 в гене фактора IX приводит к сегрегации легкой формы гемофилии В у собак. Геном Мамм. 2003; 14: 788–795. [PubMed] [Google Scholar]
183. Smith BF, Yue Y, Woods PR, Kornegay JN, Shin JH, Williams RR, Duan D. Вставка интронного элемента LINE-1 в ген дистрофина прерывает экспрессию дистрофина и приводит к болезни Дюшенна. -подобная мышечная дистрофия у породы корги. Лаборатория Инвест. 2011;91: 216–231. [Бесплатная статья PMC] [PubMed] [Google Scholar]
-подобная мышечная дистрофия у породы корги. Лаборатория Инвест. 2011;91: 216–231. [Бесплатная статья PMC] [PubMed] [Google Scholar]
184. Wolf ZT, Leslie EJ, Arzi B, Jayashankar K, Karmi N, Jia Z, Rowland DJ, Young A, Safra N, Sliskovic S, Murray JC, Wade СМ, Баннаш ДЛ. Вставка LINE-1 в DLX6 отвечает за волчью пасть и аномалии нижней челюсти в собачьей модели последовательности Пьера Робина. Генетика PLoS. 2014;10:e1004257. [Бесплатная статья PMC] [PubMed] [Google Scholar]
185. Даунс Л.М., Меллерш С.С. Вставка Intronic SINE в FAM161A, вызывающая пропуск экзонов, связана с прогрессирующей атрофией сетчатки у тибетских спаниелей и тибетских терьеров. ПЛОС Один. 2014;9:e93990. [Бесплатная статья PMC] [PubMed] [Google Scholar]
186. Clark LA, Wahl JM, Rees CA, Murphy KE. Вставка ретротранспозона в SILV отвечает за паттерн мерле у домашней собаки. Proc Natl Acad Sci U S A. 2006;103:1376–1381. [Бесплатная статья PMC] [PubMed] [Google Scholar]
187. Lin L, Faraco J, Li R, Kadotani H, Rogers W, Lin X, Qiu X, de Jong PJ, Nishino S, Mignot E. Расстройство сна Нарколепсия собак вызывается мутацией гена рецептора гипокретина (орексина) 2. Клетка. 1999;98:365–376. [PubMed] [Google Scholar]
Расстройство сна Нарколепсия собак вызывается мутацией гена рецептора гипокретина (орексина) 2. Клетка. 1999;98:365–376. [PubMed] [Google Scholar]
188. Пеле М., Тирет Л., Кесслер Дж.Л., Блот С., Пантье Дж.Дж. Экзоническая вставка SINE в гене PTPLA приводит к множественным дефектам сплайсинга и сегрегации с аутосомно-рецессивной центронуклеарной миопатией у собак. Хум Мол Жене. 2005; 14:1417–1427. [PubMed] [Google Scholar]
189. Parker HG, VonHoldt BM, Quignon P, Margulies EH, Shao S, Mosher DS, Spady TC, Elkahloun A, Cargill M, Jones PG, Maslen CL, Acland GM, Sutter NB, Куроки К., Бустаманте К.Д., Уэйн Р.К., Острандер Э.А. Экспрессированный ретроген fgf4 связан с определяющей породу хондродисплазией у домашних собак. Наука. 2009 г.;325:995–998. [Бесплатная статья PMC] [PubMed] [Google Scholar]
190. Heidmann T, Heidmann O, Nicolas JF. Ген-индикатор для демонстрации внутриклеточной транспозиции дефектных ретровирусов. Proc Natl Acad Sci U S A. 1988; 85:2219–2223. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
191. Curcio MJ, Garfinkel DJ. Одноэтапный отбор для ретротранспозиции элемента Ty1. Proc Natl Acad Sci USA. 1991; 88: 936–940. [Бесплатная статья PMC] [PubMed] [Google Scholar]
192. Wei W, Morrish TA, Alisch RS, Moran JV. Временный анализ показывает, что культивируемые клетки человека могут приспосабливаться к множеству событий ретротранспозиции LINE-1. Анальная биохимия. 2000; 284:435–438. [PubMed] [Академия Google]
193. Фриман Д.Д., Гудчайлд Н.Л., Магер Д.Л. Модифицированный индикаторный ген для отбора событий ретротранспозиции в клетках млекопитающих. Биотехнологии. 1994;17:46, 8–9, 52. [PubMed] [Google Scholar]
194. Xie Y, Rosser JM, Thompson TL, Boeke JD, An W. Характеристика ретротранспозиции L1 с помощью высокопроизводительных анализов двойной люциферазы . Нуклеиновые Кислоты Res. 2011;39:e16. [Бесплатная статья PMC] [PubMed] [Google Scholar]
195. Ostertag EM, Prak ET, DeBerardinis RJ, Moran JV, Kazazian HH. , Jr Определение кинетики ретротранспозиции L1 в культивируемых клетках. Нуклеиновые Кислоты Res. 2000; 28:1418–1423. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Jr Определение кинетики ретротранспозиции L1 в культивируемых клетках. Нуклеиновые Кислоты Res. 2000; 28:1418–1423. [Бесплатная статья PMC] [PubMed] [Google Scholar]
196. Морриш Т.А., Гилберт Н., Майерс Дж.С., Винсент Б.Дж., Стамато Т.Д., Такчоли Г.Е., Батцер М.А., Моран Дж.В. Репарация ДНК, опосредованная эндонуклеазо-независимой ретротранспозицией LINE-1. Нат Жене. 2002; 31: 159–165. [PubMed] [Google Scholar]
197. Goodier JL, Zhang L, Vetter MR, Kazazian HH., Jr Белок LINE-1 ORF1 локализуется в стрессовых гранулах с другими РНК-связывающими белками, включая компоненты РНК-интерференции РНК-индуцированное молчание сложный. Мол Селл Биол. 2007; 27:6469–6483. [Бесплатная статья PMC] [PubMed] [Google Scholar]
198. Кулпа Д.А., Моран СП. Образование частиц рибонуклеопротеина необходимо, но недостаточно для ретротранспозиции LINE-1. Хум Мол Жене. 2005; 14:3237–3248. [PubMed] [Google Scholar]
199. Кулпа Д.А., Моран Дж.В. Цис-предпочтительная активность обратной транскриптазы LINE-1 в частицах рибонуклеопротеина. Nat Struct Mol Biol. 2006; 13: 655–660. [PubMed] [Google Scholar]
Nat Struct Mol Biol. 2006; 13: 655–660. [PubMed] [Google Scholar]
200. Taylor MS, Lacava J, Mita P, Molloy KR, Huang CR, Li D, Adney EM, Jiang H, Burns KH, Chait BT, Rout MP, Boeke JD, Dai L. Протеомика сродства выявляет факторы человека-хозяина, участвующие в дискретных стадиях ретротранспозиции LINE-1. Клетка. 2013; 155:1034–1048. [Бесплатная статья PMC] [PubMed] [Google Scholar]
201. Goodier JL, Cheung LE, Kazazian HH., Jr Картирование интерактома белка ORF1 LINE1 выявило ассоциированные ингибиторы ретротранспозиции человека. Нуклеиновые Кислоты Res. 2013;41:7401–7419. [Бесплатная статья PMC] [PubMed] [Google Scholar]
202. Gilbert N, Lutz-Prigge S, Moran JV. Геномные делеции, созданные при ретротранспозиции LINE-1. Клетка. 2002; 110:315–325. [PubMed] [Google Scholar]
203. Symer DE, Connelly C, Szak ST, Caputo EM, Cost GJ, Parmigiani G, Boeke JD. Ретротранспозиция l1 человека связана с генетической нестабильностью in vivo. Клетка. 2002; 110:327–338. [PubMed] [Академия Google]
[PubMed] [Академия Google]
204. Kajikawa M, Okada N. LINEs мобилизуют SINE у угря через общую 3′-последовательность. Клетка. 2002; 111:433–444. [PubMed] [Google Scholar]
205. Сугано Т., Кадзикава М., Окада Н. Выделение и характеристика ретротранспозиционно-компетентных LINE от рыбок данио. Ген. 2006; 365:74–82. [PubMed] [Google Scholar]
206. Seleme MC, Vetter MR, Cordaux R, Bastone L, Batzer MA, Kazazian HH., Jr Обширные индивидуальные вариации способности к ретротранспозиции L1 способствуют генетическому разнообразию человека. Proc Natl Acad Sci U S A. 2006;103:6611–6616. [Бесплатная статья PMC] [PubMed] [Google Scholar]
207. Беккер К.Г., Свергольд Г.Д., Озато К., Тайер Р.Э. Связывание вездесущего ядерного фактора транскрипции YY1 с цис-регуляторной последовательностью в мобильном элементе LINE-1 человека. Хум Мол Жене. 1993; 2: 1697–1702. [PubMed] [Google Scholar]
208. Атаникар Дж. Н., Бэдж Р. М., Моран Дж. В. Сайт связывания YY1 необходим для точной инициации транскрипции LINE-1 человека. Нуклеиновые Кислоты Res. 2004; 32:3846–3855. [PMC free article] [PubMed] [Google Scholar]
Нуклеиновые Кислоты Res. 2004; 32:3846–3855. [PMC free article] [PubMed] [Google Scholar]
209. Дмитриев С.Е., Андреев Д.Е., Теренин И.М., Оловников И.А., Прасолов В.С., Меррик В.К., Шацкий И.Н. Инициация эффективного перевода под руководством 95′-нетранслируемая область человеческого ретротранспозона LINE-1 длиной 000 нуклеотидов и богатая GC мРНК является строго зависимой от кэпа, а не опосредованной внутренним сайтом посадки рибосомы. Мол Селл Биол. 2007; 27: 4685–4697. [Бесплатная статья PMC] [PubMed] [Google Scholar]
210. Ченио Т., Казелла Дж. Ф., Хайдманн Т. Члены семейства SRY регулируют ретротранспозоны LINE человека. Нуклеиновые Кислоты Res. 2000; 28: 411–415. [Бесплатная статья PMC] [PubMed] [Google Scholar]
211. Yang N, Zhang L, Zhang Y, Kazazian HH., Jr Важная роль RUNX3 в транскрипции и ретротранспозиции L1 человека. Нуклеиновые Кислоты Res. 2003;31:4929–4940. [Бесплатная статья PMC] [PubMed] [Google Scholar]
212. Нигуманн П., Редик К., Матлик К. , Спик М. Многие гены человека транскрибируются с антисмыслового промотора ретротранспозона L1. Геномика. 2002; 79: 628–634. [PubMed] [Google Scholar]
, Спик М. Многие гены человека транскрибируются с антисмыслового промотора ретротранспозона L1. Геномика. 2002; 79: 628–634. [PubMed] [Google Scholar]
213. Macia A, Munoz-Lopez M, Cortes JL, Hastings RK, Morell S, Lucena-Aguilar G, Marchal JA, Badge RM, Garcia-Perez JL. Эпигенетический контроль экспрессии ретротранспозонов в эмбриональных стволовых клетках человека. Мол Селл Биол. 2011; 31:300–316. [Бесплатная статья PMC] [PubMed] [Google Scholar]
214. Uesaka M, Nishimura O, Go Y, Nakashima K, Agata K, Imamura T. Двунаправленные промоторы являются основным источником некодирующих РНК, связанных с активацией генов, у млекопитающих. Геномика BMC. 2014;15:35. [Бесплатная статья PMC] [PubMed] [Google Scholar]
215. DeBerardinis RJ, Kazazian HH., Jr Анализ промотора из растущего подсемейства мышиных ретротранспозонов. Геномика. 1999; 56: 317–323. [PubMed] [Google Scholar]
216. Severynse DM, Hutchison CA, 3rd, Edgell MH. Идентификация регуляторной активности транскрипции в мономерной последовательности 5′-А-типа мышиного ретропозона LINE-1. Геном Мамм. 1992;2:41–50. [PubMed] [Google Scholar]
Геном Мамм. 1992;2:41–50. [PubMed] [Google Scholar]
217. Земойтель Т., Пенцкофер Т., Шульц Дж., Дандекар Т., Бадж Р., Вингрон М. Экзонизация активных мышиных L1: движущая сила эволюции транскриптома? Геномика BMC. 2007; 8:392. [Бесплатная статья PMC] [PubMed] [Google Scholar]
218. Li J, Kannan M, Trivett AL, Liao H, Wu X, Akagi K, Symer DE. Антисмысловой промотор в открытой рамке считывания 1 ретротранспозона L1 мыши инициирует экспрессию разнообразных транскриптов слияния и ограничивает ретротранспозицию. Нуклеиновые Кислоты Res. 2014;42:4546–4562. [Бесплатная статья PMC] [PubMed] [Google Scholar]
219. Форт А., Хашимото К., Ямада Д., Салимулла М., Кейя К.А., Саксена А., Бонетти А., Войнеагу И., Бертин Н., Крац А., Норо Ю., Вонг Ч., де Хун М., Андерссон Р., Санделин А., Suzuki H, Wei CL, Koseki H, Hasegawa Y, Forrest AR, Carninci P. Глубокое профилирование транскриптома стволовых клеток млекопитающих подтверждает регулирующую роль ретротранспозонов в поддержании плюрипотентности. Нат Жене. 2014; 46: 558–566. [PubMed] [Google Scholar]
Нат Жене. 2014; 46: 558–566. [PubMed] [Google Scholar]
220. Kapusta A, Kronenberg Z, Lynch VJ, Zhuo X, Ramsay L, Bourque G, Yandell M, Feschotte C. Мобильные элементы вносят основной вклад в происхождение, диверсификацию и регуляцию позвоночных. длинные некодирующие РНК. Генетика PLoS. 2013;9:e1003470. [Бесплатная статья PMC] [PubMed] [Google Scholar]
221. Kapusta A, Feschotte C. Летучая эволюция длинных некодирующих репертуаров РНК: механизмы и биологические последствия. Тенденции Жене. 2014;30:439–452. [Бесплатная статья PMC] [PubMed] [Google Scholar]
222. Guttman M, Donaghey J, Carey BW, Garber M, Grenier JK, Munson G, Young G, Lucas AB, Ach R, Bruhn L, Yang X, Amit I, Мейснер А., Регев А., Ринн Дж. Л., Рут Д. Е., Ландер Э. С. lincRNAs действуют в схемах, контролирующих плюрипотентность и дифференцировку. Природа. 2011;477:295–300. [Бесплатная статья PMC] [PubMed] [Google Scholar]
223. Macia A, Blanco-Jimenez E, Garcia-Perez JL. Ретротранспозоны в плюрипотентных клетках: влияние и новые роли в клеточной пластичности. Биохим Биофиз Акта. 2014 [Epub перед печатью]. [PubMed] [Google Scholar]
Биохим Биофиз Акта. 2014 [Epub перед печатью]. [PubMed] [Google Scholar]
224. Leibold DM, Swergold GD, Singer MF, Thayer RE, Dombroski BA, Fanning TG. Трансляция элементов ДНК LINE-1 in vitro и в клетках человека. Proc Natl Acad Sci U S A. 1990; 87:6990–6994. [Бесплатная статья PMC] [PubMed] [Google Scholar]
225. Колоша В.О., Мартин С.Л. Высокоаффинное, не специфичное к последовательности связывание РНК белком с открытой рамкой считывания 1 (ORF1) из длинного вкрапленного ядерного элемента 1 (LINE-1) J Biol Chem. 2003; 278:8112–8117. [PubMed] [Google Scholar]
226. Колоша В.О., Мартин С.Л. Свойства первого белка ORF мыши LINE-1 in vitro подтверждают его роль в образовании рибонуклеопротеиновых частиц во время ретротранспозиции. Proc Natl Acad Sci U S A. 1997; 94:10155–10160. [Бесплатная статья PMC] [PubMed] [Google Scholar]
227. Мартин С.Л. Частицы рибонуклеопротеина с РНК LINE-1 в клетках эмбриональной карциномы мыши. Мол Селл Биол. 1991; 11:4804–4807. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
228. Hohjoh H, Singer MF. Рибонуклеаза и высокая чувствительность к соли рибонуклеопротеинового комплекса, образованного ретротранспозоном LINE-1 человека. Дж Мол Биол. 1997; 271:7–12. [PubMed] [Google Scholar]
229. Martin SL, Branciforte D, Keller D, Bain DL. Тримерная структура незаменимого белка в ретротранспозиции L1. Proc Natl Acad Sci U S A. 2003;100:13815–13820. [Бесплатная статья PMC] [PubMed] [Google Scholar]
230. Basame S, Wai-lun Li P, Howard G, Branciforte D, Keller D, Martin SL. Пространственная сборка и стехиометрия связывания РНК белка LINE-1 необходимы для ретротранспозиции. Дж Мол Биол. 2006; 357: 351–357. [PubMed] [Google Scholar]
231. Хазина Е., Вайхенридер О. Ретротранспозоны не-LTR кодируют неканонические домены RRM в своей первой открытой рамке считывания. Proc Natl Acad Sci U S A. 2009;106:731–736. [Бесплатная статья PMC] [PubMed] [Google Scholar]
232. Каллахан К.Е., Хикман А. Б., Джонс К.Е., Гирландо Р., Фурано А.В. Полимеризация и свойства связывания нуклеиновых кислот белка ORF1 L1 человека. Нуклеиновые Кислоты Res. 2012;40:813–827. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Б., Джонс К.Е., Гирландо Р., Фурано А.В. Полимеризация и свойства связывания нуклеиновых кислот белка ORF1 L1 человека. Нуклеиновые Кислоты Res. 2012;40:813–827. [Бесплатная статья PMC] [PubMed] [Google Scholar]
233. Янушик К., Ли П.В., Вильярреал В., Брансифорте Д., Ву Х., Се И., Фейгон Дж., Лу Дж.А., Мартин С.Л., Clubb RT. Идентификация и решение структуры высококонсервативного С-концевого домена в ORF1p, необходимого для ретротранспозиции длинного вкрапленного ядерного элемента-1. Дж. Биол. Хим. 2007; 282:24893–24904. [PubMed] [Google Scholar]
234. Мартин С.Л. Свойства шаперона нуклеиновой кислоты ORF1p из не-LTR ретротранспозона, LINE-1. РНК биол. 2010;7:706–711. [Бесплатная статья PMC] [PubMed] [Google Scholar]
235. Мартин С.Л., Бушман Д., Ван Ф., Ли П.В., Уокер А., Каммиски Дж., Бранчифорте Д., Уильямс М.С. Замена одной аминокислоты в ORF1 резко уменьшает ретротранспозицию L1 и дает представление об активности шаперонов нуклеиновых кислот. Нуклеиновые Кислоты Res. 2008; 36: 5845–5854. [Бесплатная статья PMC] [PubMed] [Google Scholar]
2008; 36: 5845–5854. [Бесплатная статья PMC] [PubMed] [Google Scholar]
236. Evans JD, Peddigari S, Chaurasiya KR, Williams MC, Martin SL. Парные мутации отменяют и восстанавливают сбалансированные активности отжига и плавления ORF1p, необходимые для ретротранспозиции LINE-1. Нуклеиновые Кислоты Res. 2011;39: 5611–5621. [Бесплатная статья PMC] [PubMed] [Google Scholar]
237. Heras SR, Lopez MC, Garcia-Perez JL, Martin SL, Thomas MC. С-концевой домен L1Tc из ретротранспозона с недлинными концевыми повторами Trypanosoma cruzi кодирует белок, который несет два мотива цинковых пальцев C2h3 и обладает активностью шаперона нуклеиновой кислоты. Мол Селл Биол. 2005; 25:9209–9220. [Бесплатная статья PMC] [PubMed] [Google Scholar]
238. Доусон А., Хартсвуд Э., Патерсон Т., Финнеган Д.Дж. LINE-подобный подвижный элемент у дрозофилы, фактор I, кодирует белок со свойствами, сходными со свойствами ретровирусных нуклеокапсидов. EMBO J. 1997;16:4448–4455. [Статья бесплатно PMC] [PubMed] [Google Scholar]
239. Накамура М., Окада Н., Кадзикава М. Самовзаимодействие, связывание нуклеиновых кислот и активность шаперонов нуклеиновых кислот неожиданно сохраняются в уникальной ORF1p рыбок данио LINE. Мол Селл Биол. 2012; 32: 458–469. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Накамура М., Окада Н., Кадзикава М. Самовзаимодействие, связывание нуклеиновых кислот и активность шаперонов нуклеиновых кислот неожиданно сохраняются в уникальной ORF1p рыбок данио LINE. Мол Селл Биол. 2012; 32: 458–469. [Бесплатная статья PMC] [PubMed] [Google Scholar]
240. Kajikawa M, Sugano T, Sakurai R, Okada N. Низкая зависимость ретротранспозиции от белка ORF1 рыбок данио LINE, ZfL2-1. Ген. 2012; 499:41–47. [PubMed] [Академия Google]
241. Goodier JL, Mandal PK, Zhang L, Kazazian HH., Jr Дискретное субклеточное разделение РНК ретротранспозонов человека, несмотря на общий механизм вставки в геном. Хум Мол Жене. 2010;19:1712–1725. [Бесплатная статья PMC] [PubMed] [Google Scholar]
242. Martin F, Maranon C, Olivares M, Alonso C, Lopez MC. Характеристика кДНК ретротранспозона с недлинным терминальным повтором (L1Tc) из Trypanosoma cruzi: гомология первой ORF с ферментами репарации ДНК семейства обезьян. Дж Мол Биол. 1995;247:49–59. [PubMed] [Google Scholar]
243. Mol CD, Kuo CF, Thayer MM, Cunningham RP, Tainer JA. Структура и функция многофункционального фермента репарации ДНК экзонуклеазы III. Природа. 1995; 374: 381–386. [PubMed] [Google Scholar]
Mol CD, Kuo CF, Thayer MM, Cunningham RP, Tainer JA. Структура и функция многофункционального фермента репарации ДНК экзонуклеазы III. Природа. 1995; 374: 381–386. [PubMed] [Google Scholar]
244. Вайхенридер О., Репанас К., Перракис А. Кристаллическая структура нацеливающей эндонуклеазы ретротранспозона LINE-1 человека. Структура (Кэмб) 2004; 12: 975–986. [PubMed] [Google Scholar]
245. Стоимость GJ, Boeke JD. Нацеливание на интеграцию ретротранспозонов человека обусловлено специфичностью эндонуклеазы L1 к областям необычной структуры ДНК. Биохимия. 1998;37:18081–18093. [PubMed] [Google Scholar]
246. Стоимость GJ, Golding A, Schlissel MS, Boeke JD. Хроматинизация ДНК-мишени модулирует разрыв эндонуклеазой L1. Нуклеиновые Кислоты Res. 2001; 29: 573–577. [Бесплатная статья PMC] [PubMed] [Google Scholar]
247. Xiong Y, Eickbush TH. Происхождение и эволюция ретроэлементов на основе их последовательностей обратной транскриптазы. EMBO J. 1990; 9: 3353–3362. [Бесплатная статья PMC] [PubMed] [Google Scholar]
248. Malik HS, Burke WD, Eickbush TH. Возраст и эволюция ретромобильных элементов, не являющихся LTR. Мол Биол Эвол. 1999;16:793–805. [PubMed] [Google Scholar]
Malik HS, Burke WD, Eickbush TH. Возраст и эволюция ретромобильных элементов, не являющихся LTR. Мол Биол Эвол. 1999;16:793–805. [PubMed] [Google Scholar]
249. Евгеньев М.Б., Архипова И.Р. Пенелопоподобные элементы — новый класс ретроэлементов: распространение, функции и возможное эволюционное значение. Цитогенет Геном Res. 2005; 110: 510–521. [PubMed] [Google Scholar]
250. Домброски Б.А., Фэн К., Матиас С.Л., Сассаман Д.М., Скотт А.Ф., Казазян Х.Х., младший, Боке Д.Д. Анализ in vivo обратной транскриптазы ретротранспозона L1 человека в Saccharomyces cerevisiae. Мол Селл Биол. 1994; 14:4485–449.2. [PMC free article] [PubMed] [Google Scholar]
251. Пискарева О., Денмухаметова С., Шматченко В. Функциональная обратная транскриптаза, кодируемая LINE-1 человека из инфицированных бакуловирусом клеток насекомых. Protein Expr Purif. 2003; 28: 125–130. [PubMed] [Google Scholar]
252. Bibillo A, Eickbush TH. Высокая процессивность обратной транскриптазы из ретротранспозона с недлинным концевым повтором. Дж. Биол. Хим. 2002; 277:34836–34845. [PubMed] [Google Scholar]
Дж. Биол. Хим. 2002; 277:34836–34845. [PubMed] [Google Scholar]
253. Пискарева О., Шматченко В. Полимеризация ДНК обратной транскриптазой ретротранспозона L1 человека на собственной матрице in vitro. ФЭБС лат. 2006; 580: 661–668. [PubMed] [Академия Google]
254. Monot C., Kuciak M., Viollet S., Mir AA, Gabus C., Darlix JL, Cristofari G. Специфичность и гибкость праймирования обратной транскрипции l1 в несовершенных Т-трактах. Генетика PLoS. 2013;9:e1003499. [PMC free article] [PubMed] [Google Scholar]
255. Пискарева О., Эрнст С., Хиггинс Н., Шматченко В. Карбокси-концевой сегмент человеческого белка LINE-1 ORF2 участвует в связывании РНК. Открытая биография ФЭБС. 2013;3:433–437. [Бесплатная статья PMC] [PubMed] [Google Scholar]
256. Alisch RS, Garcia-Perez JL, Muotri AR, Gage FH, Moran JV. Нетрадиционная трансляция ретротранспозонов LINE-1 млекопитающих. Гены Дев. 2006; 20: 210–224. [Бесплатная статья PMC] [PubMed] [Google Scholar]
257. Li PW, Li J, Timmerman SL, Krushel LA, Martin SL. Дицистронная РНК из мышиного ретротранспозона LINE-1 содержит внутренний сайт посадки рибосомы выше каждой ORF: значение для ретротранспозиции. Нуклеиновые Кислоты Res. 2006; 34: 853–864. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Дицистронная РНК из мышиного ретротранспозона LINE-1 содержит внутренний сайт посадки рибосомы выше каждой ORF: значение для ретротранспозиции. Нуклеиновые Кислоты Res. 2006; 34: 853–864. [Бесплатная статья PMC] [PubMed] [Google Scholar]
258. An W, Dai L, Niewiadomska AM, Yetil A, O’Donnell KA, Han JS, Boeke JD. Характеристика синтетического человеческого ретротранспозона LINE-1 ORFeus-Hs. ДНК мафии. 2011;2:2. [Бесплатная статья PMC] [PubMed] [Google Scholar]
259. Хан Дж.С., Боке Дж.Д. Высокоактивный синтетический ретротранспозон млекопитающих. Природа. 2004; 429:314–318. [PubMed] [Google Scholar]
260. Инголия Н.Т., Гаеммагами С., Ньюман Дж. Р., Вайсман Дж. С. Полногеномный анализ трансляции in vivo с разрешением нуклеотидов с использованием профилирования рибосом. Наука. 2009; 324: 218–223. [Бесплатная статья PMC] [PubMed] [Google Scholar]
261. Славофф С.А., Митчелл А.Дж., Швайд А.Г., Кабили М.Н., Ма Дж., Левин Дж.З., Каргер А.Д., Будник Б.А., Ринн Дж. Л., Сагателян А. Пептидомное открытие коротких Пептиды, кодируемые открытой рамкой считывания, в клетках человека. Nat Chem Biol. 2013;9: 59–64. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Л., Сагателян А. Пептидомное открытие коротких Пептиды, кодируемые открытой рамкой считывания, в клетках человека. Nat Chem Biol. 2013;9: 59–64. [Бесплатная статья PMC] [PubMed] [Google Scholar]
262. Усдин К., Фурано А.В. Структура богатой гуанином последовательности полипурин: полипиримидин на правом конце крысиного элемента L1 (LINE). Дж. Биол. Хим. 1989; 264:15681–15687. [PubMed] [Google Scholar]
263. Moran JV, DeBerardinis RJ, Kazazian HH., Jr Перетасовка экзонов путем ретротранспозиции L1. Наука. 1999; 283:1530–1534. [PubMed] [Google Scholar]
264. Faulkner GJ, Kimura Y, Daub CO, Wani S, Plessy C, Irvine KM, Schroder K, Cloonan N, Steptoe AL, Lassmann T, Waki K, Hornig N, Arakawa T, Takahashi H, Kawai J, Forrest AR, Suzuki H, Hayashizaki Y, Hume DA, Orlando V, Grimmond SM, Carninci P. Регулируемый транскриптом ретротранспозонов клеток млекопитающих. Нат Жене. 2009 г.;41:563–571. [PubMed] [Google Scholar]
265. Луан Д.Д., Корман М.Х., Якубчак Д.Л. , Эйкбуш Т.Х. Обратная транскрипция РНК R2Bm инициируется разрывом в хромосомном сайте-мишени: механизм ретротранспозиции, отличной от LTR. Клетка. 1993; 72: 595–605. [PubMed] [Google Scholar]
, Эйкбуш Т.Х. Обратная транскрипция РНК R2Bm инициируется разрывом в хромосомном сайте-мишени: механизм ретротранспозиции, отличной от LTR. Клетка. 1993; 72: 595–605. [PubMed] [Google Scholar]
266. Mandal PK, Ewing AD, Hancks DC, Kazazian HH. Jr. Обогащение процессированных транскриптов псевдогенов в L1-рибонуклеопротеиновых частицах. Хум Мол Жене. 2013;22:3730–3748. [Бесплатная статья PMC] [PubMed] [Google Scholar]
267. Дутко Дж.А., Кенни А.Е., Гамаше Э.Р., Курсио М.Дж. Факторы распада мРНК с 5′ на 3′ локализуются совместно с Ty1 gag и человеческим APOBEC3G и способствуют ретротранспозиции Ty1. Дж Вирол. 2010; 84: 5052–5066. [PMC free article] [PubMed] [Google Scholar]
268. Ларсен Л.С., Белякова-Бетелл Н., Биланчоне В., Чжан М., Ламса А., Дасильва Р., Хэтфилд Г.В., Нагашима К., Сандмейер С. Нуклеокапсид Ty3 контролирует локализацию сборка частиц. Дж Вирол. 2008; 82: 2501–2514. [Бесплатная статья PMC] [PubMed] [Google Scholar]
269. Ларсен Л.С., Чжан М. , Белякова-Бетелл Н., Биланчоне В., Ламса А., Нагашима К., Нажди Р., Косака К., Ковачевич В., Ченг Дж., Балди П., Хэтфилд Г.В., Сандмейер С. Капсидные мутации Ty3 обнаруживают ранние и поздние функции амино-концевого домена. Дж Вирол. 2007; 81: 6957–6972. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Белякова-Бетелл Н., Биланчоне В., Ламса А., Нагашима К., Нажди Р., Косака К., Ковачевич В., Ченг Дж., Балди П., Хэтфилд Г.В., Сандмейер С. Капсидные мутации Ty3 обнаруживают ранние и поздние функции амино-концевого домена. Дж Вирол. 2007; 81: 6957–6972. [Бесплатная статья PMC] [PubMed] [Google Scholar]
270. Kubo S, Seleme MC, Soifer HS, Perez JL, Moran JV, Kazazian HH, Jr, Kasahara N. Ретротранспозиция L1 в неделящихся и первичных соматических клетках человека. Proc Natl Acad Sci U S A. 2006;103:8036–8041. [Бесплатная статья PMC] [PubMed] [Google Scholar]
271. Гудвин Т.Дж., Орманди Дж.Э., Поултер Р.Т. L1-подобные не-LTR ретротранспозоны дрожжей Candida albicans. Карр Жене. 2001; 39: 83–91. [PubMed] [Google Scholar]
272. Dong C, Poulter RT, Han JS. LINE-подобная ретротранспозиция у Saccharomyces cerevisiae. Генетика. 2009; 181:301–311. [Бесплатная статья PMC] [PubMed] [Google Scholar]
273. Kinsey JA. Транснуклеарная ретротранспозиция Tad элемента Neurospora. Proc Natl Acad Sci U S A. 1993;90:9384–9387. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Proc Natl Acad Sci U S A. 1993;90:9384–9387. [Бесплатная статья PMC] [PubMed] [Google Scholar]
274. Xie Y, Mates L, Ivics Z, Izsvak Z, Martin SL, An W. Деление клеток способствует эффективной ретротранспозиции в стабильной репортерной клеточной линии L1. ДНК мафии. 2013;4:10. [Бесплатная статья PMC] [PubMed] [Google Scholar]
275. Ши X, Селуанов А, Горбунова В. Для ретротранспозиции L1 необходимы клеточные деления. Мол Селл Биол. 2007; 27: 1264–1270. [Бесплатная статья PMC] [PubMed] [Google Scholar]
276. Стоимость GJ, Feng Q, Jacquier A, Boeke JD. Обратная транскрипция in vitro, нацеленная на элемент L1 человека. ЕМБО Дж. 2002; 21:5899–5910. [Бесплатная статья PMC] [PubMed] [Google Scholar]
277. Christensen SM, Eickbush TH. Обратная транскрипция, ориентированная на мишень R2: упорядоченные этапы расщепления и полимеризации белковыми субъединицами, асимметрично связанными с ДНК-мишенью. Мол Селл Биол. 2005; 25:6617–6628. [Бесплатная статья PMC] [PubMed] [Google Scholar]
278. Ostertag EM, Kazazian HH., Jr Двойное праймирование: предполагаемый механизм создания инверсий в ретротранспозиции L1. Геном Res. 2001; 11:2059–2065. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Ostertag EM, Kazazian HH., Jr Двойное праймирование: предполагаемый механизм создания инверсий в ретротранспозиции L1. Геном Res. 2001; 11:2059–2065. [Бесплатная статья PMC] [PubMed] [Google Scholar]
279. Zingler N, Willhoeft U, Brose HP, Schoder V, Jahns T, Hanschmann KM, Morrish TA, Lower J, Schumann GG. Анализ 5′-соединений ретротранспозонов LINE-1 и Alu человека предлагает альтернативную модель прикрепления 5′-концов, требующую соединения концов, опосредованного микрогомологией. Геном Res. 2005; 15: 780–789. [Бесплатная статья PMC] [PubMed] [Google Scholar]
280. Gasior SL, Wakeman TP, Xu B, Deininger PL. Ретротранспозон человека LINE-1 создает двухцепочечные разрывы ДНК. Дж Мол Биол. 2006; 357: 1383–139.3. [Бесплатная статья PMC] [PubMed] [Google Scholar]
281. Gasior SL, Roy-Engel AM, Deininger PL. ERCC1/XPF ограничивает ретротранспозицию L1. Восстановление ДНК (Амст) 2008; 7: 983–989. [Бесплатная статья PMC] [PubMed] [Google Scholar]
282. Coufal NG, Garcia-Perez JL, Peng GE, Marchetto MC, Muotri AR, Mu Y, Carson CT, Macia A, Moran JV, Gage FH. Мутированная атаксия телеангиэктазии (ATM) модулирует ретротранспозицию длинного вкрапленного элемента-1 (L1) в нервных стволовых клетках человека. Proc Natl Acad Sci U S A. 2011;108:20382–20387. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Мутированная атаксия телеангиэктазии (ATM) модулирует ретротранспозицию длинного вкрапленного элемента-1 (L1) в нервных стволовых клетках человека. Proc Natl Acad Sci U S A. 2011;108:20382–20387. [Бесплатная статья PMC] [PubMed] [Google Scholar]
283. Boboila C, Alt FW, Schwer B. Классические и альтернативные пути соединения концов для восстановления специфических для лимфоцитов и общих двухцепочечных разрывов ДНК. Ад Иммунол. 2012; 116:1–49. [PubMed] [Google Scholar]
284. Lavie L, Maldener E, Brouha B, Meese EU, Mayer J. Человеческий промотор L1: вариабельные сайты инициации транскрипции и основное влияние вышестоящей фланкирующей последовательности на активность промотора. Геном Res. 2004; 14: 2253–2260. [Бесплатная статья PMC] [PubMed] [Google Scholar]
285. Бабушок Д.В., Остертаг Э.М., Кортни С.Е., Чой Дж.М., Казазян Х.Х., мл. Интеграция L1 в модели трансгенных мышей. Геном Res. 2006; 16: 240–250. [Бесплатная статья PMC] [PubMed] [Google Scholar]
286. Итиянаги К., Накадзима Р., Кадзикава М., Окада Н. Новый анализ ретротранспозонов выявляет множественные пути мобильности, определяемые хозяевами. Геном Res. 2007; 17:33–41. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Итиянаги К., Накадзима Р., Кадзикава М., Окада Н. Новый анализ ретротранспозонов выявляет множественные пути мобильности, определяемые хозяевами. Геном Res. 2007; 17:33–41. [Бесплатная статья PMC] [PubMed] [Google Scholar]
287. Чен Дж., Раттнер А., Натанс Дж. Эффекты вставки ретротранспозона L1 на процессинг, локализацию и накопление транскриптов: уроки дегенерации сетчатки 7 мышей и последствия для геномных экология элементов L1. Хум Мол Жене. 2006;15:2146–2156. [PubMed] [Академия Google]
288. Goodier JL, Ostertag EM, Kazazian HH., Jr Трансдукция 3′-фланкирующих последовательностей является обычным явлением при ретротранспозиции L1. Хум Мол Жене. 2000; 9: 653–657. [PubMed] [Google Scholar]
289. Pickeral OK, Makalowski W, Boguski MS, Boeke JD. Частая трансдукция геномной ДНК человека, вызванная ретротранспозицией LINE-1. Геном Res. 2000; 10: 411–415. [Статья бесплатно PMC] [PubMed] [Google Scholar]
290. Holmes SE, Dombroski BA, Krebs CM, Boehm CD, Kazazian HH. , Jr Новый ретротранспонируемый элемент L1 человека из локуса LRE2 на хромосоме 1q производит химерную вставку . Нат Жене. 1994;7:143–148. [PubMed] [Google Scholar]
, Jr Новый ретротранспонируемый элемент L1 человека из локуса LRE2 на хромосоме 1q производит химерную вставку . Нат Жене. 1994;7:143–148. [PubMed] [Google Scholar]
291. Moran JV. Ретротранспозиция L1 человека: выводы и особенности, извлеченные из анализа ретротранспозиции культивируемых клеток. Генетика. 1999; 107:39–51. [PubMed] [Google Scholar]
292. Moran JV, Gilbert N. Ретротранспозоны LINE-1 млекопитающих и родственные элементы. В: Craig NL, Craigie R, Gellert M, Lambowitz AM, редакторы. Мобильная ДНК II. Вашингтон, округ Колумбия: ASM Press; 2002. стр. 836–869. [Google Scholar]
293. Эйкбуш Т. Перетасовка экзонов в ретроспективе. Наука. 1999;283:1465, 7. [PubMed] [Google Scholar]
294. Macfarlane CM, Collier P, Rahbari R, Beck CR, Wagstaff JF, Igoe S, Moran JV, Badge RM. ATLAS, специфичный для трансдукции, выявил когорту высокоактивных ретротранспозонов L1 в популяциях человека. Хум Мутат. 2013; 34: 974–985. [Бесплатная статья PMC] [PubMed] [Google Scholar]
295. Solyom S, Ewing AD, Hancks DC, Takeshima Y, Awano H, Matsuo M, Kazazian HH., Jr Патогенная орфанная трансдукция, созданная нереференсным ретротранспозоном LINE-1 . Хум Мутат. 2012;33:369–371. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Solyom S, Ewing AD, Hancks DC, Takeshima Y, Awano H, Matsuo M, Kazazian HH., Jr Патогенная орфанная трансдукция, созданная нереференсным ретротранспозоном LINE-1 . Хум Мутат. 2012;33:369–371. [Бесплатная статья PMC] [PubMed] [Google Scholar]
296. Tubio JM, Li Y, Ju YS, Martincorena I, Cooke SL, Tojo M, Gundem G, Pipinikas CP, Zamora J, Raine K, Menzies A, Roman -Гарсия П., Фуллам А., Герстунг М., Шлиен А., Тарпи П.С., Папэммануил Э., Кнаппског С., Ван Лу П., Рамакришна М., Дэвис Х.Р., Маршалл Дж., Ведж Д.С., Тиг Д.В., Батлер А.П., Ник-Зайнал С., Александров Л., Беджати С., Йейтс Л.Р., Болли Н., Муди Л., Харди С., Мартин С., Макларен С., О’Мира С., Андерсон Э., Мэддисон М., Гэмбл С., Фостер С., Уоррен Э.И., Уитакер Х., Брюэр Д., Илз Р., Купер С., Нил Д., Линч А.Г., Висакорпи Т., Исаакс В.Б., Вант Веер Л., Калдас С., Десмедт С., Сотириу С., Апарисио С., Фокенс Дж.А., Эйфьорд Дж.Е., Лахани С.Р., Томас Дж., Миклебост О., Span PN, Borresen-Dale AL, Richardson AL, Van de Vijver M, Vincent-Salomon A, Van den Eynden GG, Flanagan AM, Futreal PA, Janes SM, Bova GS, Stratton MR, McDermott U, Campbell PJ. Мобильная ДНК при раке. Обширная трансдукция неповторяющейся ДНК, опосредованная ретротранспозицией L1 в геномах рака. Наука. 2014;345:1251343. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Мобильная ДНК при раке. Обширная трансдукция неповторяющейся ДНК, опосредованная ретротранспозицией L1 в геномах рака. Наука. 2014;345:1251343. [Бесплатная статья PMC] [PubMed] [Google Scholar]
297. Hancks DC, Ewing AD, Chen JE, Tokunaga K, Kazazian HH., Jr Улавливание экзонов, опосредованное человеческим ретротранспозоном SVA. Геном Res. 2009; 19:1983–1991. [Бесплатная статья PMC] [PubMed] [Google Scholar]
298. Xing J, Wang H, Belancio VP, Cordaux R, Deininger PL, Batzer MA. Возникновение генов приматов путем трансдукции последовательности, опосредованной ретротранспозонами. Proc Natl Acad Sci U S A. 2006;103:17608–17613. [Бесплатная статья PMC] [PubMed] [Google Scholar]
299. Damert A, Raiz J, Horn AV, Lower J, Wang H, Xing J, Batzer MA, Lower R, Schumann GG. 5′-Трансдуцирующие группы ретротранспозонов SVA эффективно распространяются по всему геному человека. Геном Res. 2009 г.;19:1992–2008. [Бесплатная статья PMC] [PubMed] [Google Scholar]
300. Mine M, Chen JM, Brivet M, Desguerre I, Marchant D, de Lonlay P, Bernard A, Ferec C, Abitbol M, Ricquier D, Marsac C. Большая геномная делеция в гене PDHX, вызванная ретротранспозиционной вставкой полноразмерного элемента LINE-1. Хум Мутат. 2007; 28: 137–142. [PubMed] [Google Scholar]
Большая геномная делеция в гене PDHX, вызванная ретротранспозиционной вставкой полноразмерного элемента LINE-1. Хум Мутат. 2007; 28: 137–142. [PubMed] [Google Scholar]
301. Chou HH, Hayakawa T, Diaz S, Krings M, Indriati E, Leakey M, Paabo S, Satta Y, Takahata N, Varki A. Инактивация гидроксилазы CMP-N-ацетилнейраминовой кислоты произошло до расширения мозга в ходе эволюции человека. Proc Natl Acad Sci U S A. 2002;99:11736–11741. [Бесплатная статья PMC] [PubMed] [Google Scholar]
302. Такасу М., Хаяши Р., Маруя Э., Ота М., Имура К., Коуго К., Кобаяши С., Саджи Х., Исикава Ю., Асаи Т., Токунага К. Удаление полного гена HLA-A со вставкой ретротранспозона. Тканевые антигены. 2007; 70: 144–150. [PubMed] [Google Scholar]
303. Хан К., Сен С.К., Ван Дж., Каллинан П.А., Ли Дж., Кордо Р., Лян П., Батцер М.А. Геномные перестройки с помощью делеции, опосредованной вставкой LINE-1, в линиях человека и шимпанзе. Нуклеиновые Кислоты Res. 2005; 33: 4040–4052. [Бесплатная статья PMC] [PubMed] [Google Scholar]
304. Каллинан П.А., Ван Дж., Херке С.В., Гарбер Р.К., Лян П., Батцер М.А. Делеция, опосредованная ретротранспозицией Alu. Дж Мол Биол. 2005; 348: 791–800. [PubMed] [Google Scholar]
Каллинан П.А., Ван Дж., Херке С.В., Гарбер Р.К., Лян П., Батцер М.А. Делеция, опосредованная ретротранспозицией Alu. Дж Мол Биол. 2005; 348: 791–800. [PubMed] [Google Scholar]
305. Chen JM, Cooper DN, Ferec C, Kehrer-Sawatzki H, Patrinos GP. Геномные перестройки при наследственных заболеваниях и раке. Семин Рак Биол. 2010;20:222–233. [PubMed] [Google Scholar]
306. Ade C, Roy-Engel AM, Deininger PL. Элементы Alu: внутренний источник нестабильности генома человека. Карр Опин Вирол. 2013;3:639–645. [Статья бесплатно PMC] [PubMed] [Google Scholar]
307. Deininger PL, Moran JV, Batzer MA, Kazazian HH., Jr Мобильные элементы и эволюция генома млекопитающих. Curr Opin Genet Dev. 2003; 13: 651–658. [PubMed] [Google Scholar]
308. Eickbush TH. Репарация ретротранспозицией. Нат Жене. 2002; 31: 126–127. [PubMed] [Google Scholar]
309. Morrish TA, Garcia-Perez JL, Stamato TD, Taccioli GE, Sekiguchi J, Moran JV. Эндонуклеазо-независимая ретротранспозиция LINE-1 на теломерах млекопитающих. Природа. 2007; 446: 208–212. [PubMed] [Академия Google]
Природа. 2007; 446: 208–212. [PubMed] [Академия Google]
310. Копера Х.К., Молдован Дж.Б., Морриш Т.А., Гарсия-Перес Дж.Л., Моран Дж.В. Сходство между обратной транскриптазой длинного вкрапленного элемента-1 (LINE-1) и теломеразой. Proc Natl Acad Sci U S A. 2011;108:20345–20350. [Бесплатная статья PMC] [PubMed] [Google Scholar]
311. Belfort M, Curcio MJ, Lue NF. Теломераза и ретротранспозоны: обратные транскриптазы, сформировавшие геномы. Proc Natl Acad Sci U S A. 2011;108:20304–20310. [Бесплатная статья PMC] [PubMed] [Google Scholar]
312. Eickbush TH. Теломераза и ретротранспозоны: что появилось раньше? Наука. 1997;277:911–912. [PubMed] [Google Scholar]
313. Левис Р.В., Ганесан Р., Хаутченс К., Толар Л.А., Шин FM. Транспозоны вместо теломерных повторов на теломерах дрозофилы. Клетка. 1993; 75: 1083–1093. [PubMed] [Google Scholar]
314. Biessmann H, Valgeirsdottir K, Lofsky A, Chin C, Ginther B, Levis RW, Pardue ML. HeT-A, мобильный элемент, специально участвующий в «заживлении» сломанных концов хромосом у Drosophila melanogaster. Мол Селл Биол. 1992; 12:3910–3918. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Мол Селл Биол. 1992; 12:3910–3918. [Бесплатная статья PMC] [PubMed] [Google Scholar]
315. Гладышев ЕА, Архипова ИР. Связанные с теломерами эндонуклеазо-дефицитные пенелопоподобные ретроэлементы у различных эукариот. Proc Natl Acad Sci U S A. 2007;104:9352–9357. [Бесплатная статья PMC] [PubMed] [Google Scholar]
316. Sen SK, Huang CT, Han K, Batzer MA. Эндонуклеазо-независимая вставка обеспечивает альтернативный путь ретротранспозиции L1 в геноме человека. Нуклеиновые Кислоты Res. 2007; 35:3741–3751. [Бесплатная статья PMC] [PubMed] [Google Scholar]
317. Morisada N, Rendtorff ND, Nozu K, Morishita T, Miyakawa T, Matsumoto T, Hisano S, Iijima K, Tranebjaerg L, Shirahata A, Matsuo M, Kusuhara K. Бранхио-ото-ренальный синдром, вызванный частичной делецией EYA1 из-за вставки LINE-1. Педиатр Нефрол. 2010; 25:1343–1348. [PubMed] [Академия Google]
318. Чжун Дж., Ламбовиц А.М. Подвижность интронов группы II с использованием зарождающихся цепей на вилках репликации ДНК для запуска обратной транскрипции. EMBO J. 2003; 22: 4555–4565. [Бесплатная статья PMC] [PubMed] [Google Scholar]
EMBO J. 2003; 22: 4555–4565. [Бесплатная статья PMC] [PubMed] [Google Scholar]
319. Yoder JA, Walsh CP, Bestor TH. Метилирование цитозина и экология внутригеномных паразитов. Тенденции Жене. 1997; 13: 335–340. [PubMed] [Google Scholar]
320. Bestor TH, Bourc’his D. Замалчивание транспозонов и формирование импринта в зародышевых клетках млекопитающих. Cold Spring Harb Symp Quant Biol. 2004;69: 381–387. [PubMed] [Google Scholar]
321. Ooi SK, O’Donnell AH, Bestor TH. Метилирование цитозина млекопитающих с первого взгляда. Дж. Клеточные науки. 2009; 122: 2787–2791. [Бесплатная статья PMC] [PubMed] [Google Scholar]
322. Bourc’his D, Bestor TH. Мейотическая катастрофа и реактивация ретротранспозонов в мужских половых клетках, лишенных Dnmt3L. Природа. 2004; 431:96–99. [PubMed] [Google Scholar]
323. Soper SF, van der Heijden GW, Hardiman TC, Goodheart M, Martin SL, de Boer P, Bortvin A. Водоворот мыши, компонент nuage, необходим для сперматогенеза и репрессии транспозонов. в мейозе. Ячейка Дев. 2008; 15: 285–29.7. [Бесплатная статья PMC] [PubMed] [Google Scholar]
в мейозе. Ячейка Дев. 2008; 15: 285–29.7. [Бесплатная статья PMC] [PubMed] [Google Scholar]
324. Branco MR, Ficz G, Reik W. Раскрытие роли 5-гидроксиметилцитозина в эпигеноме. Нат Рев Жене. 2012; 13:7–13. [PubMed] [Google Scholar]
325. Сиоми М.С., Сато К., Пезич Д., Аравин А.А. Малые РНК, взаимодействующие с PIWI: авангард защиты генома. Nat Rev Mol Cell Biol. 2011;12:246–258. [PubMed] [Google Scholar]
326. Аравин А.А., Хэннон Г.Дж., Бреннеке Дж. Путь Piwi-piRNA обеспечивает адаптивную защиту в гонке вооружений транспозонов. Наука. 2007; 318: 761–764. [PubMed] [Академия Google]
327. Мэлоун К.Д., Бреннеке Дж., Дас М., Старк А., Маккомби В.Р., Сачиданандам Р., Хэннон Г.Дж. Специализированные пути piРНК действуют в зародышевой линии и соматических тканях яичника дрозофилы. Клетка. 2009; 137: 522–535. [Бесплатная статья PMC] [PubMed] [Google Scholar]
328. Brennecke J, Malone CD, Aravin AA, Sachidanandam R, Stark A, Hannon GJ. Эпигенетическая роль piРНК, унаследованных от матери, в молчании транспозонов. Наука. 2008; 322:1387–1392. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Наука. 2008; 322:1387–1392. [Бесплатная статья PMC] [PubMed] [Google Scholar]
329. Бреннеке Дж., Аравин А.А., Старк А., Дус М., Келлис М., Сачиданандам Р., Хэннон Г.Дж. Дискретные локусы, генерирующие малые РНК, как основные регуляторы активности транспозонов у дрозофилы. Клетка. 2007; 128:1089–1103. [PubMed] [Google Scholar]
330. Гунавардане Л.С., Сайто К., Нисида К.М., Миёси К., Кавамура Ю., Нагами Т., Сиоми Х., Сиоми М.С. Опосредованный слайсером механизм образования 5′-конца миРНК, ассоциированного с повторами, у дрозофилы. Наука. 2007; 315:1587–1590. [PubMed] [Google Scholar]
331. Ghildiyal M, Zamore PD. Малые молчащие РНК: расширяющаяся вселенная. Нат Рев Жене. 2009 г.;10:94–108. [Бесплатная статья PMC] [PubMed] [Google Scholar]
332. Malone CD, Hannon GJ. Малые РНК как хранители генома. Клетка. 2009; 136: 656–668. [Бесплатная статья PMC] [PubMed] [Google Scholar]
333. Аравин А.А., Сачиданандам Р., Буркхис Д., Шефер С., Пезич Д., Тот К. Ф., Бестор Т., Хэннон Г.Дж. Путь piRNA, запускаемый отдельными транспозонами, связан с метилированием ДНК de novo у мышей. Мол Ячейка. 2008; 31: 785–799. [Статья бесплатно PMC] [PubMed] [Google Scholar]
Ф., Бестор Т., Хэннон Г.Дж. Путь piRNA, запускаемый отдельными транспозонами, связан с метилированием ДНК de novo у мышей. Мол Ячейка. 2008; 31: 785–799. [Статья бесплатно PMC] [PubMed] [Google Scholar]
334. Аравин А.А., Буркис Д. Направляющие малых РНК для метилирования ДНК de novo в зародышевых клетках млекопитающих. Гены Дев. 2008;22:970–975. [Бесплатная статья PMC] [PubMed] [Google Scholar]
335. Кармелл М.А., Жирар А., ван де Кант Х.Дж., Буркхис Д., Бестор Т.Х., де Рой Д.Г., Хэннон Г.Дж. MIWI2 необходим для сперматогенеза и репрессии транспозонов в зародышевой линии самцов мышей. Ячейка Дев. 2007; 12: 503–514. [PubMed] [Google Scholar]
336. Курамочи-Миягава С., Ватанабе Т., Гото К., Тотоки Ю., Тойода А., Икава М., Асада Н., Кодзима К., Ямагути Ю., Идзири Т.В., Хата К., Ли Э., Мацуда Y, Kimura T, Okabe M, Sakaki Y, Sasaki H, Nakano T. Метилирование ДНК генов ретротранспозонов регулируется членами семейства Piwi MILI и MIWI2 в семенниках плода мышей. Гены Дев. 2008;22:908–917. [Бесплатная статья PMC] [PubMed] [Google Scholar]
2008;22:908–917. [Бесплатная статья PMC] [PubMed] [Google Scholar]
337. Рейтер М., Чума С., Танака Т., Франц Т., Старк А., Пиллаи Р.С. Потеря белка-1, содержащего взаимодействующий с Mili домен Tudor, активирует транспозоны и изменяет профиль малых РНК, ассоциированных с Mili. Nat Struct Mol Biol. 2009; 16: 639–646. [PubMed] [Google Scholar]
338. Yang N, Kazazian HH., Jr Ретротранспозиция L1 подавляется эндогенно кодируемыми малыми интерферирующими РНК в культивируемых клетках человека. Nat Struct Mol Biol. 2006; 13: 763–771. [PubMed] [Академия Google]
339. Эрас С.Р., Масиас С., Пласс М., Фернандес Н., Кано Д., Эйрас Э., Гарсия-Перес Х.Л., Касерес Х.Ф. Микропроцессор контролирует активность ретротранспозонов млекопитающих. Nat Struct Mol Biol. 2013;20:1173–1181. [Бесплатная статья PMC] [PubMed] [Google Scholar]
340. Ciaudo C, Jay F, Okamoto I, Chen CJ, Sarazin A, Servant N, Barillot E, Heard E, Voinnet O. РНК-зависимый и независимый контроль Накопление и подвижность LINE1 в эмбриональных стволовых клетках мышей. Генетика PLoS. 2013;9:e1003791. [Бесплатная статья PMC] [PubMed] [Google Scholar] Retracted
Генетика PLoS. 2013;9:e1003791. [Бесплатная статья PMC] [PubMed] [Google Scholar] Retracted
341. Faulkner GJ. Замалчивание ретротранспозонов во время эмбриогенеза: разрезы дайсера в LINE. Генетика PLoS. 2013;9:e1003944. [Статья бесплатно PMC] [PubMed] [Google Scholar]
342. Belancio VP, Hedges DJ, Deininger P. Сплайсинг РНК LINE-1 и влияние на экспрессию генов млекопитающих. Нуклеиновые Кислоты Res. 2006; 34: 1512–1521. [Бесплатная статья PMC] [PubMed] [Google Scholar]
343. Белансио В.П., Рой-Энгель А.М., Дейнингер П. Влияние множественных сайтов сплайсинга в элементах L1 человека. Ген. 2008; 411:38–45. [Бесплатная статья PMC] [PubMed] [Google Scholar]
344. Перепелица-Белансио В., Дейнингер П. Усечение РНК преждевременным полиаденилированием ослабляет активность мобильных элементов человека. Нат Жене. 2003; 35: 363–366. [PubMed] [Google Scholar]
345. Han JS, Szak ST, Boeke JD. Нарушение транскрипции ретротранспозоном L1 и последствия для транскриптомов млекопитающих. Природа. 2004; 429: 268–274. [PubMed] [Google Scholar]
Природа. 2004; 429: 268–274. [PubMed] [Google Scholar]
346. Peddigari S, Li PW, Rabe JL, Martin SL. hnRNPL и нуклеолин связывают РНК LINE-1 и функционируют как факторы-хозяева, модулируя ретротранспозицию. Нуклеиновые Кислоты Res. 2013;41:575–585. [Бесплатная статья PMC] [PubMed] [Google Scholar]
347. Дай Л., Тейлор М.С., О’Доннелл К.А., Боке Д.Д. Поли(А)-связывающий белок С1 необходим для эффективной ретротранспозиции L1 и влияет на образование L1 RNP. Мол Селл Биол. 2012; 32:4323–4336. [Бесплатная статья PMC] [PubMed] [Google Scholar]
348. Zhang A, Dong B, Doucet AJ, Moldovan JB, Moran JV, Silverman RH. РНКаза L ограничивает подвижность сконструированных ретротранспозонов в культивируемых клетках человека. Нуклеиновые Кислоты Res. 2014;42:3803–3820. [Бесплатная статья PMC] [PubMed] [Google Scholar]
349. Чиу Ю.Л., Грин В.К. Цитидиндезаминазы APOBEC3: врожденная защитная сеть, противостоящая экзогенным ретровирусам и эндогенным ретроэлементам. Анну Рев Иммунол. 2008; 26: 317–353. [PubMed] [Google Scholar]
2008; 26: 317–353. [PubMed] [Google Scholar]
350. Шихи А.М., Гэддис Н.К., Чой Д.Д., Малим М.Х. Выделение человеческого гена, ингибирующего инфекцию ВИЧ-1 и подавляемого вирусным белком Vif. Природа. 2002; 418: 646–650. [PubMed] [Google Scholar]
351. Bogerd HP, Wiegand HL, Hulme AE, Garcia-Perez JL, O’Shea KS, Moran JV, Cullen BR. Клеточные ингибиторы длинного вкрапленного элемента 1 и ретротранспозиции Alu. Proc Natl Acad Sci U S A. 2006;103:8780–8785. [Бесплатная статья PMC] [PubMed] [Google Scholar]
352. Chen H, Lilley CE, Yu Q, Lee DV, Chou J, Narvaiza I, Landau NR, Weitzman MD. APOBEC3A является мощным ингибитором аденоассоциированного вируса и ретротранспозонов. Карр Биол. 2006; 16: 480–485. [PubMed] [Google Scholar]
353. Hulme AE, Bogerd HP, Cullen BR, Moran JV. Селективное ингибирование ретротранспозиции Alu с помощью APOBEC3G. Ген. 2007; 390:199–205. [Бесплатная статья PMC] [PubMed] [Google Scholar]
354. Muckenfuss H, Hamdorf M, Held U, Perkovic M, Lower J, Cichutek K, Flory E, Schumann GG, Munk C. Белки APOBEC3 ингибируют LINE-1 человека. ретротранспозиция. Дж. Биол. Хим. 2006; 281:22161–22172. [PubMed] [Академия Google]
Белки APOBEC3 ингибируют LINE-1 человека. ретротранспозиция. Дж. Биол. Хим. 2006; 281:22161–22172. [PubMed] [Академия Google]
355. Шуман Г.Г. Белки APOBEC3: основные участники внутриклеточной защиты от ретротранспозиции, опосредованной LINE-1. Биохим Сок Транс. 2007; 35: 637–642. [PubMed] [Google Scholar]
356. Wissing S, Montano M, Garcia-Perez JL, Moran JV, Greene WC. Эндогенный APOBEC3B ограничивает ретротранспозицию LINE-1 в трансформированных клетках и эмбриональных стволовых клетках человека. Дж. Биол. Хим. 2011; 286:36427–36437. [Бесплатная статья PMC] [PubMed] [Google Scholar]
357. Horn AV, Klawitter S, Held U, Berger A, Vasudevan AA, Bock A, Hofmann H, Hanschmann KM, Trosemeier JH, Flory E, Jabulowsky RA, Han JS, Lower J, Lower R, Munk C, Schumann GG. Ограничение человеческого LINE-1 с помощью APOBEC3C не зависит от дезаминазы и опосредовано взаимодействием ORF1p, которое влияет на активность обратной транскриптазы LINE. Нуклеиновые Кислоты Res. 2014;42:396–416. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
358. Richardson SR, Narvaiza I, Planegger RA, Weitzman MD, Moran JV. APOBEC3A дезаминирует временно экспонированную одноцепочечную ДНК во время ретротранспозиции LINE-1. Элиф. 2014;3:e02008. [Бесплатная статья PMC] [PubMed] [Google Scholar]
359. Ник-Зайнал С., Ведж Д.С., Александров Л.Б., Петляк М., Батлер А.П., Болли Н., Дэвис Х.Р., Кнаппског С., Мартин С., Папаеммануил Э., Рамакришна М. , Shlien A, Simonic I, Xue Y, Tyler-Smith C, Campbell PJ, Stratton MR. Ассоциация полиморфизма числа копий зародышевой линии APOBEC3A и APOBEC3B с бременем предполагаемых APOBEC-зависимых мутаций при раке молочной железы. Нат Жене. 2014; 46: 487–49.1. [Бесплатная статья PMC] [PubMed] [Google Scholar]
360. Хелледей Т., Эштад С., Ник-Зайнал С. Механизмы, лежащие в основе мутационных сигнатур при раке человека. Нат Рев Жене. 2014;15:585–598. [Бесплатная статья PMC] [PubMed] [Google Scholar]
361. Crow YJ. Синдром Айкарди-Гутьера. Handb Clin Neurol. 2013; 113:1629–1635. [PubMed] [Google Scholar]
Handb Clin Neurol. 2013; 113:1629–1635. [PubMed] [Google Scholar]
362. Crow YJ, Rehwinkel J. Синдром Айкарди-Гутьера и родственные фенотипы: связь метаболизма нуклеиновых кислот с аутоиммунитетом. Хум Мол Жене. 2009 г.;18:R130–R136. [Бесплатная статья PMC] [PubMed] [Google Scholar]
363. Crow YJ, Leitch A, Hayward BE, Garner A, Parmar R, Griffith E, Ali M, Semple C, Aicardi J, Babul-Hirji R, Baumann C , Бакстер П., Бертини Э., Чандлер К.Е., Читаят Д., Кау Д., Дери С., Фаззи Э., Гойзе С., Кинг М.Д., Клеппер Дж., Лакомб Д., Ланци Г., Лайалл Х., Мартинес-Фриас М.Л., Матье М., Маккеун С. , Monier A, Oade Y, Quarrell OW, Rittey CD, Rogers RC, Sanchis A, Stephenson JB, Tacke U, Till M, Tolmie JL, Tomlin P, Voit T, Weschke B, Woods CG, Lebon P, Bonthron DT, Ponting КП, Джексон А.П. Мутации в генах, кодирующих субъединицы рибонуклеазы h3, вызывают синдром Айкарди-Гутьера и имитируют врожденную вирусную инфекцию головного мозга. Нат Жене. 2006;38:910–916. [PubMed] [Google Scholar]
364. Crow YJ, Hayward BE, Parmar R, Robins P, Leitch A, Ali M, Black DN, van Bokhoven H, Brunner HG, Hamel BC, Corry PC, Cowan FM, Frints SG , Klepper J, Livingston JH, Lynch SA, Massey RF, Meritet JF, Michaud JL, Ponsot G, Voit T, Lebon P, Bonthron DT, Jackson AP, Barnes DE, Lindahl T. Мутации в гене, кодирующем 3’–5 Экзонуклеаза ДНК TREX1 вызывает синдром Айкарди-Гутьера в локусе AGS1. Нат Жене. 2006; 38: 917–920. [PubMed] [Академия Google]
Crow YJ, Hayward BE, Parmar R, Robins P, Leitch A, Ali M, Black DN, van Bokhoven H, Brunner HG, Hamel BC, Corry PC, Cowan FM, Frints SG , Klepper J, Livingston JH, Lynch SA, Massey RF, Meritet JF, Michaud JL, Ponsot G, Voit T, Lebon P, Bonthron DT, Jackson AP, Barnes DE, Lindahl T. Мутации в гене, кодирующем 3’–5 Экзонуклеаза ДНК TREX1 вызывает синдром Айкарди-Гутьера в локусе AGS1. Нат Жене. 2006; 38: 917–920. [PubMed] [Академия Google]
365. Райс Г.И., Бонд Дж., Асипу А., Брюнет Р.Л., Мэнфилд И.В., Карр И.М., Фуллер Дж.С., Джексон Р.М., Лэмб Т., Бриггс Т.А., Али М., Горналл Х., Кутард Л.Р., Эби А., Аттард-Монтальто С.П. , Бертини Э., Бодемер С., Брокманн К., Брютон Л.А., Корри П.С., Десгерр И., Фацци Э., Касорла А.Г., Генер Б., Хамель Б.К., Хейберг А., Хантер М., ван дер Кнаап М.С., Кумар Р., Лагэ Л., Ландриу П.Г. , Лоуренко К.М., Маром Д., Макдермотт М.Ф., Ван дер Мерве В., Орчези С., Прендивиль Дж.С., Расмуссен М., Шалев С.А., Солер Д.М., Шинави М., Шпигель Р., Тан Т.И., Вандервер А. , Уэйкелинг Э.Л., Вассмер Э., Уиттакер Э. , Лебон П., Стетсон Д.Б., Бонтрон Д.Т., Кроу Ю.Дж. Мутации, связанные с синдромом Айкарди-Гутьера, предполагают участие SAMHD1 в качестве регулятора врожденного иммунного ответа. Нат Жене. 2009 г.;41:829–832. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Уэйкелинг Э.Л., Вассмер Э., Уиттакер Э. , Лебон П., Стетсон Д.Б., Бонтрон Д.Т., Кроу Ю.Дж. Мутации, связанные с синдромом Айкарди-Гутьера, предполагают участие SAMHD1 в качестве регулятора врожденного иммунного ответа. Нат Жене. 2009 г.;41:829–832. [Бесплатная статья PMC] [PubMed] [Google Scholar]
366. Стетсон Д.Б., Ко Дж.С., Хайдманн Т., Меджитов Р. Trex1 предотвращает внутриклеточное инициирование аутоиммунитета. Клетка. 2008; 134: 587–598. [Бесплатная статья PMC] [PubMed] [Google Scholar]
367. Zhao K, Du J, Han X, Goodier JL, Li P, Zhou X, Wei W, Evans SL, Li L, Zhang W, Cheung LE, Wang Г, Казазян Х.Х., младший, Ю.С.Ф. Модуляция ретротранспозиции LINE-1 и Alu/SVA с помощью SAMHD1, связанного с синдромом Айкарди-Гутьера. Cell Rep. 2013; 4:1108–1115. [Бесплатная статья PMC] [PubMed] [Google Scholar]
368. Райс Г.И., Кашер П.Р., Форте Г.М., Мэннион Н.М., Гринвуд С.М., Шинкевич М., Дикерсон Д.Е., Бхаскар С.С., Зампини М., Бриггс Т.А., Дженкинсон Э.М., Бачино К. А., Баттини Р., Бертини Э., Броган П.А., Брутон Л. А., Карпанелли М., Де Лаэт С., де Лонле П., дель Торо М., Десгерре И., Фацци Э., Гарсия-Касорла А., Хейберг А., Кавагути М., Кумар Р., Лин Дж. П., Лоуренко К. М., Мале AM, Маркес В., младший , Миньо С., Оливьери И., Орчези С., Прабхакар П., Расмуссен М., Робинсон Р.А., Розенберг Ф., Шмидт Дж.Л., Штайндл К., Тан Т.И., Ван дер Мерве В.Г., Вандервер А., Вассалло Г., Уэйклинг Э.Л., Вассмер Э., Уиттакер Э. , Ливингстон Дж.Х., Лебон П., Сузуки Т., Маклафлин П.Дж., Киган Л.П., О’Коннелл М.А., Ловелл С.К., Кроу Ю.Дж. Мутации в ADAR1 вызывают синдром Айкарди-Гутьера, связанный с сигнатурой интерферона I типа. Нат Жене. 2012;44:1243–1248. [Бесплатная статья PMC] [PubMed] [Google Scholar]
А., Баттини Р., Бертини Э., Броган П.А., Брутон Л. А., Карпанелли М., Де Лаэт С., де Лонле П., дель Торо М., Десгерре И., Фацци Э., Гарсия-Касорла А., Хейберг А., Кавагути М., Кумар Р., Лин Дж. П., Лоуренко К. М., Мале AM, Маркес В., младший , Миньо С., Оливьери И., Орчези С., Прабхакар П., Расмуссен М., Робинсон Р.А., Розенберг Ф., Шмидт Дж.Л., Штайндл К., Тан Т.И., Ван дер Мерве В.Г., Вандервер А., Вассалло Г., Уэйклинг Э.Л., Вассмер Э., Уиттакер Э. , Ливингстон Дж.Х., Лебон П., Сузуки Т., Маклафлин П.Дж., Киган Л.П., О’Коннелл М.А., Ловелл С.К., Кроу Ю.Дж. Мутации в ADAR1 вызывают синдром Айкарди-Гутьера, связанный с сигнатурой интерферона I типа. Нат Жене. 2012;44:1243–1248. [Бесплатная статья PMC] [PubMed] [Google Scholar]
369. Ода Х., Накагава К., Абэ Дж., Авая Т., Фунабики М., Хидзиката А., Нишикомори Р., Фунацука М., Осима Й., Сугавара Й., Ясуми Т., Като Х., Шираи Т., Охара О., Фудзита Т., Хайке Синдром Т. Айкарди-Гутьера вызывается мутациями IFIh2. Am J Hum Genet. 2014;95:121–125. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
370. Rice GI, del Toro Duany Y, Jenkinson EM, Forte GM, Anderson BH, Ariaudo G, Bader-Meunier B, Baildam EM, Battini R, Beresford MW, Казарано М., Чошан М., Симаз Р., Коллинз А.Э., Кордейро Н.Дж., Дейл Р.К., Дэвидсон Дж.Э., Де Ваэле Л., Десгерр И., Фавр Л., Фацци Э., Исидор Б., Лагэ Л., Лачман А.Р., Лебон П., Ли К., Ливингстон Дж. Х., Лоуренко К. М., Манкарди М. М., Масурель-Поле А., Макиннес И. Б., Менезес М. П., Миньо С., О’Салливан Дж., Орчези С., Пикко П. П., Рива Э., Робинсон Р. А., Родригес Д., Сальватичи Э., Скотт С., Шибовска М. , Толми Дж.Л., Вандервер А., Ванхулле С., Виейра Дж.П., Уэбб К., Уитни Р.Н., Уильямс С.Г., Вулф Л.А., Зубери С.М., Хур С., Кроу Ю.Дж. Мутации с приобретением функции в IFIh2 вызывают спектр фенотипов заболеваний человека, связанных с усилением передачи сигналов интерферона I типа. Нат Жене. 2014; 46: 503–509.. [Бесплатная статья PMC] [PubMed] [Google Scholar]
371. Zheng YH, Jeang KT, Tokunaga K. Факторы рестрикции хозяина при ретровирусной инфекции: перспективы взаимодействия вирус-хозяин. Ретровирусология. 2012;9:112. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Факторы рестрикции хозяина при ретровирусной инфекции: перспективы взаимодействия вирус-хозяин. Ретровирусология. 2012;9:112. [Бесплатная статья PMC] [PubMed] [Google Scholar]
372. Арьян-Одедра С., Суонсон К.М., Шерер Н.М., Волински С.М., Малим М.Х. Эндогенный MOV10 ингибирует ретротранспозицию эндогенных ретроэлементов, но не репликацию экзогенных ретровирусов. Ретровирусология. 2012;9:53. [Бесплатная статья PMC] [PubMed] [Google Scholar]
373. Goodier JL, Cheung LE, Kazazian HH., Jr РНК-хеликаза MOV10 является мощным ингибитором ретротранспозиции в клетках. Генетика PLoS. 2012;8:e1002941. [Бесплатная статья PMC] [PubMed] [Google Scholar]
374. Li X, Zhang J, Jia R, Cheng V, Xu X, Qiao W, Guo F, Liang C, Cen S. Хеликаза MOV10 ингибирует LINE-1 мобильность. Дж. Биол. Хим. 2013; 288:21148–21160. [Бесплатная статья PMC] [PubMed] [Google Scholar]
375. Gregersen LH, Schueler M, Munschauer M, Mastrobuoni G, Chen W, Kempa S, Dieterich C, Landthaler M. MOV10 представляет собой геликазу РНК от 5 ‘до 3’. способствуя деградации мРНК-мишени UPF1 путем транслокации вдоль 3′-UTR. Мол Ячейка. 2014; 54: 573–585. [PubMed] [Академия Google]
способствуя деградации мРНК-мишени UPF1 путем транслокации вдоль 3′-UTR. Мол Ячейка. 2014; 54: 573–585. [PubMed] [Академия Google]
376. Frost RJ, Hamra FK, Richardson JA, Qi X, Basssel-Duby R, Olson EN. MOV10L1 необходим для защиты сперматоцитов от ретротранспозонов с помощью Piwi-взаимодействующих РНК. Proc Natl Acad Sci U S A. 2010;107:11847–11852. [Бесплатная статья PMC] [PubMed] [Google Scholar]
377. Zheng K, Xiol J, Reuter M, Eckardt S, Leu NA, McLaughlin KJ, Stark A, Sachidanandam R, Pillai RS, Wang PJ. Мышиный MOV10L1 связывается с белками Piwi и является важным компонентом пути взаимодействующей с Piwi РНК (piRNA). Proc Natl Acad Sci U S A. 2010;107:11841–11846. [Бесплатная статья PMC] [PubMed] [Google Scholar]
378. Cordaux R, Batzer MA. Влияние ретротранспозонов на эволюцию генома человека. Нат Рев Жене. 2009; 10: 691–703. [Бесплатная статья PMC] [PubMed] [Google Scholar]
379. Branciforte D, Martin SL. Специфика экспрессии LINE-1 в яичках мышей, связанная с развитием и типом клеток: последствия для транспозиции. Мол Селл Биол. 1994; 14: 2584–2592. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Мол Селл Биол. 1994; 14: 2584–2592. [Бесплатная статья PMC] [PubMed] [Google Scholar]
380. Trelogan SA, Martin SL. Жестко регулируемая, специфичная для развития экспрессия первой открытой рамки считывания из LINE-1 во время эмбриогенеза мыши. Proc Natl Acad Sci U S A. 1995;92:1520–1524. [Бесплатная статья PMC] [PubMed] [Google Scholar]
381. Георгиу И., Нуцопулос Д., Димитриаду Э., Маркопулос Г., Аперги А., Лазарос Л., Ваксеваноглу Т., Пантос К., Сырроу М., Цаварас Т. Экспрессия РНК ретротранспозона и доказательства событий ретротранспозиции в ооцитах человека. Хум Мол Жене. 2009;18:1221–1228. [PubMed] [Google Scholar]
382. Ostertag EM, DeBerardinis RJ, Goodier JL, Zhang Y, Yang N, Gerton GL, Kazazian HH., Jr Мышиная модель ретротранспозиции L1 человека. Нат Жене. 2002; 32: 655–660. [PubMed] [Академия Google]
383. Атаникар Дж. Н., Морриш Т. А., Моран Дж. В. Человека в мышах. Нат Жене. 2002; 32: 562–563. [PubMed] [Google Scholar]
384. Kano H, Godoy I, Courtney C, Vetter MR, Gerton GL, Ostertag EM, Kazazian HH. , Jr Ретротранспозиция L1 происходит в основном в эмбриогенезе и создает соматический мозаицизм. Гены Дев. 2009; 23:1303–1312. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Jr Ретротранспозиция L1 происходит в основном в эмбриогенезе и создает соматический мозаицизм. Гены Дев. 2009; 23:1303–1312. [Бесплатная статья PMC] [PubMed] [Google Scholar]
385. Малки С., ван дер Хейден Г.В., О’Доннелл К.А., Мартин С.Л., Бортвин А. Роль ретротранспозона LINE-1 в истощении ооцитов плода у мышей. Ячейка Дев. 2014;29: 521–533. [Бесплатная статья PMC] [PubMed] [Google Scholar]
386. Freeman P, Macfarlane C, Collier P, Jeffreys AJ, Badge RM. Обогащение гибридизацией L1: метод прямого доступа к вставкам de novo L1 в зародышевой линии человека. Хум Мутат. 2011; 32: 978–988. [Бесплатная статья PMC] [PubMed] [Google Scholar]
387. Ван ден Хурк Дж. А., Мейдж И. С., Селеме М. С., Кано Х., Никопулос К., Хуфслоот Л. Х., Систерманс Э. А., де Вийс И. Д., Мухопадхьяй А., Пломп А. С., де Джонг П.Т., Казазян Х.Х., Кремерс Ф.П. Ретротранспозиция L1 может происходить на ранних стадиях эмбрионального развития человека. Хум Мол Жене. 2007; 16: 1587–159.2. [PubMed] [Google Scholar]
388. Гарсия-Перес Дж. Л., Маркетто М. С., Муотри А. Р., Куфал Н. Г., Гейдж Ф. Х., О’Ши К. С., Моран Дж. В. Ретротранспозиция LINE-1 в эмбриональных стволовых клетках человека. Хум Мол Жене. 2007; 16: 1569–1577. [PubMed] [Google Scholar]
Гарсия-Перес Дж. Л., Маркетто М. С., Муотри А. Р., Куфал Н. Г., Гейдж Ф. Х., О’Ши К. С., Моран Дж. В. Ретротранспозиция LINE-1 в эмбриональных стволовых клетках человека. Хум Мол Жене. 2007; 16: 1569–1577. [PubMed] [Google Scholar]
389. Гарсия-Перес Дж. Л., Морелл М., Шейс Дж. О., Кулпа Д. А., Морелл С., Картер К. С., Хаммер Г. Д., Коллинз К. Л., О’Ши К. С., Менендес П., Моран Дж. В. Эпигенетическое замалчивание сконструированных событий ретротранспозиции L1 в клетках эмбриональной карциномы человека. Природа. 2010;466:769–773. [Бесплатная статья PMC] [PubMed] [Google Scholar]
390. Wissing S, Munoz-Lopez M, Macia A, Yang Z, Montano M, Collins W, Garcia-Perez JL, Moran JV, Greene WC. Перепрограммирование соматических клеток в iPS-клетки активирует подвижность ретроэлементов LINE-1. Хум Мол Жене. 2012;21:208–218. [Бесплатная статья PMC] [PubMed] [Google Scholar]
391. Muotri AR, Chu VT, Marchetto MC, Deng W, Moran JV, Gage FH. Соматический мозаицизм в клетках-предшественниках нейронов, опосредованный ретротранспозицией L1. Природа. 2005;435:903–910. [PubMed] [Google Scholar]
Природа. 2005;435:903–910. [PubMed] [Google Scholar]
392. Coufal NG, Garcia-Perez JL, Peng GE, Yeo GW, Mu Y, Lovci MT, Morell M, O’Shea KS, Moran JV, Gage FH. Ретротранспозиция L1 в нейральных клетках-предшественниках человека. Природа. 2009; 460:1127–1131. [Бесплатная статья PMC] [PubMed] [Google Scholar]
393. Muotri AR, Marchetto MC, Coufal NG, Oefner R, Yeo G, Nakashima K, Gage FH. Ретротранспозиция L1 в нейронах модулируется MeCP2. Природа. 2010; 468:443–446. [Бесплатная статья PMC] [PubMed] [Google Scholar]
394. Бэйли Дж.К., Барнетт М.В., Аптон К.Р., Герхардт Д.Дж., Ричмонд Т.А., Де Сапио Ф., Бреннан П.М., Риццу П., Смит С., Фелл М., Талбот Р.Т., Густинич С., Фриман Т.С., Мэттик Д.С., Хьюм Д.А., Хьютинк П., Карнинчи П., Джеддело Дж. А., Фолкнер Г. Дж. Соматическая ретротранспозиция изменяет генетический ландшафт человеческого мозга. Природа. 2011; 479: 534–537. [Бесплатная статья PMC] [PubMed] [Google Scholar]
395. Evrony GD, Cai X, Lee E, Hills LB, Elhosary PC, Lehmann HS, Parker JJ, Atabay KD, Gilmore EC, Poduri A, Park PJ, Walsh Калифорния. Анализ секвенирования одиночного нейрона ретротранспозиции L1 и соматической мутации в мозге человека. Клетка. 2012; 151: 483–49.6. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Анализ секвенирования одиночного нейрона ретротранспозиции L1 и соматической мутации в мозге человека. Клетка. 2012; 151: 483–49.6. [Бесплатная статья PMC] [PubMed] [Google Scholar]
396. Li W, Prazak L, Chatterjee N, Gruninger S, Krug L, Theodorou D, Dubnau J. Активация мобильных элементов во время старения и угасание нейронов у дрозофилы . Нат Нейроски. 2013; 16: 529–531. [Бесплатная статья PMC] [PubMed] [Google Scholar]
397. Perrat PN, DasGupta S, Wang J, Theurkauf W, Weng Z, Rosbash M, Waddell S. Транспозиционно-управляемая геномная гетерогенность в мозге дрозофилы. Наука. 2013; 340:91–95. [Бесплатная статья PMC] [PubMed] [Google Scholar]
398. Miki Y, Nishisho I, Horii A, Miyoshi Y, Utsunomiya J, Kinzler KW, Vogelstein B, Nakamura Y. Нарушение гена APC ретротранспозной вставкой последовательности L1 при раке толстой кишки. Рак Рез. 1992; 52: 643–645. [PubMed] [Google Scholar]
399. Солом С., Юинг А.Д., Рарманн Э.П., Дусет Т., Нельсон Х.Х., Бернс М. Б., Харрис Р.С., Сигмон Д.Ф., Казелла А., Эрлангер Б., Уилан С., Аптон К.Р., Шукла Р., Faulkner GJ, Largaespada DA, Kazazian HH. Jr Обширная соматическая ретротранспозиция L1 при колоректальных опухолях. Геном Res. 2012;22:2328–2338. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Б., Харрис Р.С., Сигмон Д.Ф., Казелла А., Эрлангер Б., Уилан С., Аптон К.Р., Шукла Р., Faulkner GJ, Largaespada DA, Kazazian HH. Jr Обширная соматическая ретротранспозиция L1 при колоректальных опухолях. Геном Res. 2012;22:2328–2338. [Бесплатная статья PMC] [PubMed] [Google Scholar]
400. Ли Э., Искоу Р., Ян Л., Гоккумен О., Хасели П., Люкетт Л.Дж., 3-й, Лор Дж.Г., Харрис К.С., Дин Л., Уилсон Р.К., Уилер Д.А., Гиббс Р.А., Кучерлапати Р., Ли С., Харченко П.В. , Парк ПиДжей. Ландшафт соматической ретротранспозиции при раке человека. Наука. 2012; 337:967–971. [Бесплатная статья PMC] [PubMed] [Google Scholar]
401. Helman E, Lawrence MS, Stewart C, Sougnez C, Getz G, Meyerson M. Соматическая ретротранспозиция при раке человека, выявленная с помощью полногеномного и экзомного секвенирования. Геном Res. 2014; 24:1053–1063. [Бесплатная статья PMC] [PubMed] [Google Scholar]
402. Шукла Р., Аптон К.Р., Муньос-Лопес М., Герхардт Д.Дж., Фишер М.Е., Нгуен Т., Бреннан П.М., Бэйли Дж. К., Коллино А., Гислетти С., Синха С., Яннелли Ф., Радаэлли Э., Дос Сантос А., Рапуд D, Guettier C, Samuel D, Natoli G, Carninci P, Ciccarelli FD, Garcia-Perez JL, Faivre J, Faulkner GJ. Эндогенная ретротранспозиция активирует онкогенные пути при гепатоцеллюлярной карциноме. Клетка. 2013; 153:101–111. [Бесплатная статья PMC] [PubMed] [Google Scholar]
К., Коллино А., Гислетти С., Синха С., Яннелли Ф., Радаэлли Э., Дос Сантос А., Рапуд D, Guettier C, Samuel D, Natoli G, Carninci P, Ciccarelli FD, Garcia-Perez JL, Faivre J, Faulkner GJ. Эндогенная ретротранспозиция активирует онкогенные пути при гепатоцеллюлярной карциноме. Клетка. 2013; 153:101–111. [Бесплатная статья PMC] [PubMed] [Google Scholar]
403. Rodic N, Burns KH. Длинный вкрапленный элемент-1 (LINE-1): пассажир или водитель в человеческих новообразованиях? Генетика PLoS. 2013;9:e1003402. [Бесплатная статья PMC] [PubMed] [Google Scholar]
404. Булут-Карслиоглу А., Де Ла Роса-Веласкес И.А., Рамирес Ф., Баренбойм М., Ониши-Сибахер М., Аранд Дж., Галан С., Винтер Г.Э., Энгист Б. , Gerle B, O’Sullivan RJ, Martens JH, Walter J, Manke T, Lachner M, Jenuwein T. Suv39h-зависимый h4K9me3 маркирует интактные ретротранспозоны и замалчивает элементы LINE в эмбриональных стволовых клетках мыши. Мол Ячейка. 2014; 55: 277–290. [PubMed] [Google Scholar]
405. Chow JC, Ciaudo C, Fazzari MJ, Mise N, Servant N, Glass JL, Attreed M, Avner P, Wutz A, Barillot E, Greally JM, Voinnet O, Heard E. Активность LINE-1 в формировании факультативного гетерохроматина при инактивации Х-хромосомы. Клетка. 2010;141:956–969. [PubMed] [Google Scholar]
Активность LINE-1 в формировании факультативного гетерохроматина при инактивации Х-хромосомы. Клетка. 2010;141:956–969. [PubMed] [Google Scholar]
406. Bailey JA, Carrel L, Chakravarti A, Eichler EE. Молекулярные доказательства взаимосвязи между элементами LINE-1 и инактивацией Х-хромосомы: гипотеза лионских повторов. Proc Natl Acad Sci U S A. 2000;97:6634–6639. [Бесплатная статья PMC] [PubMed] [Google Scholar]
407. Lyon MF. Инактивация Х-хромосомы: повторная гипотеза. Цитогенет Клеточный Генет. 1998; 80: 133–137. [PubMed] [Google Scholar]
408. Ричардсон С.Р. Докторская диссертация. Анн-Арбор: Мичиганский университет; 2013. Механическое исследование ингибирования LINE-1, опосредованного APOBEC3. [Академия Google]
409. Бертран Э., Чартран П., Шефер М., Шеной С.М., Сингер Р.Х., Лонг Р.М. Локализация частиц мРНК ASh2 в живых дрожжах. Мол Ячейка. 1998; 2: 437–445. [PubMed] [Google Scholar]
410. Уоллес Н., Вагстафф Б.Дж., Дейнингер П.Л., Рой-Энгель А. М. Белок LINE-1 ORF1 усиливает ретротранспозицию Alu SINE. Ген. 2008; 419:1–6. [PMC free article] [PubMed] [Google Scholar]
М. Белок LINE-1 ORF1 усиливает ретротранспозицию Alu SINE. Ген. 2008; 419:1–6. [PMC free article] [PubMed] [Google Scholar]
Discrete Cosine and Sine Transforms
Select country/regionUnited States of AmericaUnited KingdomAfghanistanÅland IslandsAlbaniaAlgeriaAmerican SamoaAndorraAngolaAnguillaAntigua and BarbudaArgentinaArmeniaArubaAustraliaAustriaAzerbaijanBahamasBahrainBangladeshBarbadosBelgiumBelizeBeninBermudaBhutanBoliviaBonaire, Sint Eustatius and SabaBosnia and HerzegovinaBotswanaBrazilBritish Indian Ocean TerritoryBritish Virgin IslandsBruneiBulgariaBurkina FasoBurundiCambodiaCameroonCanadaCanary IslandsCape VerdeCayman IslandsCentral African РеспубликаЧадЧилиКитайОстров РождестваКокосовые острова (острова Килинг)КолумбияКоморские островаКонгоострова КукаКоста-РикаХорватияКубаКюрасаоКипрЧехияДемократическая Республика КонгоДанияДжибутиДоминикаДоминиканская РеспубликаЭквадорЕгипетЭль-СальвадорЭкваториальная ГвинеяЭритреяЭстонияЭфиопияФолклендские (Мальвинские) островаФарерские островаФранцияГанчияГанчияФедеративные Штаты Микро nGambiaGeorgiaGermanyGhanaGibraltarGreeceGreenlandGrenadaGuadeloupeGuamGuatemalaGuernseyGuineaGuinea-BissauGuyanaHaitiHondurasHong KongHungaryIcelandIndiaIndonesiaIranIraqIrelandIsle of ManIsraelItalyJamaicaJapanJerseyJordanKazakhstanKenyaKiribatiKuwaitKyrgyzstanLaoLatviaLesothoLiberiaLibyaLiechtensteinLuxembourgMacaoMacedoniaMadagascarMalawiMalaysiaMaldivesMaliMaltaMarshall IslandsMartiniqueMauritaniaMauritiusMayotteMexicoMoldovaMonacoMongoliaMontenegroMontserratMoroccoMozambiqueMyanmarNamibiaNepalNetherlandsNew CaledoniaNew ZealandNicaraguaNigerNiueNorfolk IslandNorth KoreaNorthern Mariana IslandsNorwayOmanPakistanPalauPanamaPapua New GuineaParaguayPeruPhilippinesPitcairnPolandPortugalPuerto RicoQatarRéunionRomaniaRwandaSaint BarthélemySaint HelenaSaint Kitts and NevisSaint LuciaSaint Martin (French part)Saint Pierre and MiquelonSaint Vincent and the GrenadinesSamoaSan MarinoSao Tome and PrincipeSaudi ArabiaSenegalSerbiaSeychellesSierra LeoneSingaporeSint Maarten (Dutch part)Slovakia SloveniaSolomon IslandsSomaliaSouth AfricaSouth Georgia and the South Sandwich IslandsSouth KoreaSouth SudanSpainSri LankaSudanSurinameSvalbard and Jan MayenSwazilandSwedenSwitzerlandSyriaTaiwanTajikistanTanzaniaThailandTimor LesteTogoTokelauTongaTrinidad and TobagoTunisiaTurkeyTurkmenistanTurks and Caicos IslandsTuvaluUgandaUkraineUnited Arab EmiratesUruguayUS Virgin IslandsUzbekistanVanuatuVatican CityVenezuelaVietnamWallis and FutunaWestern SaharaYemenZambiaZimbabwe
Варианты покупкиЭлектронная книга $225,00
Налог с продаж рассчитывается при оформлении заказа
Бесплатная доставка по всему миру
Нет минимального заказа
Описание
Дискретное косинусное преобразование (DCT) используется во многих научных, инженерных и исследовательских сообществ и, в частности, в области сжатия данных. Быстрые алгоритмы и приложения DCT Type II (DCT-II) стали основой многих установленных международных стандартов кодирования изображений/видео. С тех пор другие формы DCT и дискретного синусоидального преобразования (DST) были подробно исследованы. В этом новом издании представлен полный набор дискретных тригонометрических преобразований DCT и DST, включая их определения, общие математические свойства и связи с оптимальным преобразованием Карунена-Лоэва (KLT), с акцентом на быстрые алгоритмы (одномерные и двумерные). ) и целочисленные аппроксимации DCT и DST для их эффективной реализации в целочисленной области. DCT и DST — это преобразования с действительным знаком, которые отображают целочисленные сигналы в коэффициенты с плавающей запятой. Чтобы исключить операции с плавающей запятой, были предложены различные методы целочисленных аппроксимаций для построения и гибкой генерации семейства целочисленных преобразований DCT и DST с произвольной точностью и производительностью. Целочисленные DCT/DST с недорогой и маломощной реализацией могут заменить соответствующие вещественные преобразования в беспроводных и спутниковых системах связи, а также в портативных вычислительных приложениях.
Быстрые алгоритмы и приложения DCT Type II (DCT-II) стали основой многих установленных международных стандартов кодирования изображений/видео. С тех пор другие формы DCT и дискретного синусоидального преобразования (DST) были подробно исследованы. В этом новом издании представлен полный набор дискретных тригонометрических преобразований DCT и DST, включая их определения, общие математические свойства и связи с оптимальным преобразованием Карунена-Лоэва (KLT), с акцентом на быстрые алгоритмы (одномерные и двумерные). ) и целочисленные аппроксимации DCT и DST для их эффективной реализации в целочисленной области. DCT и DST — это преобразования с действительным знаком, которые отображают целочисленные сигналы в коэффициенты с плавающей запятой. Чтобы исключить операции с плавающей запятой, были предложены различные методы целочисленных аппроксимаций для построения и гибкой генерации семейства целочисленных преобразований DCT и DST с произвольной точностью и производительностью. Целочисленные DCT/DST с недорогой и маломощной реализацией могут заменить соответствующие вещественные преобразования в беспроводных и спутниковых системах связи, а также в портативных вычислительных приложениях. Книга представляет собой подробный экскурс по ортогональным/ортонормированным матрицам DCT и DST, их матричным факторизациям и целочисленным аппроксимациям. Есть надежда, что книга послужит ценным справочником для промышленности, научных кругов и исследовательских институтов при разработке целочисленных DCT и DST, а также источником вдохновения для дальнейших углубленных исследований.
Книга представляет собой подробный экскурс по ортогональным/ортонормированным матрицам DCT и DST, их матричным факторизациям и целочисленным аппроксимациям. Есть надежда, что книга послужит ценным справочником для промышленности, научных кругов и исследовательских институтов при разработке целочисленных DCT и DST, а также источником вдохновения для дальнейших углубленных исследований.
Основные характеристики
- Представление полного набора DCT и DST в контексте всего класса дискретных унитарных синусоидальных преобразований: происхождение, определения, общие математические свойства, взаимосвязи и отношения к оптимальному преобразованию Карунена-Лоэва (KLT)
- Унифицированная обработка с быстрыми реализациями DCT и DST: основанные на быстром вращении алгоритмы, полученные в виде рекурсивных разреженных матричных факторизаций матрицы преобразования, включая одномерные и двумерные случаи
- Подробное представление различных методов и подходов к проектированию целочисленной аппроксимации DCT и DST с использованием основных концепций линейной алгебры, теории матриц и матричных вычислений, ведущих к их эффективной реализации без множителей в реальном времени или в целом к обратимым реализациям преобразования целого числа в целое
- Полный список дополнительных ссылок, отражающих последние/последние разработки в области эффективных реализаций DCT и DST, в основном одно-, двух-, трех- и многомерных быстрых алгоритмов DCT/DST, включая последние активные темы исследований за период времени с 1990 по настоящее время
Читательская аудитория
Ученые, исследователи и инженеры (научно-исследовательские институты, университеты и компании). Он также подходит для аспирантов, изучающих электротехнику и информатику
Он также подходит для аспирантов, изучающих электротехнику и информатику
Содержание
- 1. Дискретные косинусные и синусоидальные преобразования
2. Определения и общие свойства
3. Преобразование Карунена-Лоева и оптимальная декорреляция
4. Быстро Алгоритмы DCT/DST
5. Целочисленные дискретные косинусные/синусные преобразования
Об авторах
Владимир Британак
В. Британак окончил математический факультет в 1978 г., получил степень RNDr. степень в области теоретической кибернетики и математической информатики в 1987 г. на факультете математики и физики Университета Коменского в Братиславе и степень доктора наук. степень (эквивалент доктора философии) в области компьютерных наук в 1995 Словацкой академии наук в Братиславе. В настоящее время он работает в Институте информатики Словацкой академии наук в Братиславе в качестве старшего научного сотрудника. Его исследовательские интересы включают цифровую обработку изображений и сигналов, дискретные ортогональные синусоидальные преобразования и наборы фильтров с косинусной/синусоидальной модуляцией идеальной реконструкции (MDCT/MDST): быстрые алгоритмы, матричные факторизации и целочисленные аппроксимации. Он опубликовал несколько статей в реферируемых журналах. Он является членом IEEE.
Он опубликовал несколько статей в реферируемых журналах. Он является членом IEEE.
Принадлежности и опыт работы
Словацкая академия наук, Институт информатики, Братислава, Словацкая Республика
Патрик Йип
До выхода на пенсию в 2004 году Пэт Йип активно работал в области цифровой обработки сигналов. Он опубликовал множество статей в технических журналах и читал курсы инженерной математики. Несколько докторантов завершили свои исследования под его руководством.
Принадлежности и опыт
Университет Макмастера, кафедра математики и статистики, Гамильтон, Канада
К. Р. Рао
К. Р. Рао получил степень доктора наук по электротехнике в Университете Нью-Мексико, Альбукерке, в 1966 г. С 1966 г. он работает в Техасский университет в Арлингтоне, где он в настоящее время является профессором электротехники. Он вместе с двумя другими исследователями представил дискретное косинусное преобразование в 1975 году, которое с тех пор стало очень популярным в цифровой обработке сигналов. Он является соавтором книг «Ортогональные преобразования для цифровой обработки сигналов» (Springer-Verlag, 1975). Также записано для слепых шрифтом Брайля Королевским национальным институтом для слепых: «Быстрые преобразования: анализ и приложения» (Academic Press, 1982), «Дискретное косинусное преобразование: алгоритмы, преимущества, приложения» (Academic Press, 1990). Он редактировал эталонный том «Дискретные преобразования и их приложения» (Van Nostrand Reinhold, 1985). Он был соредактором эталонного тома «Телеконференции» (Van Nostrand Reinhold, 1985). Он является соавтором книг «Методы и стандарты кодирования изображений/видео/аудио» (Prentice Hall, 19).96), «Пакетная видеосвязь по сетям ATM» (Прентис Холл, 2000 г.) и «Мультимедийные системы связи» (Прентис Холл, 2002 г.). Он был соредактором справочника «Справочник по преобразованию и сжатию данных» (CRC Press, 2001 г.), «Качество цифрового видеоизображения и перцептивное кодирование» (совместно с Х. Р. Ву), Тейлор и Фрэнсис, ноябрь 2005 г.
Он является соавтором книг «Ортогональные преобразования для цифровой обработки сигналов» (Springer-Verlag, 1975). Также записано для слепых шрифтом Брайля Королевским национальным институтом для слепых: «Быстрые преобразования: анализ и приложения» (Academic Press, 1982), «Дискретное косинусное преобразование: алгоритмы, преимущества, приложения» (Academic Press, 1990). Он редактировал эталонный том «Дискретные преобразования и их приложения» (Van Nostrand Reinhold, 1985). Он был соредактором эталонного тома «Телеконференции» (Van Nostrand Reinhold, 1985). Он является соавтором книг «Методы и стандарты кодирования изображений/видео/аудио» (Prentice Hall, 19).96), «Пакетная видеосвязь по сетям ATM» (Прентис Холл, 2000 г.) и «Мультимедийные системы связи» (Прентис Холл, 2002 г.). Он был соредактором справочника «Справочник по преобразованию и сжатию данных» (CRC Press, 2001 г.), «Качество цифрового видеоизображения и перцептивное кодирование» (совместно с Х. Р. Ву), Тейлор и Фрэнсис, ноябрь 2005 г. , «Введение в мультимедийные коммуникации: Applications, Middleware, Networking» (совместно с Z.S. Bojkovic и D.A. Milovanovic), Wiley, 2005. Некоторые из его книг были переведены на японский, китайский, корейский и русский языки, а также опубликованы в виде азиатских изданий (в мягкой обложке). Он был внешним экзаменатором для аспирантов университетов Австралии, Канады, Гонконга, Индии, Сингапура, Таиланда и Тайваня. Он был приглашенным профессором в нескольких университетах — от 3 до 7 недель и 1/2 месяца (Австралия, Япония, Корея, Сингапур и Таиланд). Он проводил семинары/учебники по стандартам кодирования видео/аудио
, «Введение в мультимедийные коммуникации: Applications, Middleware, Networking» (совместно с Z.S. Bojkovic и D.A. Milovanovic), Wiley, 2005. Некоторые из его книг были переведены на японский, китайский, корейский и русский языки, а также опубликованы в виде азиатских изданий (в мягкой обложке). Он был внешним экзаменатором для аспирантов университетов Австралии, Канады, Гонконга, Индии, Сингапура, Таиланда и Тайваня. Он был приглашенным профессором в нескольких университетах — от 3 до 7 недель и 1/2 месяца (Австралия, Япония, Корея, Сингапур и Таиланд). Он проводил семинары/учебники по стандартам кодирования видео/аудио
Принадлежности и опыт
Техасский университет в Арлингтоне, факультет электротехники, Техас, США
Рейтинги и обзоры
Написать отзыв
В настоящее время нет обзоров для «Дискретные косинусные и синусоидальные преобразования» 31 Dgenome кодируется повторами LINE и SINE
Трехмерный геном, кодируется повторами LINE и SINE
Скачать PDF
- Обзор исследования
- Опубликовано:
- Чжэньюй Лян 1 и
- Сян-Дун Фу ORCID: orcid.org/0000-0001-5499-8732 1
Клеточные исследования том 31 , страницы 603–604 (2021)Процитировать эту статью
3092 доступа
2 Цитаты
20 Альтметрический
Сведения о показателях
Субъекты
- Структура хроматина
- Длинные некодирующие РНК
У вас есть полный доступ к этой статье через ваше учреждение.
Скачать PDF
Скачать PDF
Ретротранспозоны встроены в различные области генома, многие из которых дают начало транскриптам, обогащенным повторами. В недавней статье Клеточные исследования , Лу и др. показывают, что ретротранспозоны L1 и B1 отдельно сгруппированы в геномах млекопитающих, где транскрипция и транскрипты L1 играют жизненно важную роль в организации формирования трехмерного генома.
Эукариотический геном иерархически упакован в ядре в виде многоуровневых структурных единиц, что позволяет транскрипции генов и репликации ДНК происходить пространственно и временно регулируемым образом. 1 Высокопроизводительное картирование взаимодействий ДНК-ДНК показывает, что геном человека разделен на два домена компартмента, названные А и В, с различной транскрипционной активностью, модификациями гистонов и ядерным позиционированием. 2 В основе клетчатой структуры компартментов A/B лежат локально взаимодействующие последовательности, соответствующие топологически ассоциированным доменам (TAD), которые также участвуют в предпочтительных гомотипических (т. е. A–A и B–B) дальних взаимодействиях через всю хромосому (рис. 1).
2 В основе клетчатой структуры компартментов A/B лежат локально взаимодействующие последовательности, соответствующие топологически ассоциированным доменам (TAD), которые также участвуют в предпочтительных гомотипических (т. е. A–A и B–B) дальних взаимодействиях через всю хромосому (рис. 1).
a Дифференциальное распределение элементов Alu/B1 и L1 в геноме мыши с преимущественной локализацией Alu/B1 в компартментах A и L1 в компартментах B на карте ДНК-ДНК Hi-C (получено из рис. 1f в 4 ). b Проиллюстрирована гомотипическая кластеризация этих богатых повторами элементов, что приводит к клетчатой картине частых взаимодействий A-A и B-B (красный), но относительно редких взаимодействий A-B или B-A (синий).
Изображение в полный размер
Прошлые исследования были сосредоточены на изучении принципа формирования этих геномных структур. Например, было обнаружено, что ингибирование транскрипции Pol II или истощение РНК ослабляет границы TAD и уменьшает компартментальные взаимодействия B-типа, хотя большинство гомотипических взаимодействий A-A и B-B в значительной степени сохраняются. 3 Следовательно, хотя геном динамически регулируется, его основные организационные единицы кажутся достаточно стабильными, что ставит вопрос о том, закодирована ли архитектура генома в его первичных последовательностях ДНК.
Например, было обнаружено, что ингибирование транскрипции Pol II или истощение РНК ослабляет границы TAD и уменьшает компартментальные взаимодействия B-типа, хотя большинство гомотипических взаимодействий A-A и B-B в значительной степени сохраняются. 3 Следовательно, хотя геном динамически регулируется, его основные организационные единицы кажутся достаточно стабильными, что ставит вопрос о том, закодирована ли архитектура генома в его первичных последовательностях ДНК.
В недавней статье Cell Research , 4 Сяохуа Шен и его коллеги систематически рассматривали этот фундаментальный вопрос, обнаружив, что ретротранспозоны, встроенные в геномы млекопитающих, особенно элементы Alu/B1 и L1, преимущественно обогащены эухроматическими компартментами A и гетерохроматическими отсеков B соответственно. В соответствии со специфическими маркерами хроматина, связанными с этими элементами, короткие и длинные перемежающиеся ядерные элементы (SINE и LINE) по-разному распределяются в трехмерном (3D) пространстве ядра, причем SINE (особенно Alu у людей и B1 у мышей) локализованы внутри ядра, в то время как LINE (особенно L1 как у людей, так и у мышей) вокруг периферии ядра и ядрышка. Этот паттерн консервативен среди типов клеток у млекопитающих, реконструируется во время клеточного цикла и устанавливается de novo в раннем эмбриогенезе, что указывает на то, что эти обогащенные повторами элементы играют важную роль в инструктировании геномной укладки посредством гомотипической кластеризации. Остается нерешенным вопрос, опосредованы ли такие гомотипические взаимодействия ДНК-белковыми взаимодействиями, РНК-белковыми взаимодействиями или обоими.
Этот паттерн консервативен среди типов клеток у млекопитающих, реконструируется во время клеточного цикла и устанавливается de novo в раннем эмбриогенезе, что указывает на то, что эти обогащенные повторами элементы играют важную роль в инструктировании геномной укладки посредством гомотипической кластеризации. Остается нерешенным вопрос, опосредованы ли такие гомотипические взаимодействия ДНК-белковыми взаимодействиями, РНК-белковыми взаимодействиями или обоими.
Подмножество SINE и LINE активно транскрибируется в геномах млекопитающих. Учитывая возрастающие доказательства РНК-облегченных геномных взаимодействий на уровне хроматина, 5 роль РНК, происходящих из L1, была исследована в текущем исследовании во время клеточного цикла и раннего эмбриогенеза, когда хроматин подвергается динамической реорганизации. Блокирование транскриптов L1 с помощью антисмыслового морфолино (AMO) у эмбрионов мышей ясно продемонстрировало функциональную потребность транскриптов L1 в установлении гомотипической кластеризации для разделения компартментов A и B и обеспечения их дифференциальной локализации в ядре. Хотя не все богатые повторами последовательности в геноме транскрибируются, недавнее исследование показало, что эти РНК способны нацеливаться на родственные последовательности как в цис-, так и в транс- 6 и результирующие характеристики хроматина, такие как связывание h4K9me3 и HP1α, могут затем управлять гомотипическими взаимодействиями.
Хотя не все богатые повторами последовательности в геноме транскрибируются, недавнее исследование показало, что эти РНК способны нацеливаться на родственные последовательности как в цис-, так и в транс- 6 и результирующие характеристики хроматина, такие как связывание h4K9me3 и HP1α, могут затем управлять гомотипическими взаимодействиями.
По сравнению с транскриптами L1 функциональный вклад SINE РНК в разделение генома остается менее ясным. Технически, истощение транскриптов, происходящих из B1, приводит к тяжелой гибели клеток, что препятствует функциональной оценке этих РНК при образовании эухроматина. Теоретически различные кодирующие и некодирующие РНК, включая транскрипты SINE, могут работать вместе, чтобы способствовать образованию различных транскрипционных узлов как части А-компартментов. В связи с этим стоит отметить, что транскрипты SINE способны напрямую связывать Pol II, подмножество которых (Alu, но не scAlu у человека, B2, но не B1 у мышей) может даже подавлять транскрипцию Pol II при тепловом шоке. условия. 7 Это может изменить статус эухроматина, особенно при сверхэкспрессии транскриптов SINE. Учитывая, что различные ретротранспозоны индуцируются первыми во время активации зиготного генома, 8 можно предположить, что формирование компартментов В может происходить раньше, чем компартментов А, последние из которых могут формироваться полусамоорганизованным образом после общей структуры. гетерохроматиновых территорий. 9
условия. 7 Это может изменить статус эухроматина, особенно при сверхэкспрессии транскриптов SINE. Учитывая, что различные ретротранспозоны индуцируются первыми во время активации зиготного генома, 8 можно предположить, что формирование компартментов В может происходить раньше, чем компартментов А, последние из которых могут формироваться полусамоорганизованным образом после общей структуры. гетерохроматиновых территорий. 9
И последнее, но не менее важное: активные ретротранспозоны, встроенные в геномы млекопитающих, вероятно, способствуют организации трехмерного генома посредством образования РНК-опосредованных капель, разделенных фазами. Эта концепция на самом деле применима как к гетерохроматину, так и к эухроматину, образуя отдельные внутриядерные компартменты. 10 Функциональное различие заключается в том, что РНК, происходящие из повторов, первоначально способствуют образованию гетерохроматина, который затем становится в значительной степени молчащим при транскрипции, тогда как различные кодирующие и некодирующие РНК помогают зародышам образования эухроматина, чтобы управлять и поддерживать активную транскрипцию. В обоих этих процессах зарождающиеся РНК являются не только продуктами, но и ключевыми архитектурными компонентами трехмерного генома.
В обоих этих процессах зарождающиеся РНК являются не только продуктами, но и ключевыми архитектурными компонентами трехмерного генома.
Ссылки
Rowley, M.J. & Corces, V.G. Нац. Преподобный Жене. 19 , 789–800 (2018).
КАС Статья Google ученый
Либерман-Эйден, Э. и др. Наука 326 , 289–293 (2009).
КАС Статья Google ученый
Barutcu, A.R., Blencowe, BJ & Rinn, JL EMBO Rep. 20 , e48068 (2019).
КАС Статья Google ученый
Lu, J.Y. et al. Сотовые Res . https://doi.org/10.1038/s41422-020-00466-6 (2021 г.).
Li, X. & Fu, X. D. Nat. Преподобный Жене.
 20 , 503–519 (2019).
20 , 503–519 (2019).КАС Статья Google ученый
Хао Ю. и др. Рез. генома. 30 , 1570–1582 (2020).
КАС Статья Google ученый
Маринер, П. Д. и др. Мол. Сотовый 29 , 499–509 (2008).
КАС Статья Google ученый
Hendrickson, P.G. et al. Нац. Жене. 49 , 925–934 (2017).
КАС Статья Google ученый
Фальк, М. и др. Природа 570 , 395–399 (2019).
КАС Статья Google ученый
Henninger, J.E. et al. Cell 184 , 207–225 (2021).
КАС Статья Google ученый
Ссылки для скачивания
Информация об авторе
Авторы и организации
Кафедра клеточной и молекулярной медицины, Калифорнийский университет, Сан-Диего, Ла-Хойя, Калифорния,
-0651, USAZhengyu Liang & Xiang-Dong Fu
Авторы
- Zhengyu Liang
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Xiang-Dong Fu
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Автор, ответственный за корреспонденцию
Сян-Дун Фу.
Права и разрешения
Перепечатка и разрешения
Об этой статье
Эта статья цитируется
Сравнительная трехмерная архитектура генома позвоночных
- Диян Ли
- Менгнан Он
- Минчжоу Ли
ВМС Биология (2022)
Для чего используются синусоидальные стержни и синусоидальные пластины?
Когда дело доходит до измерения углов деталей или измерения угловых деталей, синусоидальная линейка и синусоидальная пластина невероятно важны.
Но что такое синусоидальная полоса и синусоидальная пластина? Что именно они делают?
Чтобы точно измерить угол или прямолинейность детали, которая не является плоской с самого начала, можно использовать синусоидальный прибор для выравнивания поверхности. По сути, поместив измеряемый объект или деталь на синусоидальный стержень или синусоидальную пластину, вы можете поднять конец на необходимую высоту, ориентируя объект или деталь параллельно. Таким образом, использование синусоидальной линейки или синусоидальной пластины упрощает общий процесс измерения при работе с углами.
Сам синусоидальный стержень представляет собой стальной стержень, изготовленный из тонко отшлифованной или притертой стали, обычно не шире 1 дюйма. На каждом конце синусоидального стержня расположен прецизионный цилиндр, который упирается в упоры, выточенные на нижней поверхности. Синусоидальные стержни могут иметь верхний и нижний концы, а также верхнюю и нижнюю поверхности, что позволяет использовать их в любом направлении и с любой стороны. Чтобы удерживать объект или деталь на месте, вдоль синусоидального стержня проделаны отверстия, в которые могут быть помещены упоры или зажимы. Синусоидальные блоки идентичны синусоидам, но немного шире.
Чтобы удерживать объект или деталь на месте, вдоль синусоидального стержня проделаны отверстия, в которые могут быть помещены упоры или зажимы. Синусоидальные блоки идентичны синусоидам, но немного шире.
Синусоидальные пластины также похожи по конструкции, но имеют несколько важных отличий. Синусоидальная пластина имеет гораздо более широкую структуру (как поверхностная пластина) и соединена шарниром с опорной пластиной в нижнем цилиндре. Синусоидальная линейка, синусоидальный блок и синусоидальная пластина используются в основном в качестве инструментов для точных измерений, но при определенных обстоятельствах синусоидальные пластины также могут использоваться в качестве фиксирующих устройств.
При использовании синусоидальной линейки и синусоидальной пластины процесс идентичен. Измерительные блоки используются для размещения синусоидального стержня или синусоидальной пластины под правильным углом. Измерительный блок или блоки размещаются непосредственно под верхним цилиндром синусоидального стержня или синусоидальной пластины, образующей прямоугольную призму.
Этот треугольник становится основой для ваших измерений при использовании синусоидального стержня или пластины, что позволяет вам рассчитать выходной сигнал на основе уравнения синуса, что и дало название этим инструментам.
Вертикальная грань треугольника проходит через мерный блок и заканчивается на оси верхнего цилиндра. Основание треугольника будет располагаться горизонтально над поверхностью стержня или пластины и заканчиваться на оси нижнего цилиндра. Гипотенуза треугольника заканчивается в каждом из двух цилиндров синусоидального стержня или синусоидальной пластины и лежит параллельно верхней поверхности инструмента. Цилиндры синусоидального стержня или пластины используются в качестве точек контакта во время измерений и гарантируют, что длина гипотенузы треугольника останется стабильной независимо от того, какой угол необходим. Как правило, центры каждого цилиндра находятся на математически удобном расстоянии друг от друга (обычно 5 дюймов или 10 дюймов).
Чтобы использовать синусоидальную линейку или синусоидальную пластину, необходимо применить тригонометрию. Несмотря на то, что с математической точки зрения концепция синусоидального стержня и пластины пугает, она невероятно полезна при выполнении сложных точных измерений и проста, если вы понимаете основные принципы. По сути, цель синусоидального стержня или синусоидальной пластины и связанных с ними калибровочных блоков состоит в том, чтобы сформировать точный угол. Центральное место в этом процессе занимает треугольник ABC, образованный синусоидальным стержнем или пластиной. Любой угол ϴ можно задать, используя известную длину стороны AB, а затем рассчитав высоту стороны BC по формуле BC = AB x sin(ϴ). Кроме того, угол ϴ определяется формулой ϴ = asin(BC/AB). Из-за сложности этих вычислений, а также из-за наличия нескольких движущихся частей, необходимых для выполнения измерений с использованием синусоидальных линейок и синусоидальных пластин, вам нужно быть осторожным при использовании этих инструментов. Синусоидальная линейка и синусоидальная пластина являются прекрасными инструментами для точного измерения при работе с наклонными деталями или объектами. Знание того, как правильно использовать эти инструменты, требует терпения и практики.
Синусоидальная линейка и синусоидальная пластина являются прекрасными инструментами для точного измерения при работе с наклонными деталями или объектами. Знание того, как правильно использовать эти инструменты, требует терпения и практики.
Видео уроки
Shop для синусоидальных баров
Посмотреть наш выбор синусоидальных стержней здесь
Часто задаваемые вопросы
- Как насыщенные шарниры и Sine Plates.
Синусоидальная планка и синусоидальная пластина на практике используются одинаково. Используя калибровочные блоки, синусоидальный стержень или синусоидальную пластину размещают под нужным углом или под углом, который соответствует заготовке. При размещении непосредственно под верхним цилиндром образуется прямоугольная призма ABC, которая затем служит основой для ваших измерений. Этот треугольник позволяет применить уравнение синуса из тригонометрии для расчета диапазона измерений.
 Отсюда и получили свое название синусоидальные стержни и синусоидальные пластины. Для теперь сформированного треугольника вертикальная грань заканчивается на оси верхнего цилиндра и проходит через калибровочный блок, основание горизонтально над поверхностью синусоидального стержня или синусоидальной пластины и заканчивается на оси нижнего цилиндра, а гипотенуза лежит параллельно верхней поверхности заготовки и заканчивается в каждом из двух цилиндров инструмента. Во время измерений цилиндры обеспечивают постоянство длины гипотенузы треугольника при любых изменениях угла и используются в качестве точек контакта при измерении. Наиболее распространенные расстояния между центрами каждого цилиндра составляют 5 дюймов и 10 дюймов, что делает необходимые расчеты более удобными.
Отсюда и получили свое название синусоидальные стержни и синусоидальные пластины. Для теперь сформированного треугольника вертикальная грань заканчивается на оси верхнего цилиндра и проходит через калибровочный блок, основание горизонтально над поверхностью синусоидального стержня или синусоидальной пластины и заканчивается на оси нижнего цилиндра, а гипотенуза лежит параллельно верхней поверхности заготовки и заканчивается в каждом из двух цилиндров инструмента. Во время измерений цилиндры обеспечивают постоянство длины гипотенузы треугольника при любых изменениях угла и используются в качестве точек контакта при измерении. Наиболее распространенные расстояния между центрами каждого цилиндра составляют 5 дюймов и 10 дюймов, что делает необходимые расчеты более удобными.- Как рассчитывается необходимая высота блока мер при использовании синусоидальных стержней и синусоидальных пластин?
Формула Sin B = b/a используется для расчета требуемой высоты концевых мер, необходимых для измерения.
 В этой формуле B — искомый угол, b — высота треугольника, a — длина гипотенузы. В качестве примера предположим, что мы хотим измерить прямолинейность поверхности детали, имеющей угол 27° от другой эталонной поверхности той же детали, и мы используем синусоидальный стержень или синусоидальную пластину, расстояние между которыми составляет 5 дюймов. два центра цилиндра. Используя квалифицированный калькулятор, мы завершаем формулу Sin 27 ° = b / 5 ″, что дает нам ответ b = 2,270 ″. Затем мы знаем необходимую высоту мерных блоков для размещения под верхним цилиндром синусоидального стержня или синусоидальной пластины, что делает поверхность детали непараллельной на 27° с поверочной пластиной. Тогда поверхность детали будет параллельна поверхностной пластине, если ее поместить лицом в противоположном направлении и поверх синусоидальной планки или синусоидальной пластины. Отсюда мы можем измерить деталь на прямолинейность, плоскостность, точность углов или любое другое количество возможных измерений.
В этой формуле B — искомый угол, b — высота треугольника, a — длина гипотенузы. В качестве примера предположим, что мы хотим измерить прямолинейность поверхности детали, имеющей угол 27° от другой эталонной поверхности той же детали, и мы используем синусоидальный стержень или синусоидальную пластину, расстояние между которыми составляет 5 дюймов. два центра цилиндра. Используя квалифицированный калькулятор, мы завершаем формулу Sin 27 ° = b / 5 ″, что дает нам ответ b = 2,270 ″. Затем мы знаем необходимую высоту мерных блоков для размещения под верхним цилиндром синусоидального стержня или синусоидальной пластины, что делает поверхность детали непараллельной на 27° с поверочной пластиной. Тогда поверхность детали будет параллельна поверхностной пластине, если ее поместить лицом в противоположном направлении и поверх синусоидальной планки или синусоидальной пластины. Отсюда мы можем измерить деталь на прямолинейность, плоскостность, точность углов или любое другое количество возможных измерений.
- Насколько точны синусоидальные линейки и синусоидальные пластины при проведении измерений?
Измерение угловых деталей и измерение конкретных углов для разных деталей может быть сложной задачей. Учитывая сложный характер этих задач, использование синусоидальных стержней и синусоидальных пластин для выполнения этих задач делает их намного проще. Однако процесс использования синусоидальных стержней и синусоидальных пластин может быть сложным и требует определенных вычислений. Учитывая все это, использование этих инструментов может быть очень точным, но вам нужно знать, что вы делаете. Значения синусоиды между различными угловыми измерениями могут изменяться с большой скоростью при низких значениях и очень медленно при высоких значениях. Из-за этого достигаемая точность зависит от угла, который вы измеряете. Степень точности и прецизионности, которую вы можете получить при использовании синусоидальных стержней и синусоидальных пластин, намного выше при работе с более пологими углами, чем при работе с более крутыми.
 Один из советов, который поможет устранить эту разницу, — использовать дополнение угла (90° – x) при расчетах для углов больше 45 градусов. В целом, точность синусоидальных полос и синусоидальных пластин ограничена из-за сложного количества элементов, участвующих в выполнении измерения, и степени вашего опыта в расчете окончательных измерений. Однако это отличные инструменты, незаменимые при работе с и без того сложной проблемой угловых заготовок.
Один из советов, который поможет устранить эту разницу, — использовать дополнение угла (90° – x) при расчетах для углов больше 45 градусов. В целом, точность синусоидальных полос и синусоидальных пластин ограничена из-за сложного количества элементов, участвующих в выполнении измерения, и степени вашего опыта в расчете окончательных измерений. Однако это отличные инструменты, незаменимые при работе с и без того сложной проблемой угловых заготовок.
- Синусоида
Синусоидальный стержень представляет собой прецизионный измерительный инструмент, изготовленный из тонко отшлифованной или притертой стали, используемый для измерения углов деталей или для измерения угловых деталей. Как правило, синусоидальный стержень имеет ширину не более 1 дюйма и имеет прецизионный цилиндр, расположенный на каждом конце, упирающийся в упоры, врезанные в нижнюю поверхность инструмента.
 Синусоидальный стержень используется с калибровочными блоками для удержания инструмента под углом, который образует прямоугольный треугольник ABC. Сформированный треугольник затем используется для вычисления измерения угла с использованием тригонометрии и формулы ϴ = asin(BC/AB), отсюда и их название.
Синусоидальный стержень используется с калибровочными блоками для удержания инструмента под углом, который образует прямоугольный треугольник ABC. Сформированный треугольник затем используется для вычисления измерения угла с использованием тригонометрии и формулы ϴ = asin(BC/AB), отсюда и их название.- Синусоидальный блок
Блок синуса представляет собой прецизионный измерительный инструмент, изготовленный из тонко отшлифованной или притертой стали, используемый для измерения углов деталей или для измерения угловых деталей. Синусоидальный блок — это синусоидальная полоса шириной более 1 дюйма, но недостаточно широкая, чтобы быть синусоидальной пластиной. Блоки синусоид построены и функционируют идентично линейкам синусоид.
- Синусоидальная пластина
Синусоидальная пластина представляет собой прецизионный измерительный инструмент, изготовленный из тонко отшлифованной или притертой стали, используемый для измерения углов деталей или для измерения угловых деталей.



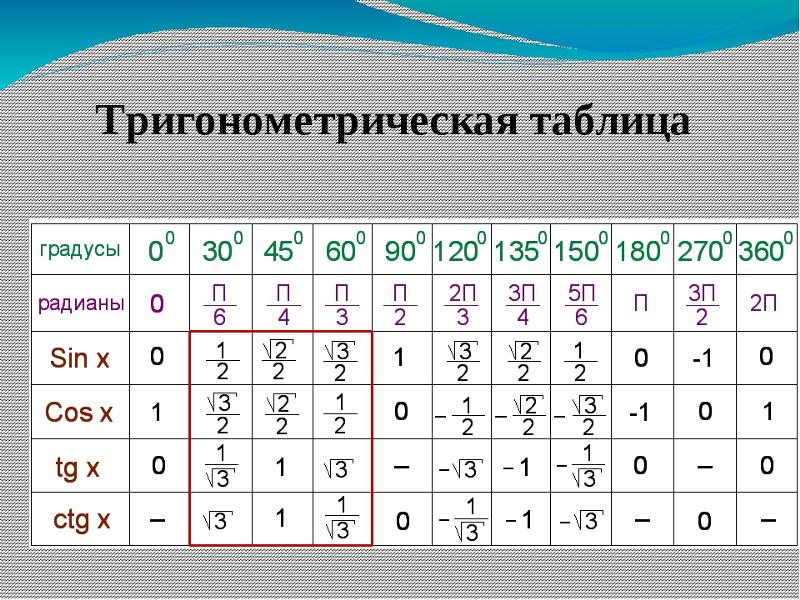



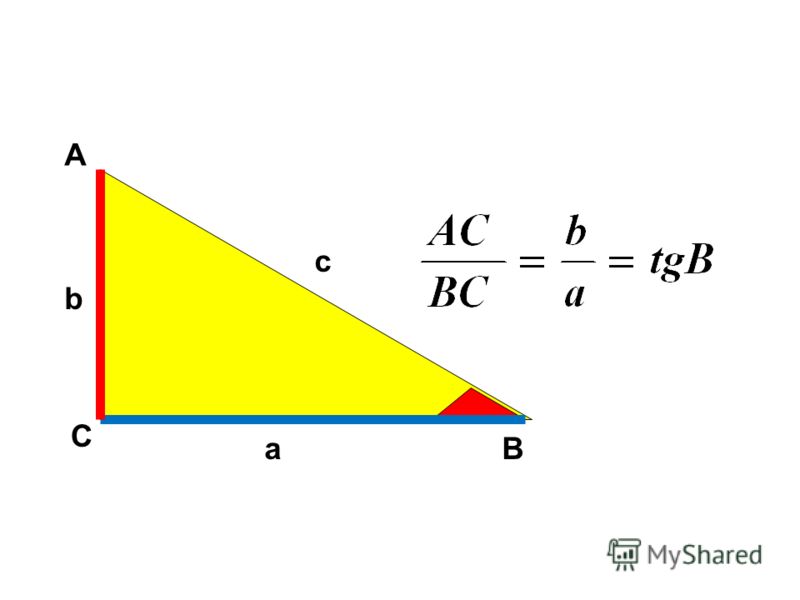
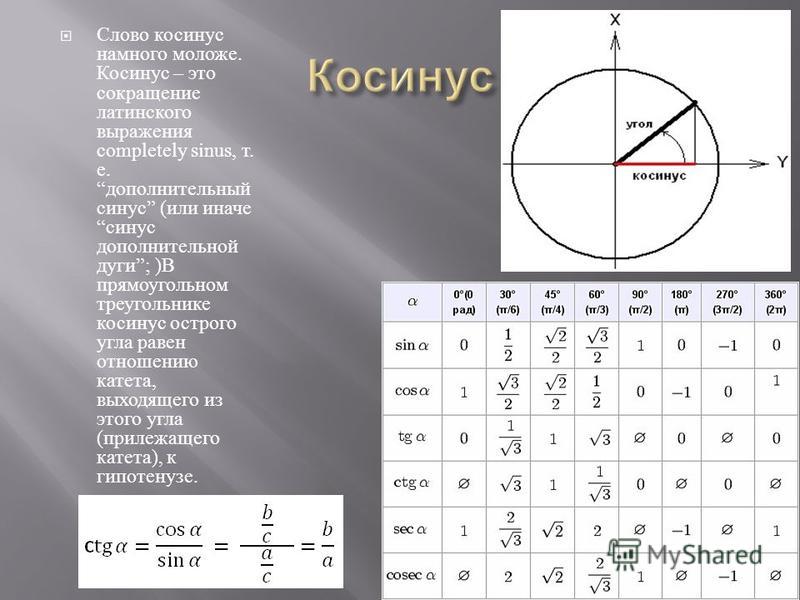

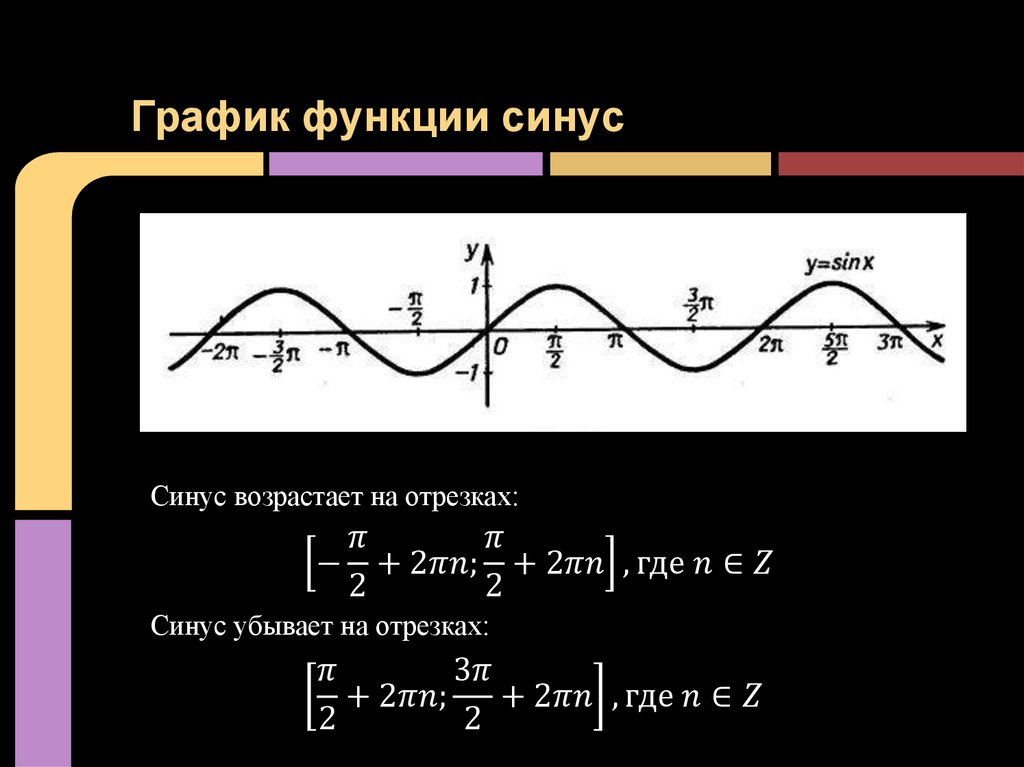 Вичман 1,2
Вичман 1,2 
 Prog Nucleic Acid Res Mol Biol. 2000; 64: 255–9.4.
Prog Nucleic Acid Res Mol Biol. 2000; 64: 255–9.4.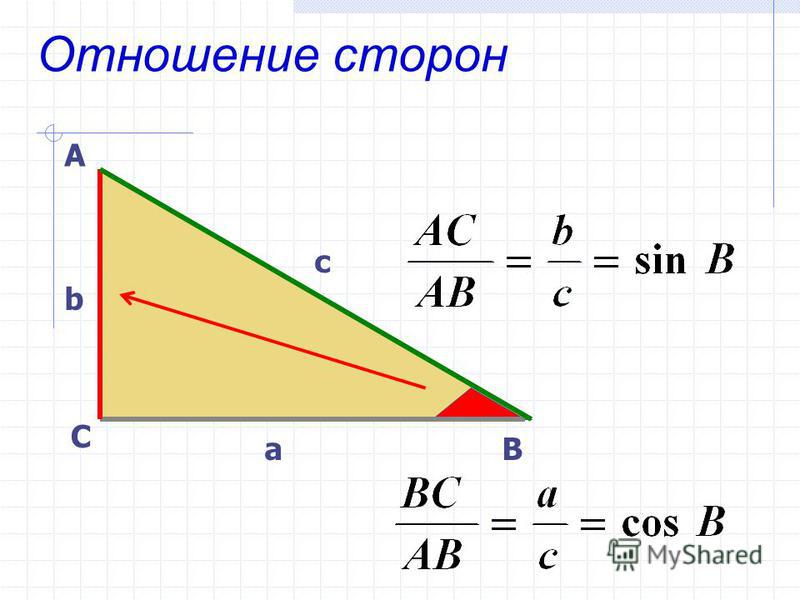 Нат Жене. 2003;35(1):41–8.
Нат Жене. 2003;35(1):41–8. Дж Мол Биол. 2001;310(1):1–26.
Дж Мол Биол. 2001;310(1):1–26. Proc Natl Acad Sci U S A. 2004;101(39):13994–4001.
Proc Natl Acad Sci U S A. 2004;101(39):13994–4001. , Фурано А.В. Эволюция длинных вкраплений повторяющейся ДНК (L1, LINE 1), выявленная в результате анализа семейства ДНК L1 древних грызунов. Дж Мол Эвол. 1993;36(1):9–20.
, Фурано А.В. Эволюция длинных вкраплений повторяющейся ДНК (L1, LINE 1), выявленная в результате анализа семейства ДНК L1 древних грызунов. Дж Мол Эвол. 1993;36(1):9–20. Ретротранспозиция L1 в нейральных клетках-предшественниках человека. Природа. 2009 г.;460(7259):1127–31.
Ретротранспозиция L1 в нейральных клетках-предшественниках человека. Природа. 2009 г.;460(7259):1127–31. Proc Natl Acad Sci U S A. 2008;105(11):4220–5.
Proc Natl Acad Sci U S A. 2008;105(11):4220–5. Генетика. 2000;154(4):1809–17.
Генетика. 2000;154(4):1809–17.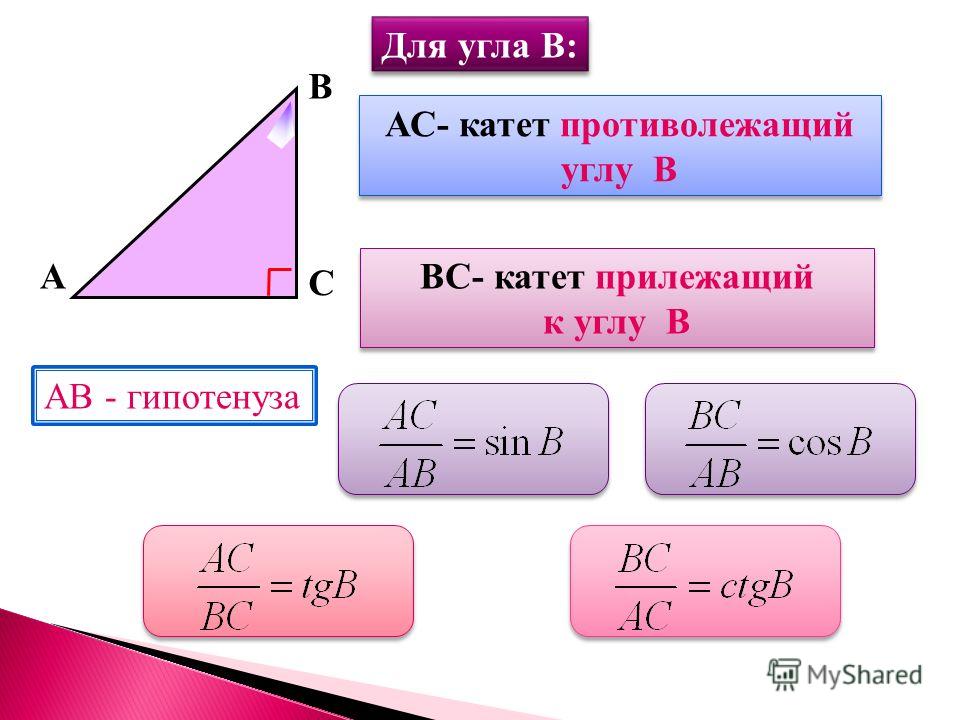 Дж Вирол. 2005;79(23): 14698–707.
Дж Вирол. 2005;79(23): 14698–707. Х., Тедфорд Р.Х. Калибровка большой американской развязки. Наука. 1979; 204 (4390): 272–9.
Х., Тедфорд Р.Х. Калибровка большой американской развязки. Наука. 1979; 204 (4390): 272–9. 2014. http://www.genomesize.com.
2014. http://www.genomesize.com. Вена: R Foundation for Statistical Computing; 2013.
Вена: R Foundation for Statistical Computing; 2013. □_\square□
□_\square□

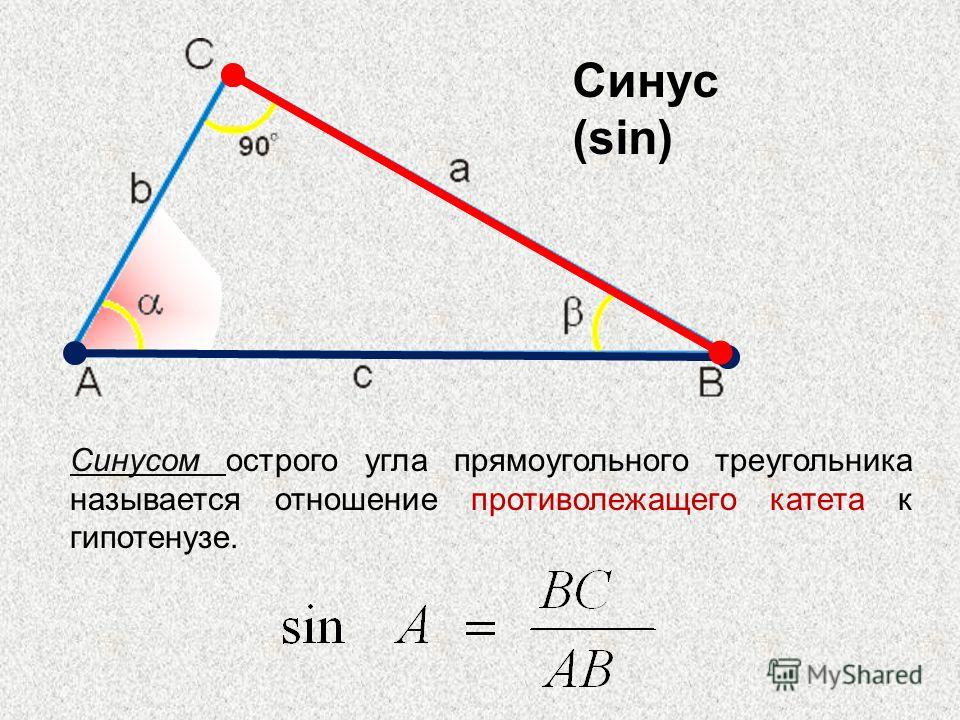 Следовательно, он равен cosθ \cos\theta cosθ. □_\квадрат□
Следовательно, он равен cosθ \cos\theta cosθ. □_\квадрат□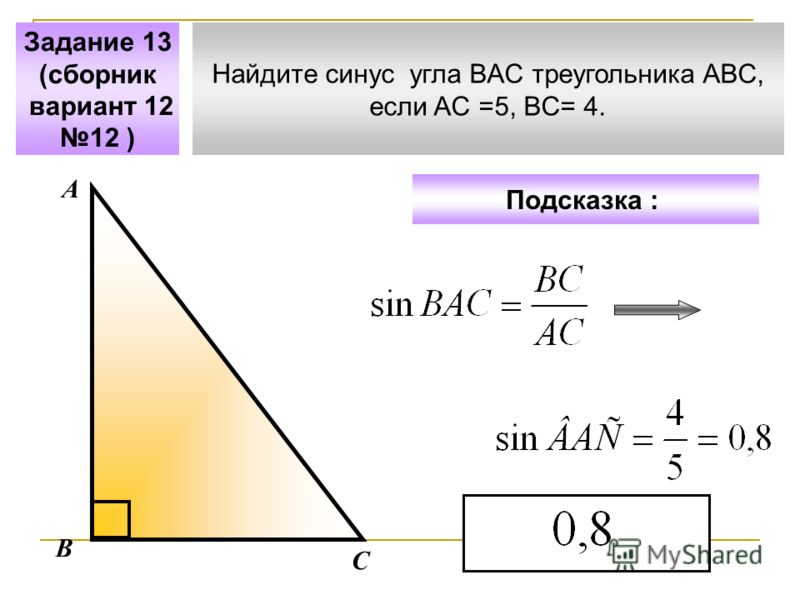 в разделе Преобразование графика.
в разделе Преобразование графика.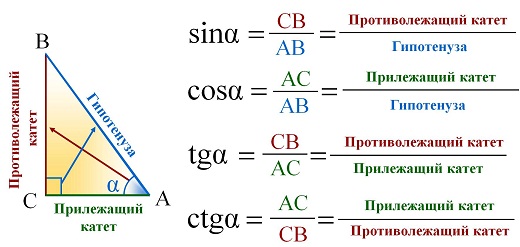 Интерпретация значений показывает следующее:
Интерпретация значений показывает следующее: Таким образом, наибольшее возможное значение равно ∣a∣+d |a| + d ∣a∣+d, а наименьшее возможное значение равно −∣a∣+d — |a| + d −∣a∣+d. □_\квадрат□
Таким образом, наибольшее возможное значение равно ∣a∣+d |a| + d ∣a∣+d, а наименьшее возможное значение равно −∣a∣+d — |a| + d −∣a∣+d. □_\квадрат□